- 1详解Nginx代理WordPress搭建个人网站系统_wordpress nginx
- 22024华为OD机试真题指南宝典—持续更新(JAVA&Python&C++&JS)【彻底搞懂算法和数据结构—算法之翼】
- 3es6.0 java,【ES】ElasticSearch基本概念阐述
- 4这个API Hub太厉害了,太适合接口测试了,收录了钉钉企业微信等开放Api的利器
- 5Egret - timer
- 6mongoDB安装报错:exception in initAndListen: IllegalOperation: Attempted to create a lock file on a read-...
- 7产品经理面试必备常见问题及解析_产品经理职级晋升答辩常见问题及答案
- 8elasticsearch8.5.3 实战工具类 + JAVA调用方法_elasticsearch 8.5 java api
- 9Python之时间与时间戳转换_python转格林时间戳
- 10oppo三面面经,现已拿到offer。十月份我快乐了,你呢?_oppo 专家 offer
基于SE3等变网络与Diffusion模型的分子生成工具 TargetDiff 评测_mmgbsa dg bind值和docking score值
赞
踩
TargetDiff是来源于ICLR2023文章:3D Equivariant Diffusion for Target-Aware Molecule Generation and Affinity Prediction。该文章基于 SE(3)-equivariant network,开发了非自回归的,具有旋转和平移不变性的,口袋为条件的分子扩散生成模型TargetDiff。文章链接:https://openreview.net/pdf?id=kJqXEPXMsE0

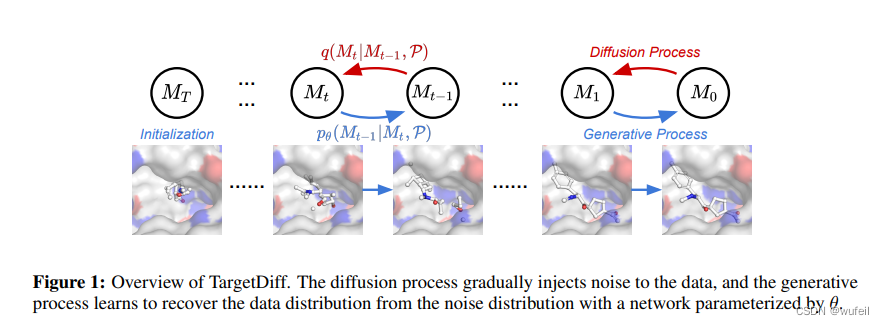
文章指出,与liGAN, AR, Pocket2Mol三种3D分子生成模型相比,TargetDiff的原子间距离分布更为合理,生成分子的中心与真实分子的RMSD更为接近,但是在成环上,会出现比例较高的七元环。

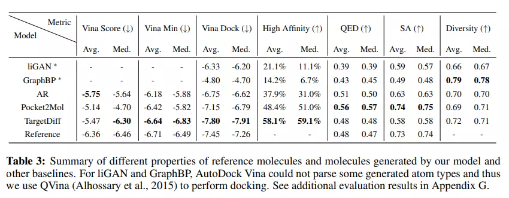
更为有趣的是,文章指出,TargetDiff生成的分子具有更好的对接Vina能量值,如下图:

项目环境
复制项目代码:
git clone https://github.com/guanjq/targetdiff.git环境安装
- conda create -n TargetDiff python=3.8
- conda activate TargetDiff
- #cuda 11.3, pytroch 版本大于1.9.0,PYG>2.0
- conda install pytorch==1.12.1 torchvision==0.13.1 torchaudio==0.12.1 cudatoolkit=11.3 -c pytorch
- conda install pyg -c pyg
-
- conda install rdkit openbabel tensorboard pyyaml easydict python-lmdb -c conda-forge
- # For Vina Docking
- pip install meeko==0.1.dev3 scipy pdb2pqr vina==1.2.2
- python -m pip install git+https://github.com/Valdes-Tresanco-MS/AutoDockTools_py3
由于numpy版本的原因,需要更新一下:
pip install numpy==1.23.0torch-cluster也要安装一下:
pip install torch-cluster生成分子
python scripts/sample_for_pocket.py configs/sampling.yml --pdb_path examples/1h36_A_rec_1h36_r88_lig_tt_docked_0_pocket10.pdbconfigs/sampling.yml是生成分子的配置文件,制定了采样所使用的预训练模型,采样分子的数量为10, 采样步数为1000,具体内容如下:
- model:
- checkpoint: ./pretrained_models/pretrained_diffusion.pt
-
- sample:
- seed: 2021
- num_samples: 10
- num_steps: 1000
- pos_only: False
- center_pos_mode: protein
- sample_num_atoms: prior
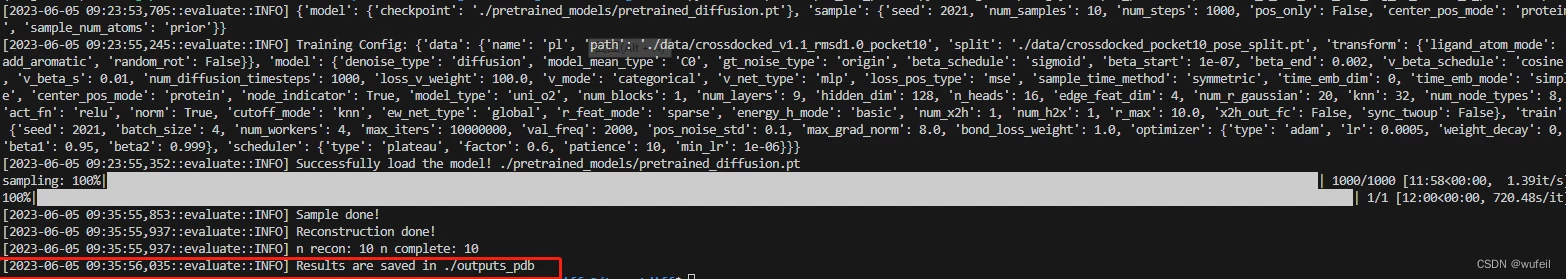
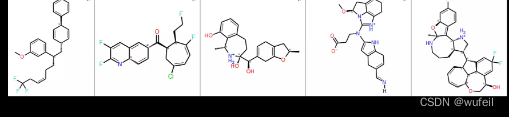
运行结束后,会生成./out_pdbs文件夹生成每一个分子的sdf文件。里面有一些分子特别奇怪,比如三个并环等。如下:

我们使用了靶点X的口袋,进行测试,口袋结构如下(口袋结构狭长,口袋范围10A):

使用上述方法,生成了10个分子,经过ligpre, docking以后,得到了5个不重复且有效的分子,对接分数和MMGBSA结合能分别如下图:
其中有一个分子的docking score < -7,且MMGBSA dG Bind < -70;从生成分子能结合在口袋的角度来说,结果还是比较有效的。
训练TargrtDiff模型
python scripts/train_diffusion.py configs/training.yml输出过程如下:
在训练过程中,会生成logs_diffusion文件夹,记录训练过程,模型的临时文件保存在:logs_diffusion/training_2023_06_15__05_58_43/checkpoints。


