- 1mac笔记本当做服务器记录_如何将mac电脑制作成服务器
- 22024年GitHub上最流行前25大Python开源项目,你收藏了吗?_github上python专门用于分割石头和沙粒的事物_python 开源项目
- 3git的ssh方式对接码云_git跟码云连不上咋回事
- 4华为OD 技术综合面,手撕代码真题整理(七):字符串的不重复子串 | 二叉树的最大路径和_华为od面试手撕代码python
- 5手把手教你使用Git_git 手把手
- 6运维人员福音!自定义插件为运维提供更多可能
- 7微信小程序面试题汇总(一)(1)
- 8记录druid-spring-boot-starter对springboot2.3.1版本的MySQL数据库密码加密过程,附带yml配置文件的配置_springboot 2.3.11.release对应druid-spring-boot-start
- 9JQuery + CSS 实现的考试系统答题卡_答题卡 已答未答 展示所有 css
- 10基于Android的运动健康管理系统_android健康运动源码
高性价比发文典范——101种机器学习算法组合革新骨肉瘤预后模型_r 101种算法机器学习构建
赞
踩
随着高通量测序技术的飞速发展和多组学分析的广泛应用,科研人员在探索生物学奥秘时经常遇到一个令人又爱又恼的问题:如何从浩如烟海的数据中挖掘出潜在的疾病关联靶点?又如何构建一个全面而有效的诊断或预后模型?只有通过优雅的数据挖掘、精致的结果展示、深入的讨论分析,并且辅以充分的湿实验验证,我们才能锻造出一篇兼具深度与广度的“干湿结合”佳作。
在这期文章里,纽科生物携手同济大学、上海交通大学和上海第六人民医院的顶尖专家,共同带来一篇全新的研究成果。这篇文章刚于今年二月鲜亮登场,发表在《Clinical and Translational Medicine》(影响因子10.6)杂志上。我们提供了关键的转录组测序与数据分析支持,本次“官方”解读旨在为广大生物学研究者呈现一条清晰的研究路径和分析流程。我们将重点解答研究者们最关注的几个问题:数据从哪里来?如何进行分析?如何选择靶点?怎样建立关联?希望这次深入浅出的分享,能激发您的研究灵感,助您一臂之力。
作为一篇有灵魂的文章,我们也有幸邀请到文章一作,王永杰博士为各位读者分享他的科研心路历程。
文章标题:Identifying squalene epoxidase as a metabolic vulnerability in high-risk osteosarcoma using an artificial intelligence-derived prognostic index
中文标题:利用人工智能得到的预后指数(artificial intelligence-derived prognostic index,AIDP1),发现鲨烯环氧酶(SQLE)在高危骨肉瘤中的代谢脆弱性
研究背景
骨肉瘤(OSA)作为最常见的原发性恶性骨肿瘤,其五年存活率低,且治疗过程极具挑战性。目前,在临床上缺少有效的患者风险分级模型,这大大限制了针对个体患者的治疗策略的制定。为了改善这一现状,本研究致力于发现新的生物标志物,以便更准确地识别高风险的OSA患者。
研究思路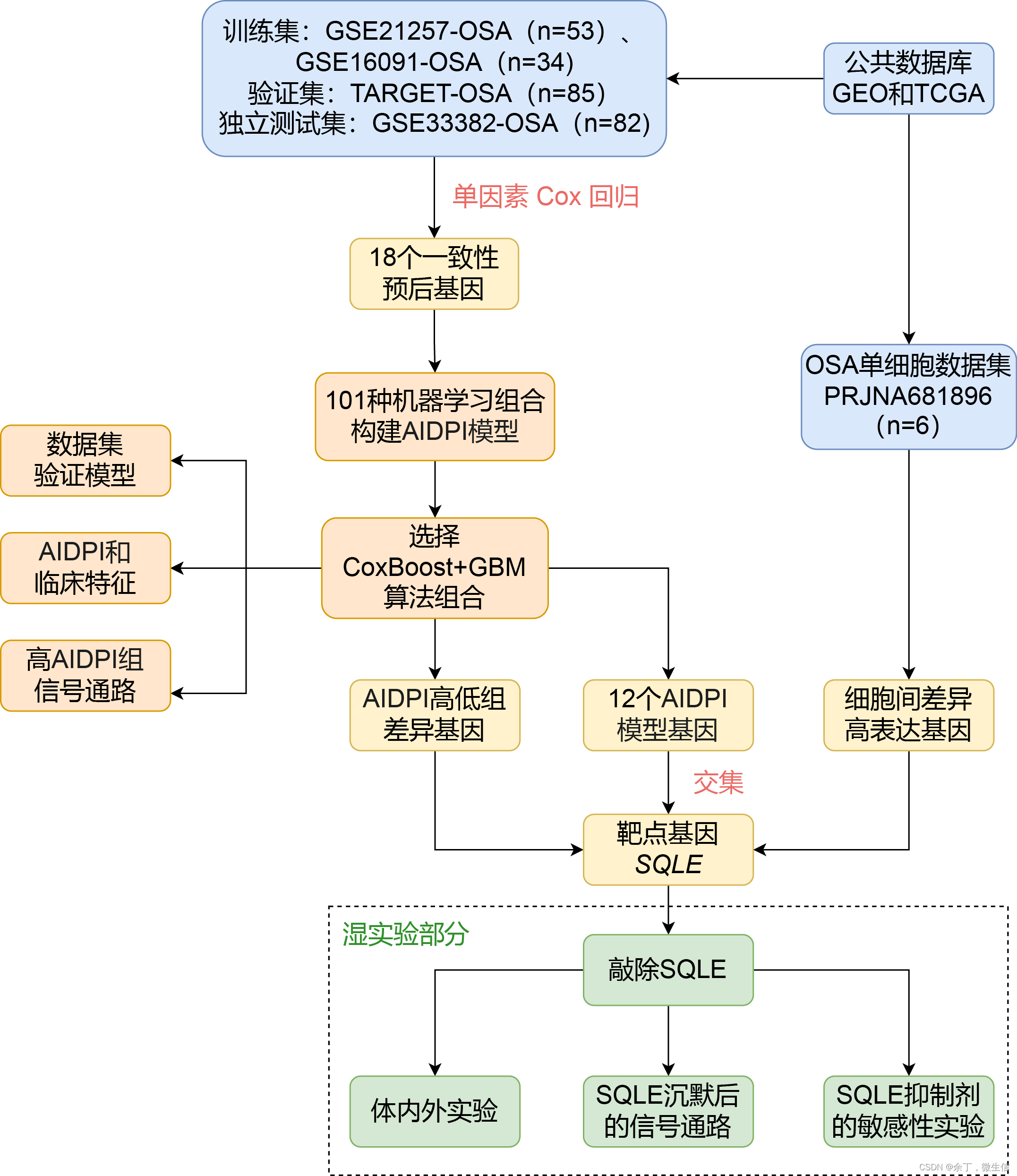
研究优势
- 干湿结合,生信数据挖掘+湿实验验证,高性价比;
- 方法学的多重筛选,多种传统机器学习算法的叠加,综合分析,结果可靠;
- Bulk-seq + scRNA-seq,粗细粒度分析结合。
Q1:来源解析——如何获取数据?
本篇文章生信部分的数据均来自公共数据库。主要使用到的数据集如下表所示:
表1. AIDP1模型构建和靶基因选择所使用的数据集
Q2:如何最大化分析方法价值,构建模型挖掘数据?
AIDPI模型的开发和验证
“机器学习”、“101种算法组合”听起来似乎既复杂又深奥,但熟悉生信的小伙伴们或多或少都听过和使用过几种机器学习算法,例如随机生存森林(RSF)、LASSO回归和支持向量机(SVM)等,这些算法在关键基因的识别中提供了极大的帮助。
绝大多数文章使用的机器学习算法至多3到5种,或类型单一没有组合,而本研究突破了这一瓶颈,创新性地组合了常用的一系列机器学习算法,包括LASSO、GBM、RSF、plsRcox、StepCox、SuperPC、ridge、Survival-SVM、CoxBoost和Enet。就像我们通常在LASSO回归之前使用Cox分析一样,作者将RSF、LASSO、CoxBoost、StepCox这些具备降维和变量筛选功能的算法作为第一步,与其他算法组合起来,于是产生了101种不同的机器学习算法组合。
在所有组合中,CoxBoost和GBM组合的mean C-index最高,预测性能最好,因此选择CoxBoost + GBM作为AIDPI模型(图1)。

图1. 101种算法组合的C-index(此处仅展示Top15算法组合,完整组合请参见文章原文)
Q3:如何充分利用模型,探究对疾病的影响?
AIDPI和临床特征构建预后模型
单因素Cox回归分析表明(图2A),AIDPI可作为OSA患者的预后生物标志物,并同年龄、MSTS分期、Huvos分级和肿瘤原发部位一起,与患者生存显著相关。此外,基于AIDPI、年龄、MSTS分期和原发肿瘤部位构建的列线图在预测OSA患者的生存概率方面,表现优于其他单独的临床特征,验证了其作为预后预测工具的优越性(图2B)。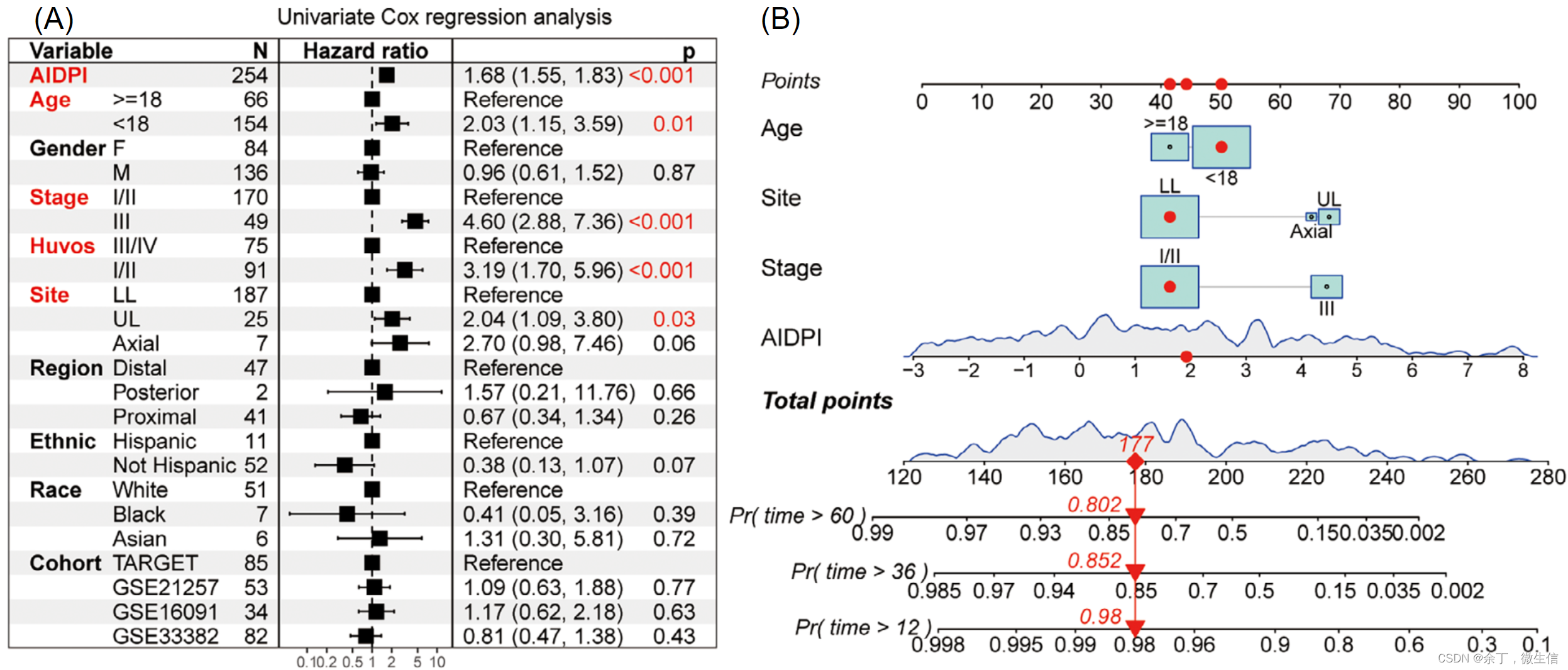
图2. 基于AIDPI和其他临床病理特征对OSA患者进行生存预测
高AIDPI患者的失调通路
GSEA显示在高AIDPI组中,MYC靶基因、胆固醇稳态和mTORC1信号通路等基因集富集增强,而与凋亡和特定免疫反应相关的基因集则发生负富集(图3A)。KEGG富集分析揭示了对OSA进展至关重要的通路,如PI3K-Akt信号通路、细胞因子与细胞因子受体相互作用、破骨细胞分化等(图3B)。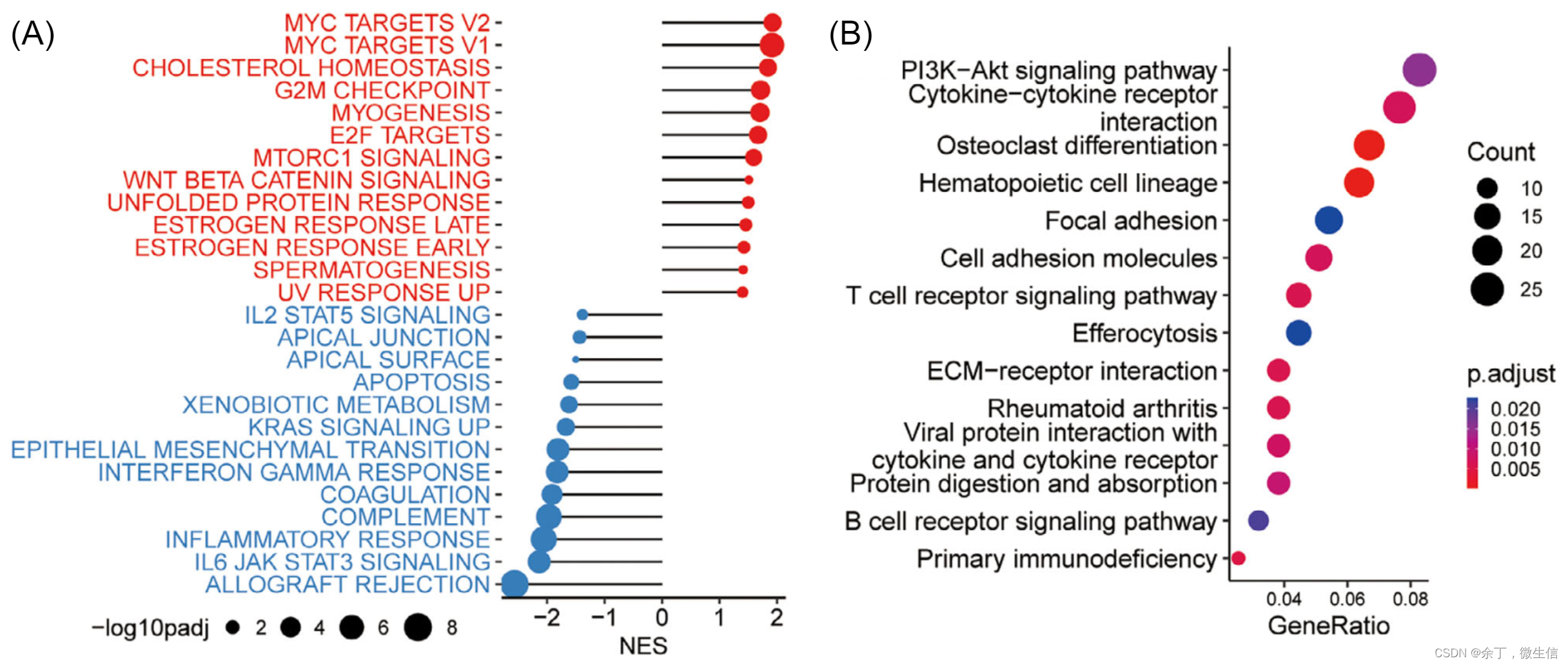
图3. 高AIDPI组患者的通路分析
Q4:如何利用单细胞数据提升颗粒度,进一步挖掘疾病核心?
单细胞分析确定高AIDPI患者核心治疗靶点
我们已经通过构建模型,获得了AIPDI差异基因和AIPDI模型基因。为了进一步缩小基因的范围,作者随后进行了单细胞分析,从6个OSA活检样本中得到了9种细胞类型:OSA细胞、B细胞、内皮细胞、骨髓细胞、NK细胞、成骨细胞、浆细胞、间质细胞和T细胞。对不同细胞类型进行差异比较,以鉴定每种细胞特异性的高表达基因(图4A)。其中,只有8%的DEGs(高、低AIDPI组差异基因)在OSA细胞中表达(图4B)。将单细胞得到的特异性高表达基因、高、低AIDPI组差异基因和AIDPI模型的12个基因取交集(图4C),最终得到3个核心基因:CORT、MYC和SQLE(图4D)。
图4. OSA单细胞数据集分析
Q5:如何寻找关键靶点,与疾病创造关联?
SQLE过表达与OSA进展密切相关
根据canSAR数据库(https://cansar.ai/),只有MYC和SQLE编码的蛋白具备可药用结构,成为高AIDPI患者的潜在治疗靶点。鉴于众多研究已经表明MYC的表达升高与OSA的不良预后相关,作者选择将重点放在SQLE上,进一步探索其作为治疗靶标的潜力。
与正常相邻组织和可能的祖细胞(成骨细胞OB和间充质干细胞MSC)相比,OSA中SQLE表达明显增加(图5A、B)。此外,Huvos I/II级的OSA样本中SQLE表达升高,在MTX(甲氨蝶呤)耐药的Saos2细胞系中也观察到相同的趋势(图5C)。SQLE基因与MYC基因在基因组上邻近,并且二者的拷贝数和mRNA表达水平都呈现出强相关性,这可能导致OSA中SQLE和MYC mRNA水平的同步升高(图5D-H)。SQLE因其位于细胞质中的代谢酶特性,相对于位于细胞核内、难以直接靶向的MYC蛋白而言,提供了一个更易于靶向的治疗靶点。
图5. OSA中SQLE的过表达与肿瘤进展有关
Q6:如何进行湿实验验证?
敲除SQLE阻碍了OSA的进展
为了深入研究SQLE对OSA进展的影响,作者在高内源性SQLE蛋白表达的MNNG和U2OS细胞系中进行了SQLE基因的敲除(图6A),随即发现细胞的恶性表型受到了抑制,包括增殖、集落形成、迁移和侵袭(图6B-E)。此外,在不存在和存在药物治疗的情况下,SQLE沉默都会促进细胞凋亡(图6F、G),并增强肿瘤细胞对 MAP方案(甲氨蝶呤MTX、顺铂DDP和阿霉素ADM)的药物敏感性(图6H)。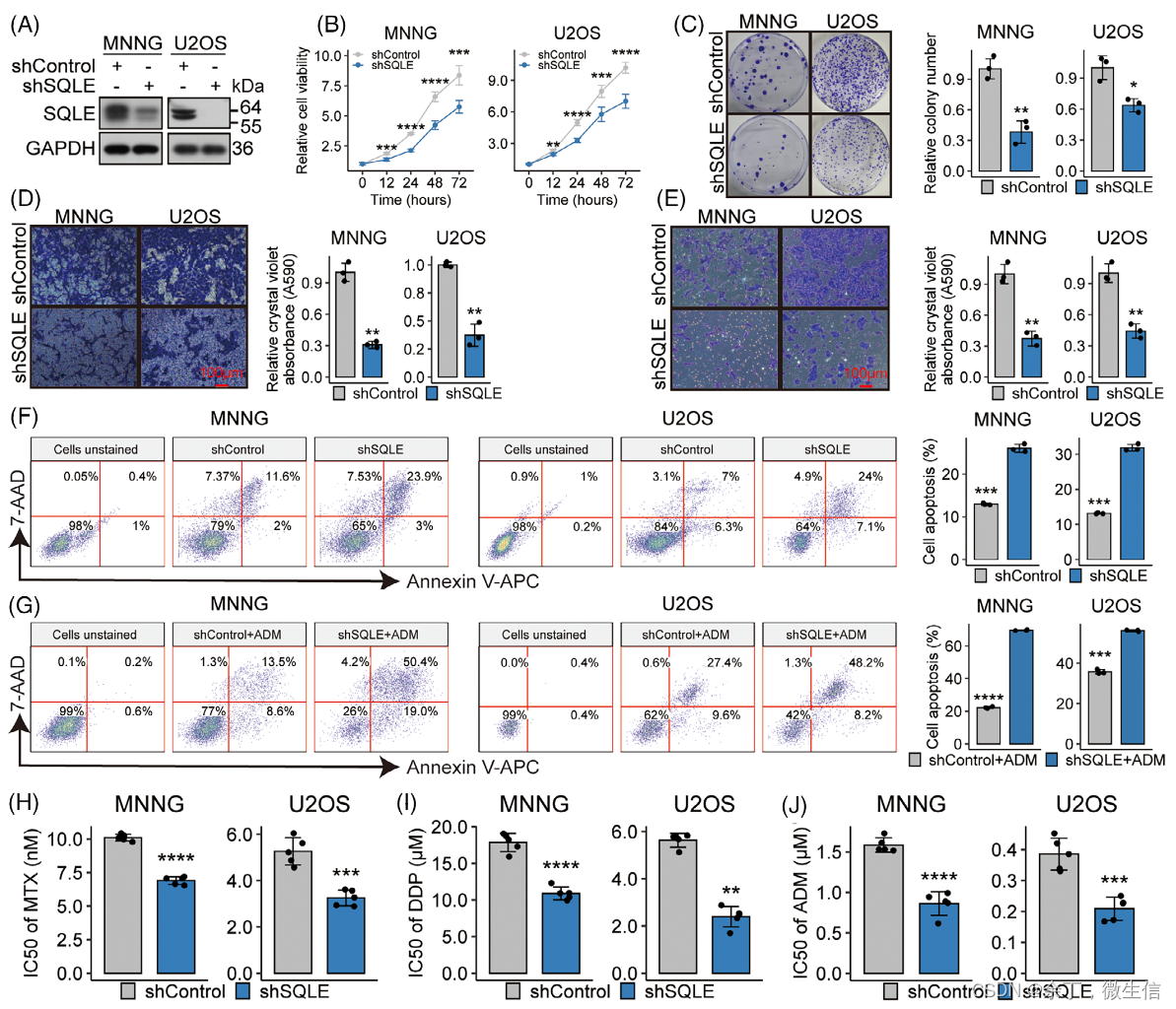
图6. SQLE沉默在体内外均可阻碍OSA的进展
在体内外实验中确认SQLE敲除能有效抑制OSA进展之后,作者进一步探讨了SQLE在分子层面对OSA的作用机制。对敲除SQLE的细胞系进行RNA测序后发现,SQLE沉默导致胆固醇稳态基因组发生显著下调(图7A、B),同时导致PI3K-Akt信号通路的显著富集(图7C)和PI3K-Akt-mTOR通路的显著下调(图7D)。此外,SQLE mRNA水平与三种PI3K/mTOR通路拮抗剂的AAC 值显示出强相关性(图7E),表明SQLE mRNA水平可能与此通路的激活有关。后续利用Rescue实验和蛋白印迹实验证明SQLE沉默降低了细胞内胆固醇水平,导致FAK/PI3K/Akt/mTOR信号通路失活,最终抑制OSA进展(图7F-H)。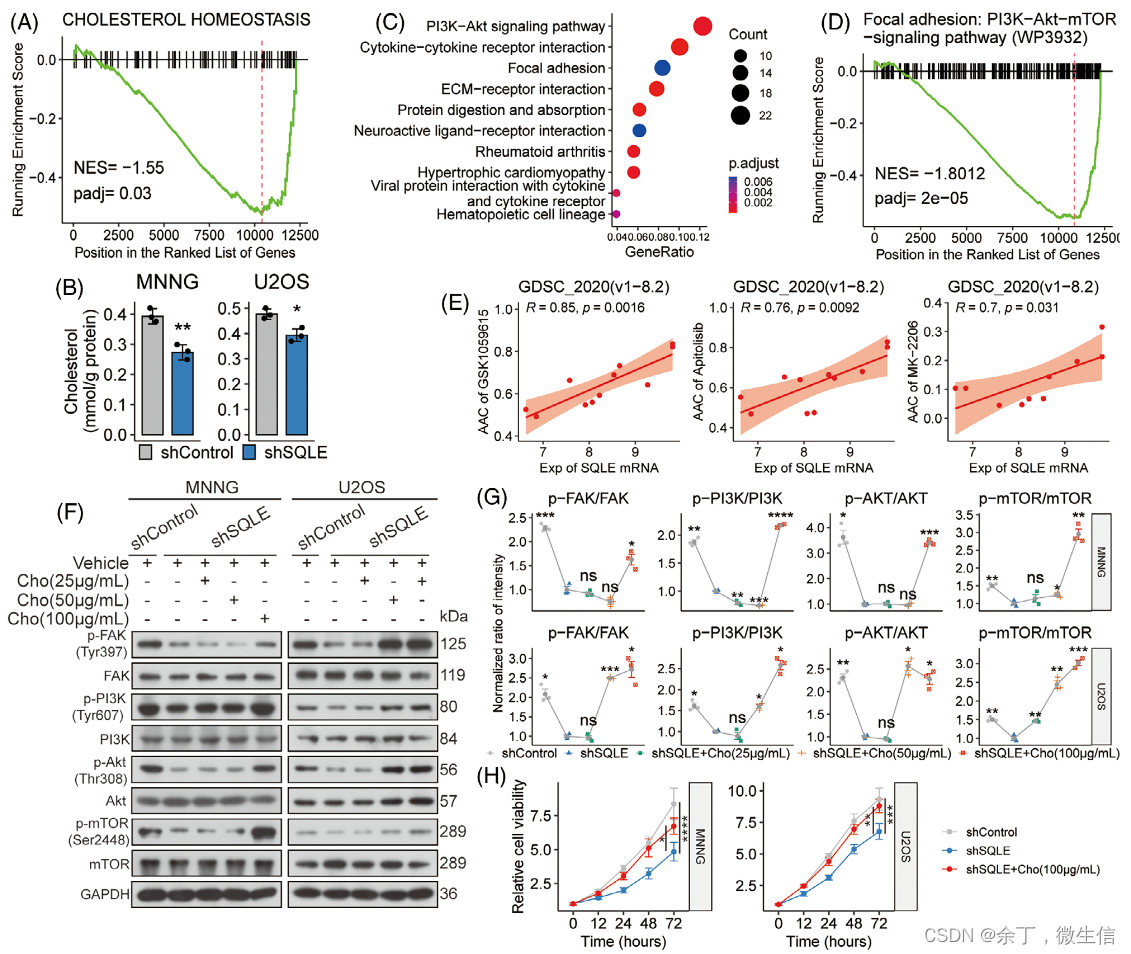
图7. SQLE沉默可通过降低胆固醇和抑制FAK/PI3K/Akt/mTOR通路来阻止OSA的进展
Q7:如何升华文章,将研究成果应用在疾病治疗中?
SQLE抑制剂阻止了OSA的进展并增强化疗效果
鉴于SQLE能够阻碍OSA的进展,作者考虑到SQLE抑制剂可能具有治疗效果。因此,作者采用了哺乳动物SQLE抑制剂FR194738进行实验,结果表明FR194738能显著抑制OSA细胞的增殖和集落形成,并有效降低细胞内胆固醇水平(图8A-H)。此外,FR194738与常用化疗药物特别是DDP联合使用时,抗肿瘤效果增强(图8I)。这些结果表明FR194738通过靶向SQLE不仅单独有效,而且能增强传统化疗药物的疗效,提供了一种可能的新策略来提高OSA患者的治疗反应。这为未来临床试验和治疗方案的设计提供了重要依据。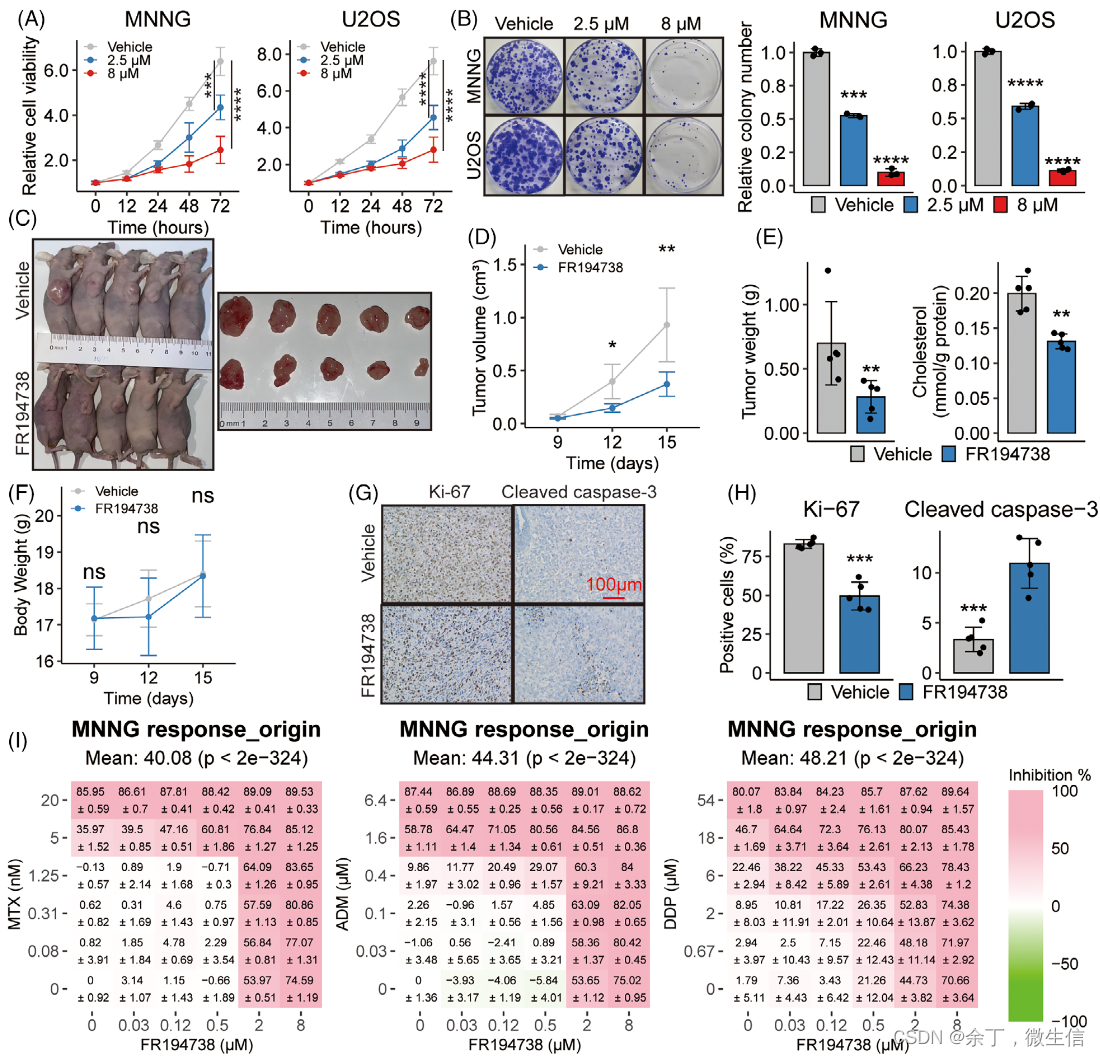
图8. SQLE抑制剂FR194738可抑制OSA进展并增进化疗疗效
亮点结论
- 通过机器学习构建了新的OSA预后标志物AIDPI,用于区分胆固醇代谢失调的高风险OSA患者。
- 抑制SQLE能够阻碍OSA的进展,其机制是通过减少胆固醇的生成和抑制FAK/PI3K/Akt/mTOR信号通路。
- 在体内实验中,SQLE抑制剂能够抑制OSA的进展,并增强化疗的效果。
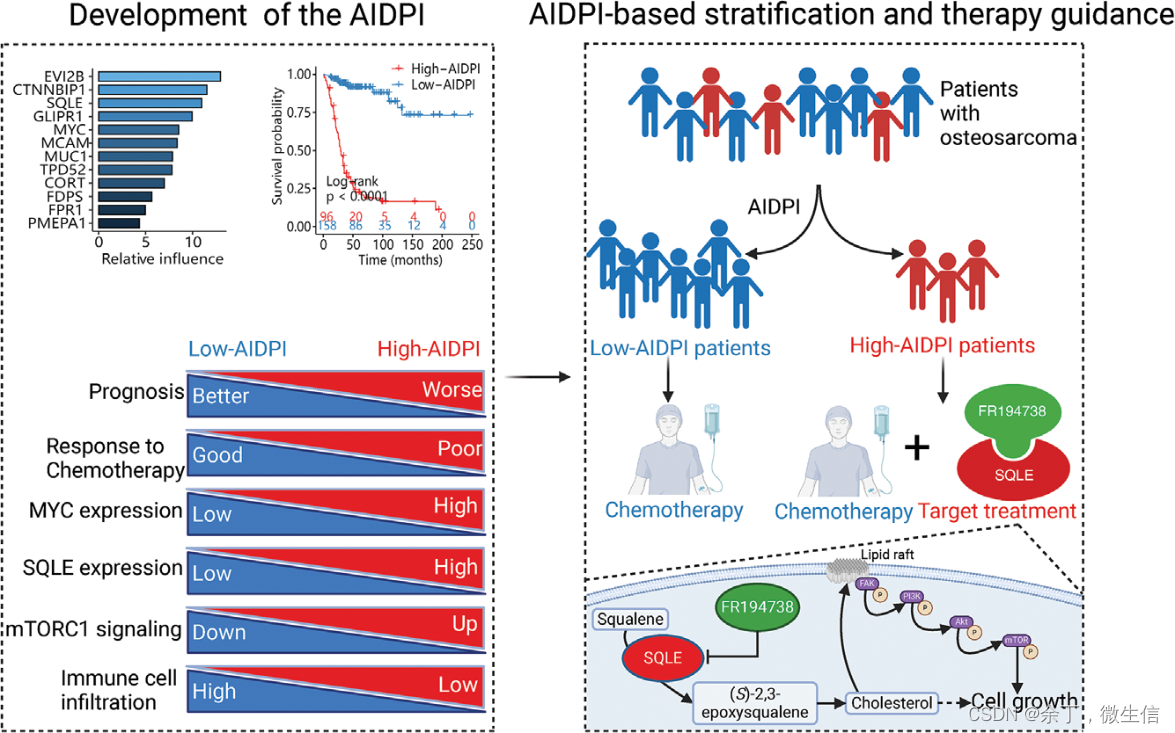
本篇文章思路明确,行文结构清晰。由此推而广之,当将此思路应用于类似问题时,我们需要考虑以下几个方面以优化研究方法(Call Back):
- 适用范围:该研究方法主要适用于肿瘤分析。针对非肿瘤疾病,由于缺乏生存状态和生存时间的信息,应用思路时需适当调整替换分析方法。
- 数据获取:如何有效地获取所需数据,如何整合和利用多个数据集;
- 分析方法的运用:考虑多种数据分析方法,最大化机器学习在特征选择中的潜力,获得可靠的分析结果;
- 模型性能与解释力:优秀的模型不仅表现出卓越的预测性能,还能帮助发现并且解释不同表型(计算角度)之间的生物学差异;
- 综合分析的深度与广度:结合粗、细粒度(Bulk Seq + Single cell Seq),多维度(snATAC + scRNA)分析,深入挖掘分子层面的差异;
- 结果展示:清晰展示研究成果,特别是将新发现的靶点基因与疾病联系在一起。
- 实验辅助:湿实验结果验证,增强研究说服力;药物敏感性实验,升华文章价值。
本文亮点在于生信分析和实验设计的结合,小伙伴们如果对这类分析思路的文章感兴趣,对上述问题有了较为深刻的理解,请不要错过机会,抓紧时间联系我们吧!纽科生物同您一起,前瞻未来的科学发现!
作者写在最后:
非常感谢纽科生物对我的文章做出的解读,并邀请我在这分享一些心得。毕业季的气氛中,我也有诸多感慨和回忆想要分享。
相比上海骨科创新材料领域的其他研究者来说,我的科研成果几乎微不足道。尽管如此,我对自己感到满意,因为我已竭尽全力。每个人的起点不同,可调动的资源也各不相同,短期内的成果自然千差万别。我的满意并非源于文章本身,而是因为我证实了自己的想法是行得通的,我找到了自己的道路。《道德经》中说:“有道无术,术尚可求;有术无道,止于术。”这句古训根据个人经历有着不同的解读。
2017年,我被推免为骨科学术型硕士,但不幸的是,我在临床工作了近2年半,我的实验技能仍停留在本科水平。然而,在不幸中有幸,我有机会在全国最大的骨肿瘤中心之一学习,遇到了许多同龄的骨肉瘤患者。或许是由于我天生的同理心,我渴望找到有效的治疗方法来帮助这些患者,这成了我研究的初衷。因此,我果断将研究方向从骨质疏松转向骨肉瘤。
作为新手的我,所在的课题组也缺乏研究方向和经验。幸运的是,我的导师具有丰富的临床经验,他设计了一套利用临床、影像等指标预测骨肉瘤耐药性的方法,这启发我将这些指标扩展到两万多个个mRNA的表达量,开展数据驱动的研究。这就是我当时找到的“道”。有了这一明确的研究方向,我便开始学习必要的技术。我也曾尝试过建立自己的骨肉瘤多组学数据库,奈何人微言轻,能调动的资源实在有限。我只能转而学习R语言,提升数据挖掘能力,利用公开的数据集开展自己的研究。从2021年开始,我通过生信技能数的在线课程逐步提高自己的生信分析技术,直到2024年2月份才得以发表这一篇文章。尽管有人质疑使用公开数据的价值,也有人认为SQLE在很多肿瘤中都被报道过,缺乏创新性,我却对自己的研究持有不同看法。
我不认同仅追求热门技术的研究方式,也不理解为何“缺乏创新性”的观点会存在。我从未对任何技术抱有过盲目的热情,因为我相信,我们医生的研究应以解决临床问题为核心,将问题拆解成基础科学问题,如果能用最最简单且成熟的技术回答这些问题那就再好不过了。我认为一个研究的创新性在于,我回答了这个问题,且比前人做得更好,这就足够创新。我认为研究中最重要的是逻辑思辨和自我诘问,是条理清晰地讲述自己的故事,这也是研究的乐趣所在。
我相信,每位直接面对患者的医生都有自己的“道”。只要时间足够,他们一定能学会足够的“术”来解决这些问题。如果临床工作太忙,找到一个可靠的团队合作也是一个有效的解决方案。


