- 1CTF学习路线指南(附刷题练习网址)_ctf wargame
- 2计算机发展史_1957年电子管计算机
- 3html接入百度地图
- 4Bugku CTF---where is flag 5_bugkuwhere is flag 5
- 5Swin Transformer——披着CNN外皮的transformer,解决多尺度序列长问题
- 6MQTT入门指南(一)ubuntu16.04上安装MQTT服务端_ppa:mosquitto-dev 不存在
- 7分布式限流——Redis + Lua实现滑动窗口算法
- 8oracle 去掉末尾四位_oracle去掉后四位
- 9微信支付指纹上传服务器,指纹安全吗,支付宝、微信支付会上传用户的指纹吗...
- 10中国互联网20年简史(1998-2018),告诉你本质是什么、规律是什么
生信项目之NCBI下载数据集详解
赞
踩
目录
一、NCBI简介
NCBI(National Center for Biotechnology Information)的中文名称是美国生物信息技术中心,该网站是美国医学图书馆(NLM)的一部分。National Center for Biotechnology Information (nih.gov)
该网站提供了下载数据集、上传数据集、在线分析数据集以及提供了部分生物信息学工具如(BLAST:一款序列比对工具)等功能。本文主要介绍如何在NCBI上下载数据集。
二、SRA数据集的下载
SRA数据集的下载是指通过其他学者上传好的SRA编号直接下载相应的数据集以用于学习实验等。
SRA(The Sequence Read Archive)数据库是NCBI旗下用于存储高通量测序数据的数据库。
SRA数据集的下载方式有很多种,在这列举了最常用的几种方法:直接在NCBI官网进行搜素下载;利用NCBI提供的工具SRA-Toolkit下载;利用Python网络爬虫进行下载。
2.1利用NCBI官网进行搜索下载
论文中的作者可能会提供SRA号在论文的Dataset部分,当我们获得此SRA号之后我们可以在NCBI的搜索栏中直接输入该SRA号。本文以序列号SRR5908499为例。
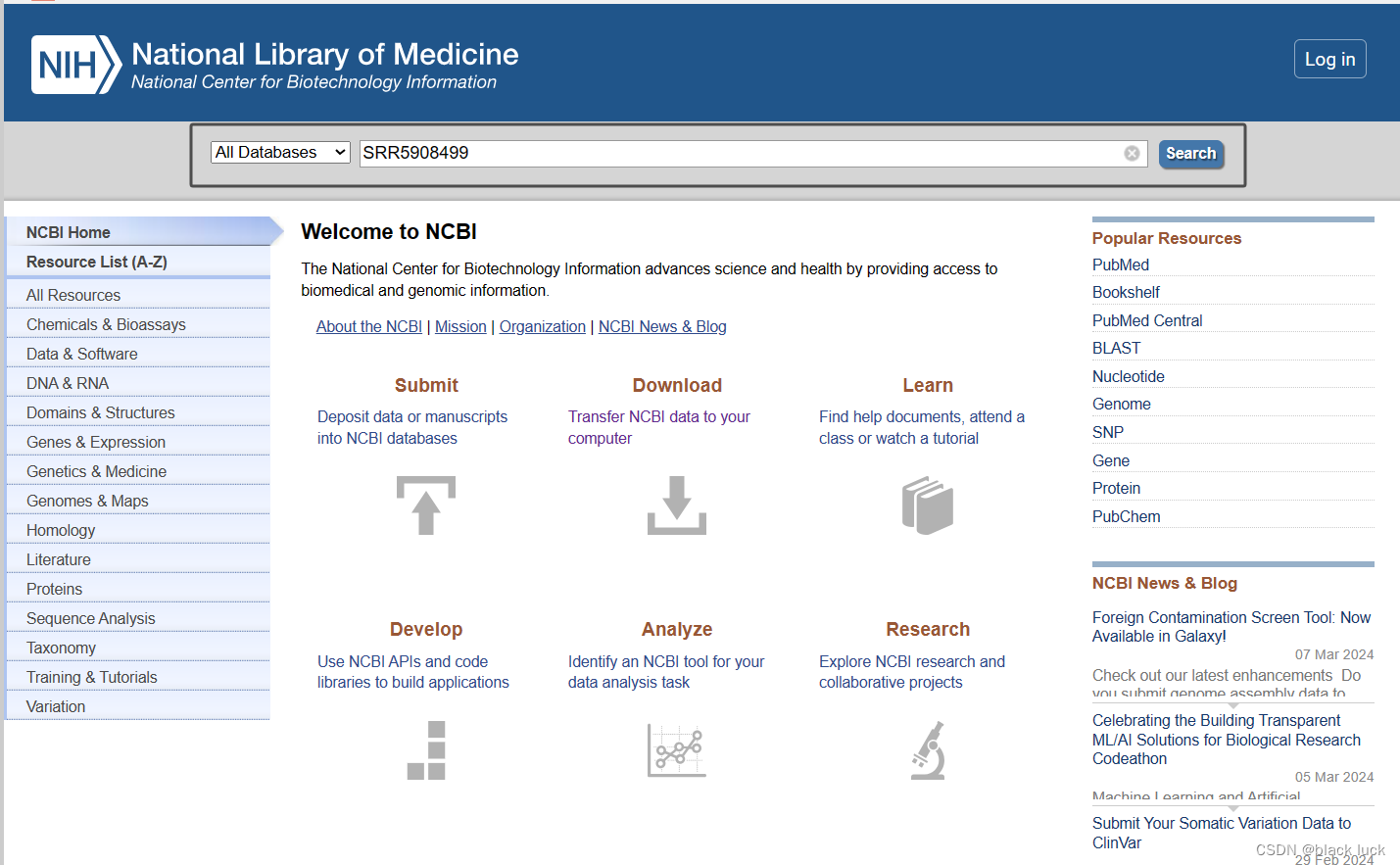
点击查询(search)后即可出现该SRA文件相关的生物体信息。
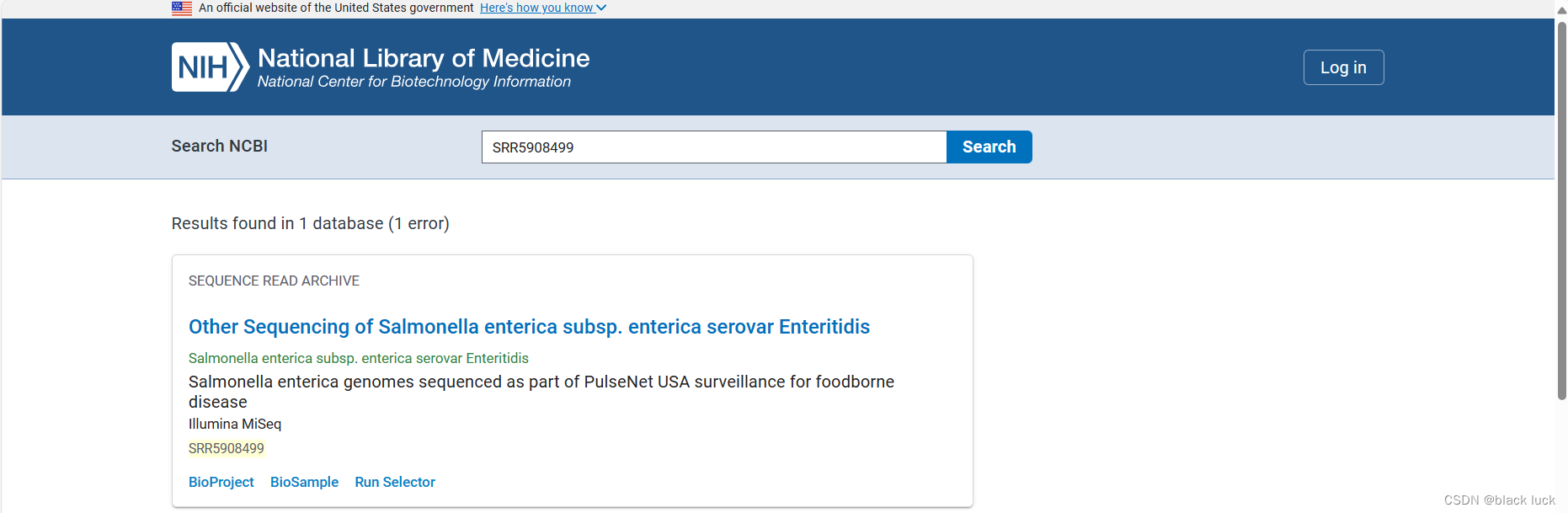
然后点进该生物体,则可以看到完整的信息,然后点击该SRA文件。
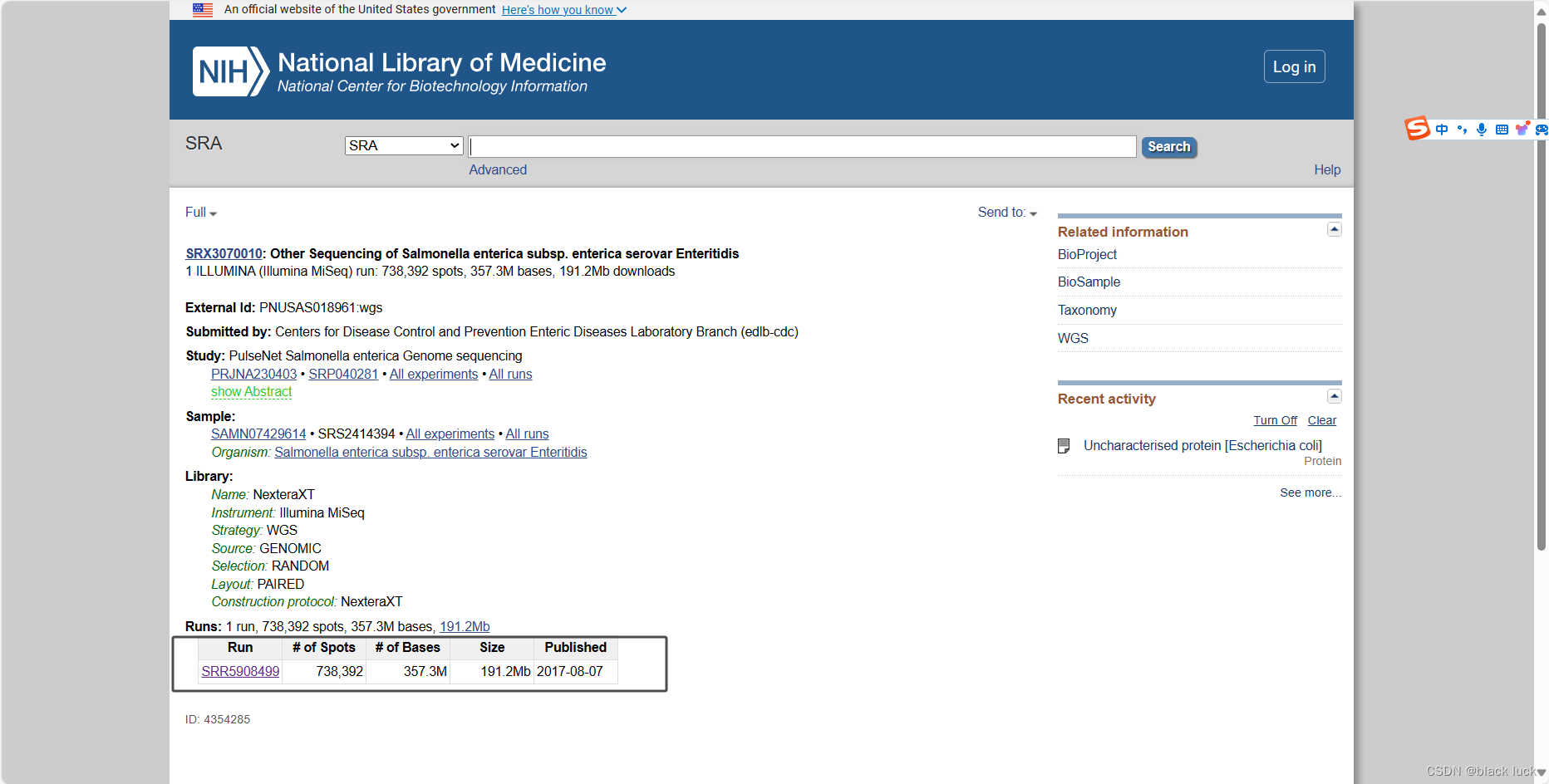
即可出现该SRA文件的详细介绍,点击上方的download即可下载该文件。
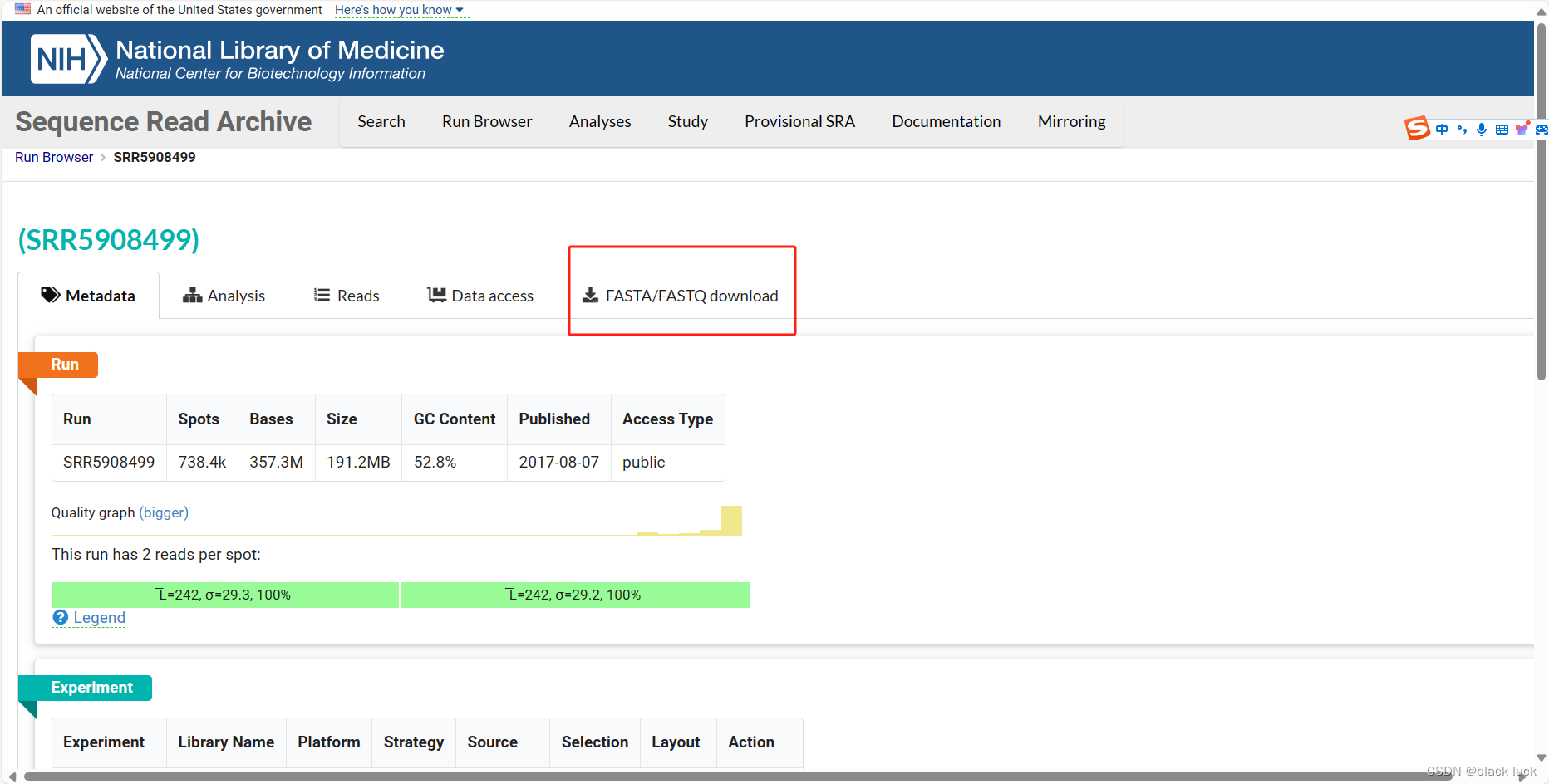
2.2利用NCBI提供的工具SRA-Toolkit进行下载
NCBI官网中提供了SRA-Toolkit工具的下载方式,当然也可以通过github连接进行下载。https://github.com/ncbi/sra-tools
再该GitHub连接中有详细的工具介绍和该工具的安装及使用步骤的等,在这里只做简单的介绍。
①下载单条SRA文件的命令
如下为linux系统下的命令,在你想要保存的文件路径下输入一下命令即可下载单条SRA文件。
prefetch SRR5908499②下载多条SRA文件的命令
将你要下在的SRA文件的序列号保存在你的txt文件中,如例保存在ALLSRA.txt文件中下载。如下为Linux系统下输入的命令。
prefetch --option-file ALLSRA.txt更多的SRA-Toolkit工具箱的功能请见GitHub官网。
2.3利用Python网络爬虫进行下载SRA文件
通过编写代码块的方式进行实现
三、通过网页搜索的形式下载数据集
NCBI可以通过检索物种信息、txid、accession等形式下载数据集。
3.1通过检索物种信息下载数据集
这里以检索bacteria,细菌物种为例。
首先在搜索栏输入bacteria。
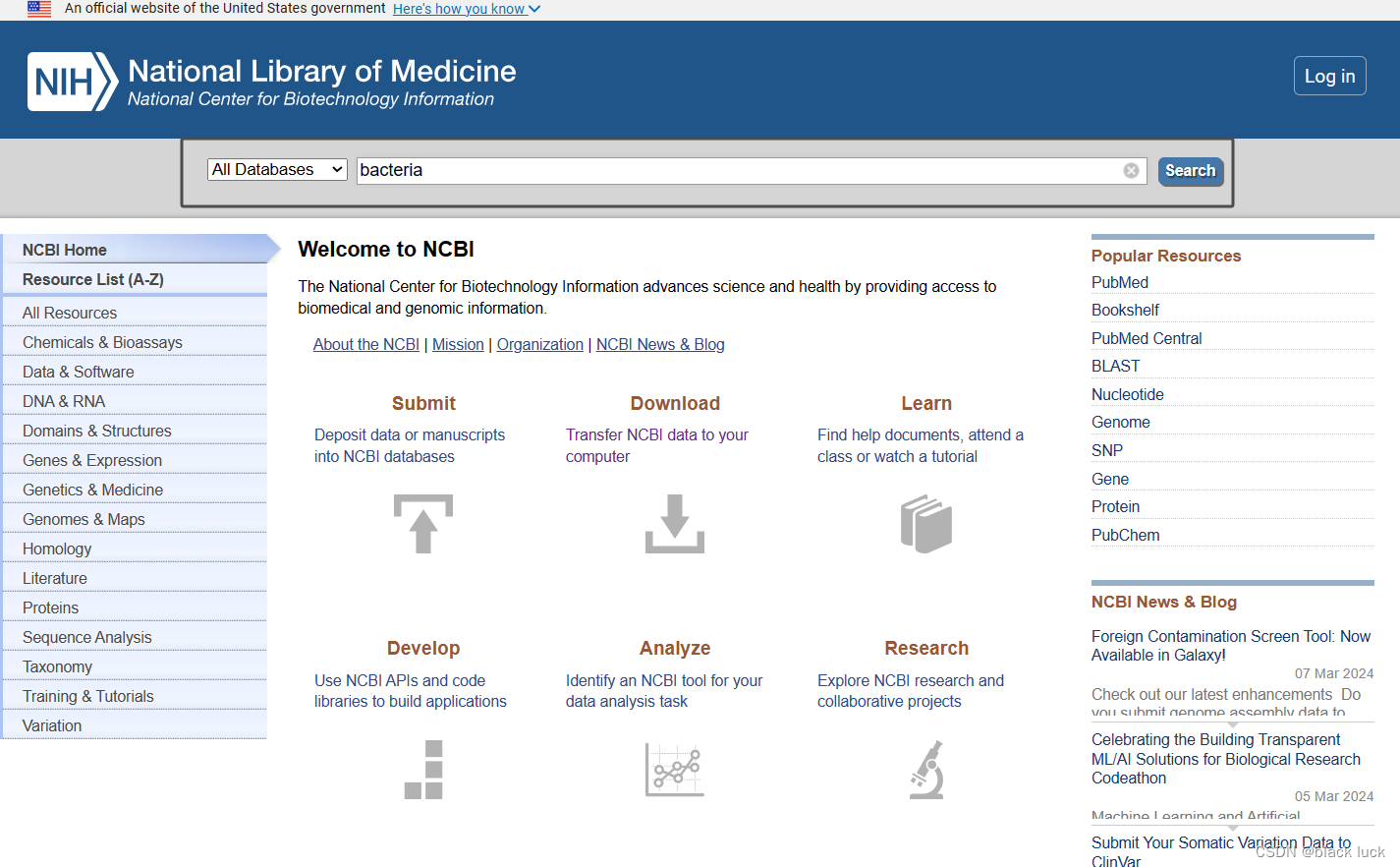
然后选择打算使用的数据库,NCBI官网会提供多种数据库,这里我们选用的是核苷酸序列的assembly数据集,assembly存储的大多是经过验证过的数据集,可以保证一定的真实性。
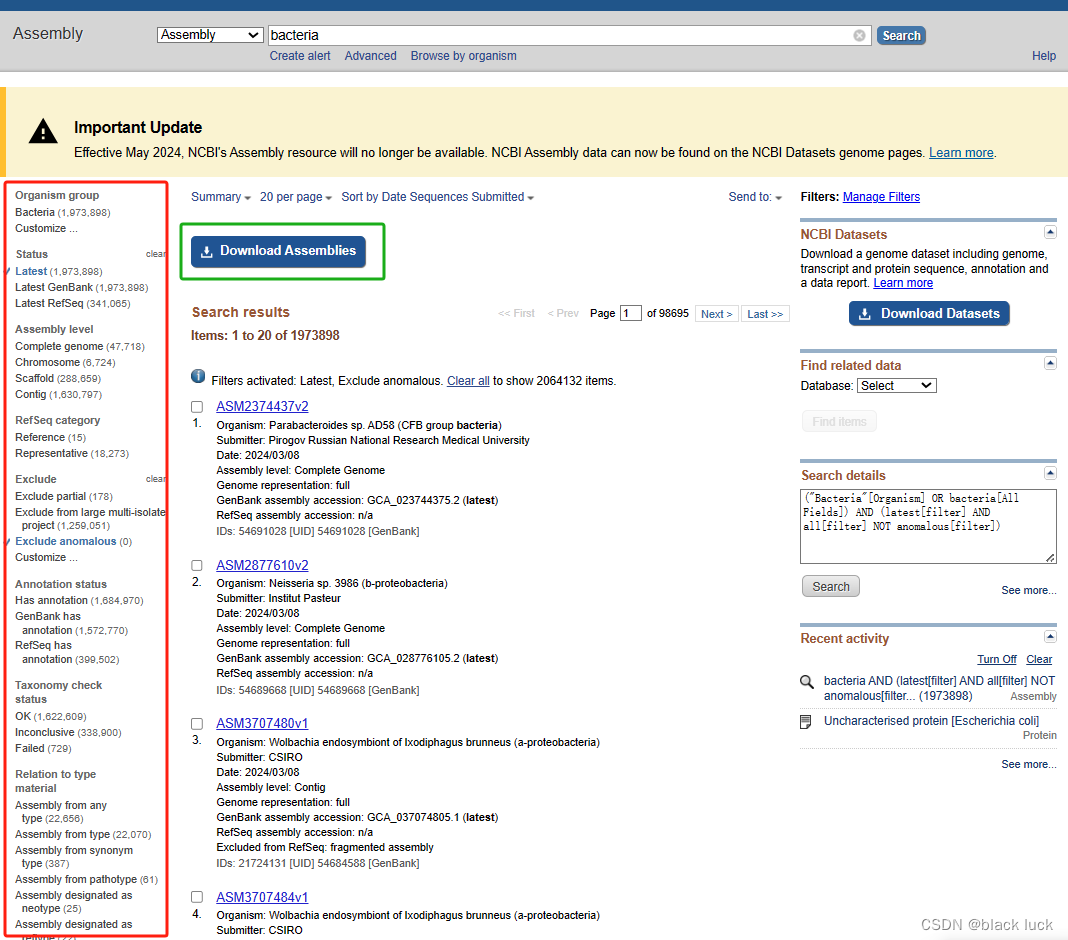
左侧功能栏可以按照数据集的需求进行选择。中间也可以选择需要下载的数据,或选择全部下载,然后点击上方的下载即可下载数据集。
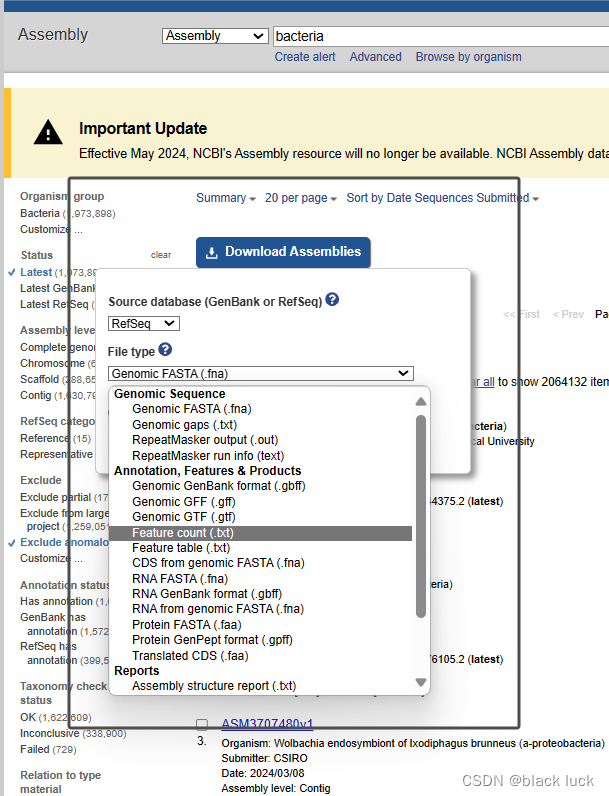
点击下载数据集后即可选择需要保存的格式选择需要下载的路径进行下载所需的数据集。
3.2利用txid进行下载数据集
txid指物种的分类索引号,这里以txid=34为例,首先在搜索栏输入taxid号。

然后点击进入txid为34的板块
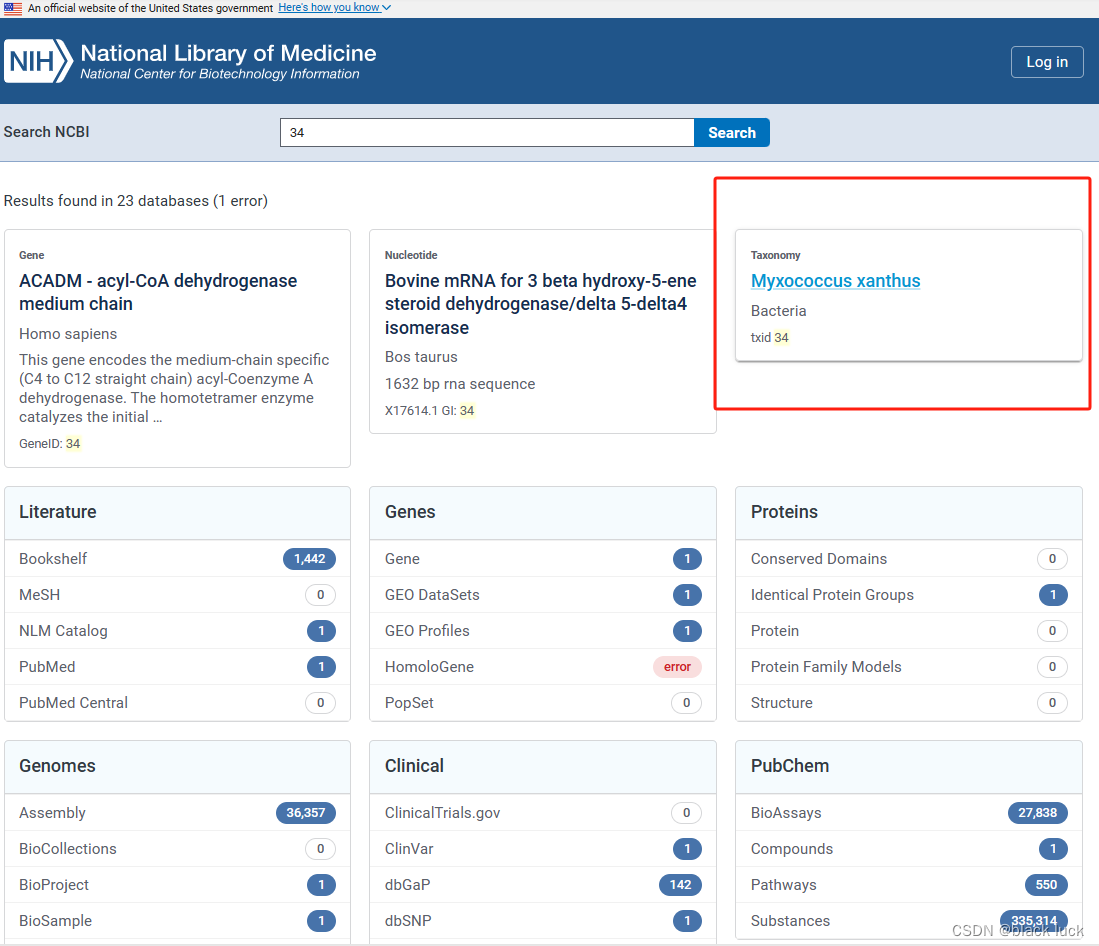
进入后即可按照上述方法进行下载该数据集。
3.3利用accession号进行下载数据集
accession号指生物体的索引号,通过该索引号可以下载到该生物体的序列信息等信息。
方法如上,这里就不做过多解释了。
其他形式的下载方法雷同,若要下载多条txid号或accession号的数据集,可以通过工具下载或者Python爬虫的方式进行下载数据集,首先将需要下载的索引号放入txt文件中进行下载。下面我将介绍如何使用工具的方法进行下载数据集,Python爬虫的方法可以在CSDN或GitHub中搜索获得
四、使用NCBI提供的下载工具下载NCBI数据集
使用NCBI-genome-download下载数据集,该工具可在如下github连接中进行下载。GitHub - kblin/ncbi-genome-download: Scripts to download genomes from the NCBI FTP serversg
该Github连接中也详细写有如何下载、安装、使用该工具进行数据集下载,这里不再过多赘述。使用方法类似于SRA-Toolkit工具的使用方法。
五、总结
这三种方法是目前比较常用的下载数据集的方法,当然还用其他很多下载数据集的方法,具体的下载数据集的方法要按照该生信项目的需求进行下载。



