- 1超实用的30个 Python 案例,建议收藏_python学习案例
- 2Python小白(python数据分析与可视化)从入门到精通_python小白(python数据分析与可视化)从入门到精通-csdn博客
- 3meanshift算法图解以及在图像聚类、目标跟踪中的应用_灰度图像聚类算法
- 4深度学习算法中的深度强化学习(Deep Reinforcement Learning)_deep reinforcement learning for strate
- 5Logback快速实践_ch.qos.logback.core.rolling
- 6微信小程序使用wxs实现购物车贝塞尔曲线_小程序贝塞尔曲线广告
- 7数据结构试卷及答案(七)
- 8Linux和Hadoop常用命令操作_linux删除hdfs中的文件命令
- 9后台语言返回的二进制流音频文件在微信浏览器兼容性问题_html5的audio标签播放二进制文件流
- 10树的高度与深度
项目集锦 | 易基因DNA羟甲基化5hmC测序研究成果_5hmc-seq
赞
踩
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
羟甲基化5hmC是哺乳动物基因组上的第六碱基,在发育、衰老、神经退行性疾病、复杂疾病及肿瘤发生过程中起重要作用。本期以科研内容为序,总结近期易基因羟甲基化合作研究成果。
01 宫颈癌 WGBS + oxWGBS

标题:An epigenomic landscape of cervical intraepithelial neoplasia and cervical cancer using single-base resolution methylome and hydroxymethylome
发表时间:2021年7月19日
期刊:Clinical & Translational Medicine
影响因子:11.492
技术方法:WGBS、oxWGBS、RNA-seq

研究思路:

研究摘要:
本研究采用oxBS-seq方法绘制从健康宫颈到CIN到CC样本在单碱基分辨率下的全基因组5mC/5hmC图谱。分析CC进展过程中差异甲基化区域(DMR)和差异羟甲基化区域(DhMR)变化趋势。GO/KEGG富集分析与DMRs相关基因 (DAGs) 和DhMRs相关基因 (DhAGs)。个性化分析DAGs和DhAGs甲基化和羟甲基化水平与基因表达的关系。多组学综合分析甲基化/羟甲基化与基因组变异的相关性。对甲基化/羟甲基化与组蛋白修饰关联分析。通过多组学分析证实了甲基化/羟甲基化与宫颈癌发生中的 CNV 无关,且甲基化/羟甲基化的发生远早于CNV。确定了八个预后相关基因,可以作为CC治疗的新靶点,这为表观遗传学在肿瘤发生和进展中的筛查诊断及预后提供了重要数据支持。
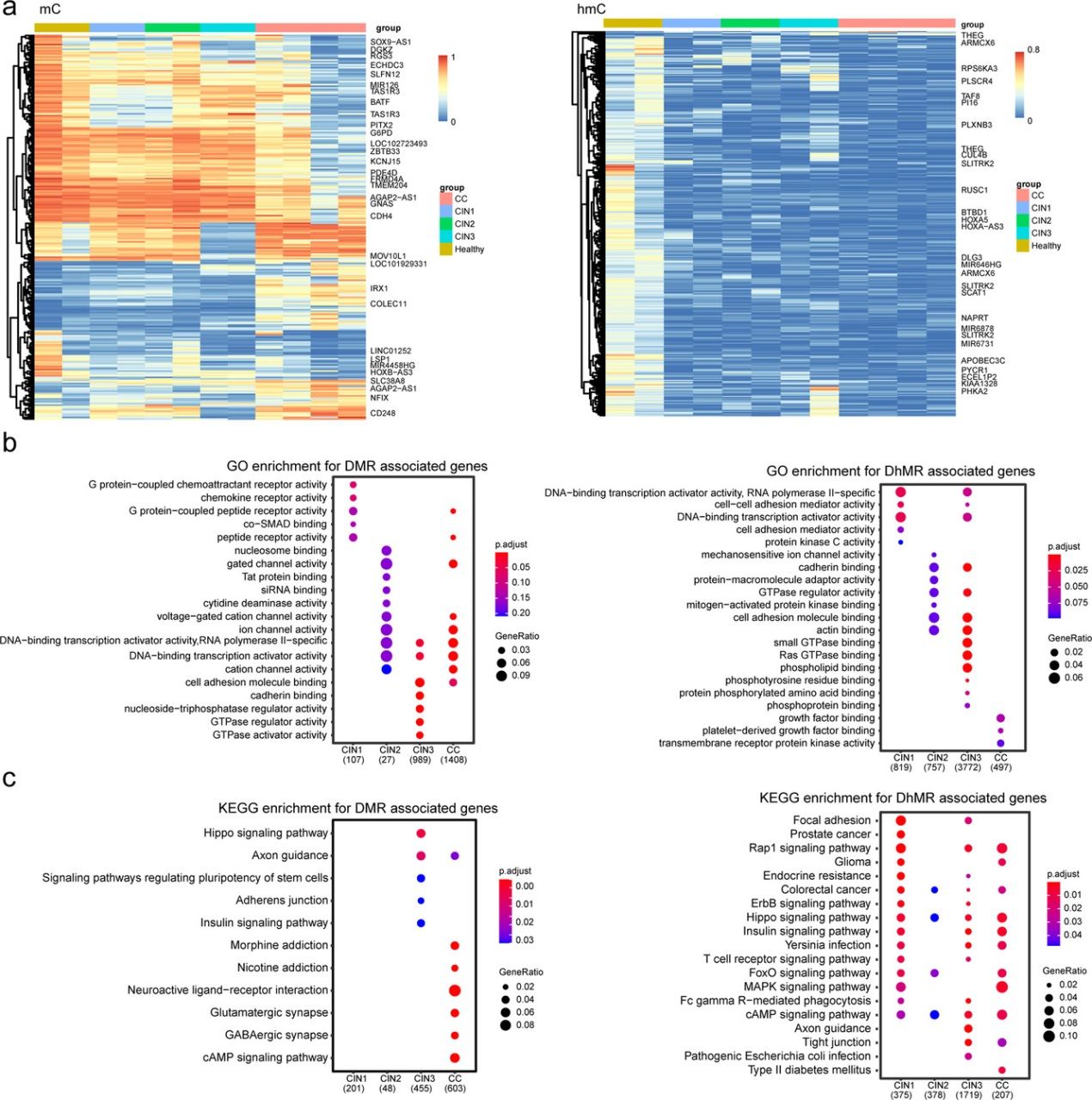
图:基于DMRs和DhMRs的功能相关基因
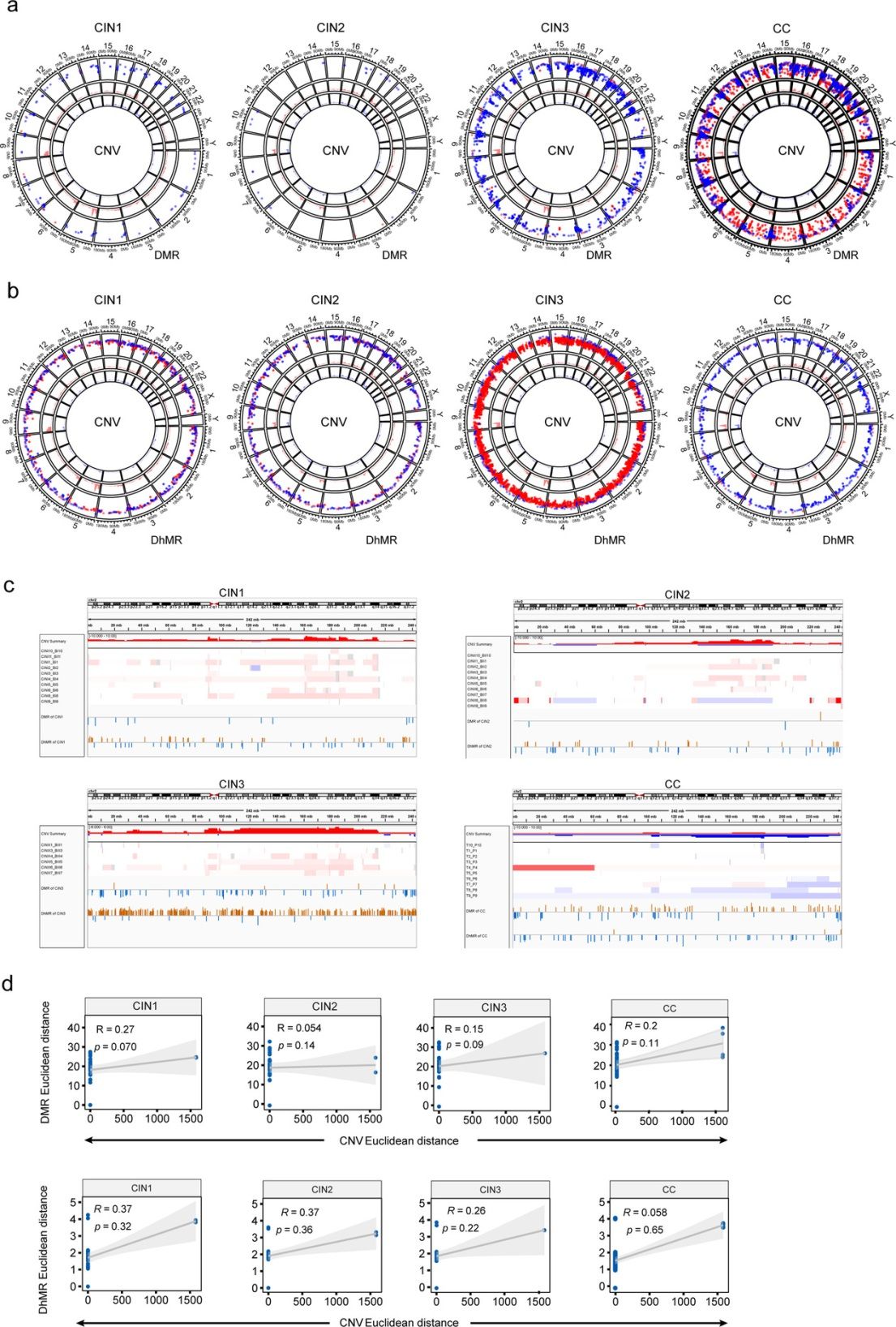
图:甲基化/羟甲基化与基因组变异的整合分析(结合CNV数据)
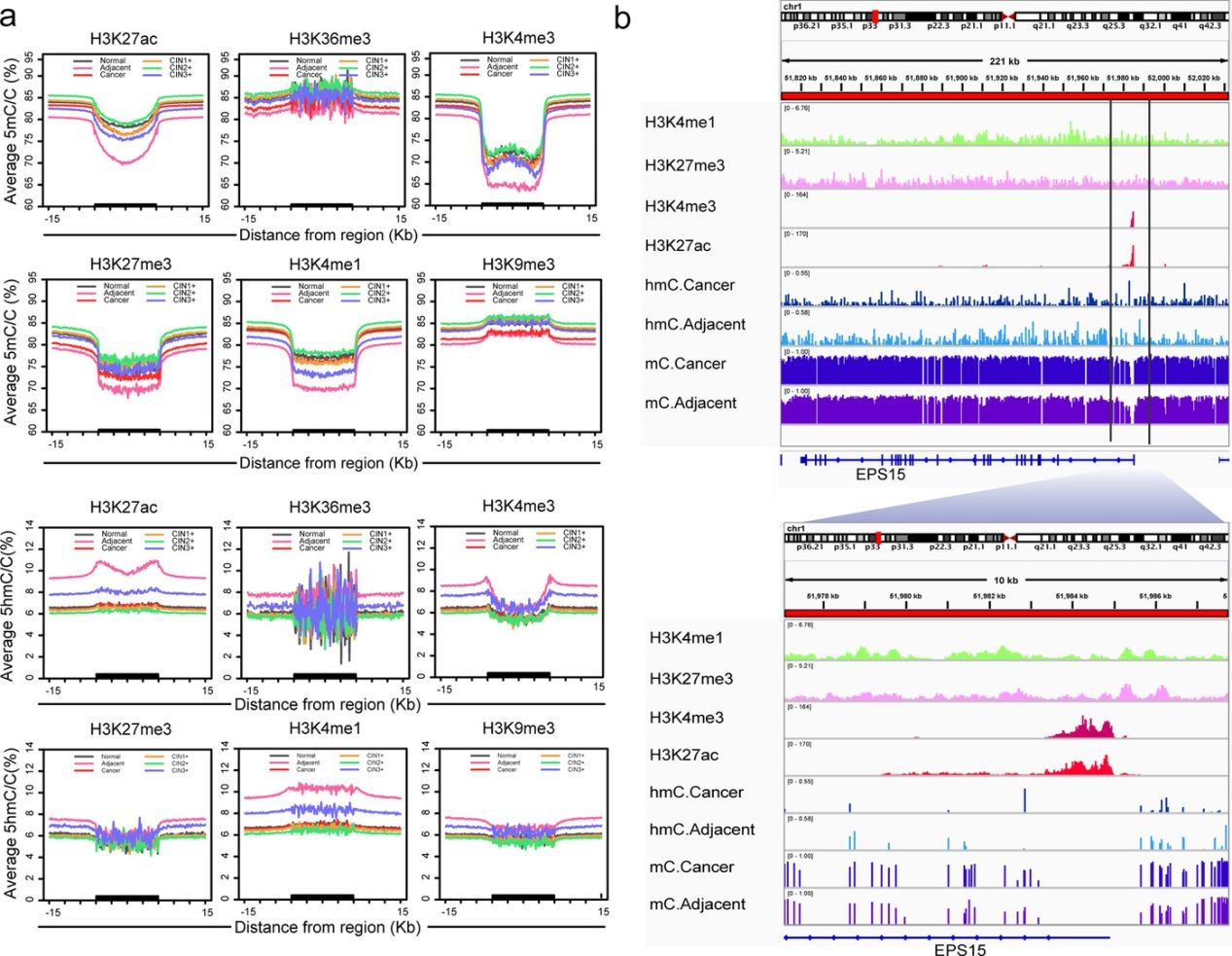
图:甲基化和羟甲基化与组蛋白修饰的整合分析(结合ENCODE数据)
02 牦牛发育:oxRRBS、RRBS

标题:Genome-Wide DNA Methylation and Hydroxymethylation Changes Revealed Epigenetic Regulation of Neuromodulation and Myelination in Yak Hypothalamus
发表时间:2021年9月27日
期刊:FRONT GENET
影响因子:4.599
技术方法:oxRRBS、RRBS
研究摘要:
5-甲基胞嘧啶 (5mC) 和 5-羟甲基胞嘧啶 (5hmC) 都是神经发育中重要的表观遗传修饰。然而很少有研究在自然高海拔条件下鉴定动物大脑区域的全基因组5mC和5hmC模式。本研究利用RRBS+oxRRBS技术鉴定牦牛和牛的大脑、脑干、小脑、下丘脑的基因组5mC和5hmC位点,绘制全基因组DNA甲基化和羟甲基化图谱,并进行DNA甲基化/羟甲基化差异化分析和对应转录组的关联分析。发现牦牛和牛的下丘脑和其他大脑区域的5mC和5hmC存在显著差异,鉴定出差异甲基化区域(DMR)和差异羟甲基化区域(DhMR),其中大多数彼此重叠。最后,验证了DMRs和DhMRs调控的差异表达基因(DEGs)可能在神经调节和髓鞘形成中发挥重要作用。总之结果表明,5mC和5hmC介导的表观遗传调控可能广泛影响下丘脑的发育及其生物学功能,有助于提高高海拔条件的生理适应性。
材料方法:
从Riwoqe牦牛和三江牛(牛)中各选取三只54个月大的母牛,所选取的母牛在过去三代中没有直接或间接的血缘关系,在当地私人农场以同样的饮食喂养,牦牛和牛的平均活重为188.3公斤和163.0公斤。在饿一天后进行人道处死,解剖后迅速分离大脑、脑干、小脑和下丘脑,从中各收集1 cm3样本在液氮中快速冷冻并在-80°C下储存直至RNA和DNA提取。
关键图形:
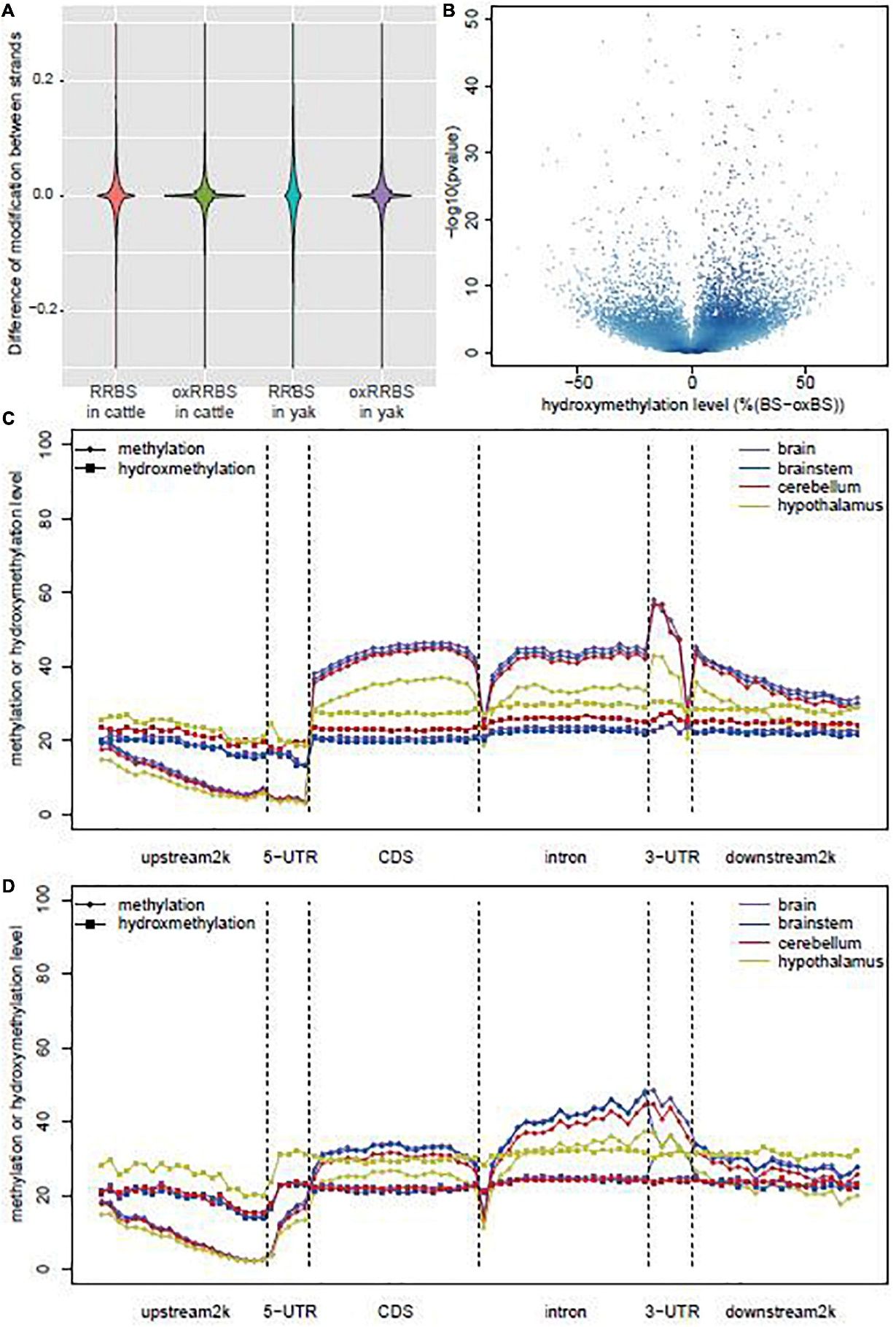
图:RRBS+oxRRBS绘制牦牛和牛的大脑、脑干、小脑和下丘脑DNA甲基化和羟甲基化图谱
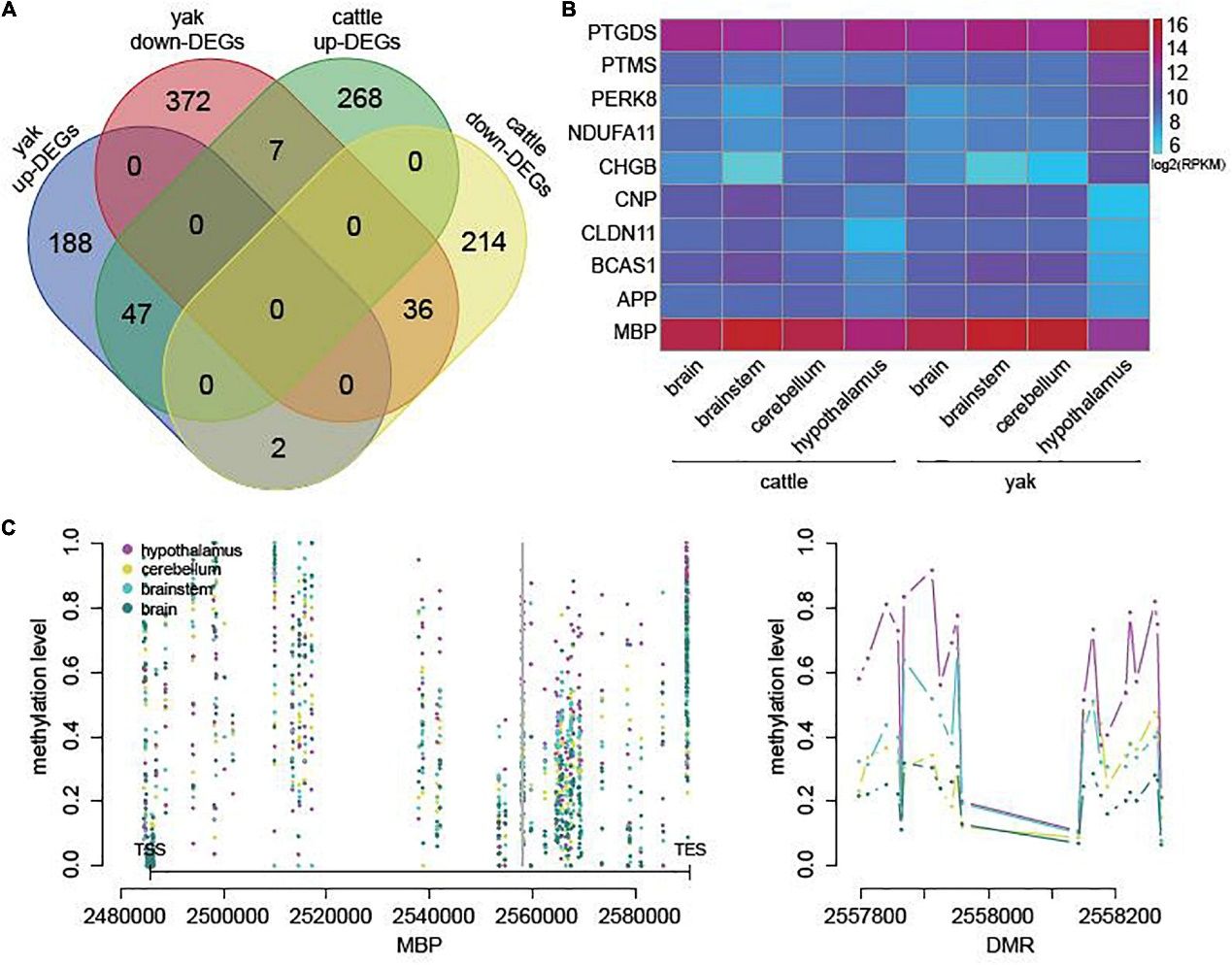
图:受DMR或DhMR调控基因
03 非小细胞肺癌 MeDIP-seq、hMeDIP-seq

标题:A 17 gene panel for non-small -cell lung cancer prognosis identified through integrative epigenomic-transcriptomic analyses of hypoxia-induced epithelial–mesenchymal transition
时间:2019
期刊:Molecular Oncology
影响因子:IF 6.603
技术平台:MeDIP-seq、hMeDIP-seq、ChIP-seq、RNA-seq
摘要:
研究人员分别对在正常和缺氧条件下的非小细胞肺癌(NSCLC)细胞系进行全基因组表观遗传修饰和转录表达测序分析,分析显示低氧诱导A549和HCC827细胞的上皮间充质转化(EMT)以及表观遗传修饰和转录基因表达均呈现动态改变。最后,研究人员确定了17个基因组成的panel,包括参与EMT、缺氧反应、糖代谢和表观遗传修饰基因。这17个gene panel能够清楚地将非小细胞肺癌患者分为三组,三组NSCLC患者的总生存期存在明显差异。本研究可能可以作为进一步选择非小细胞肺癌基因标志物的基础,对NSCLC患者的预后分层具有潜在指导作用。

研究结果:
(1)缺氧诱导非小细胞肺癌细胞EMT

Figure 1:
(A)Wersten Blot验证缺氧诱导因子表达(HIF)
(B)RT-PCR验证血管内皮因子表达(VEGFA)
(C)细胞形态显微观察
(D)Wersten Blot检测其他EMT特征蛋白的表达
(E、F)肿瘤细胞迁移实验
(2)与缺氧反应、EMT和糖代谢相关的广泛基因表达变化
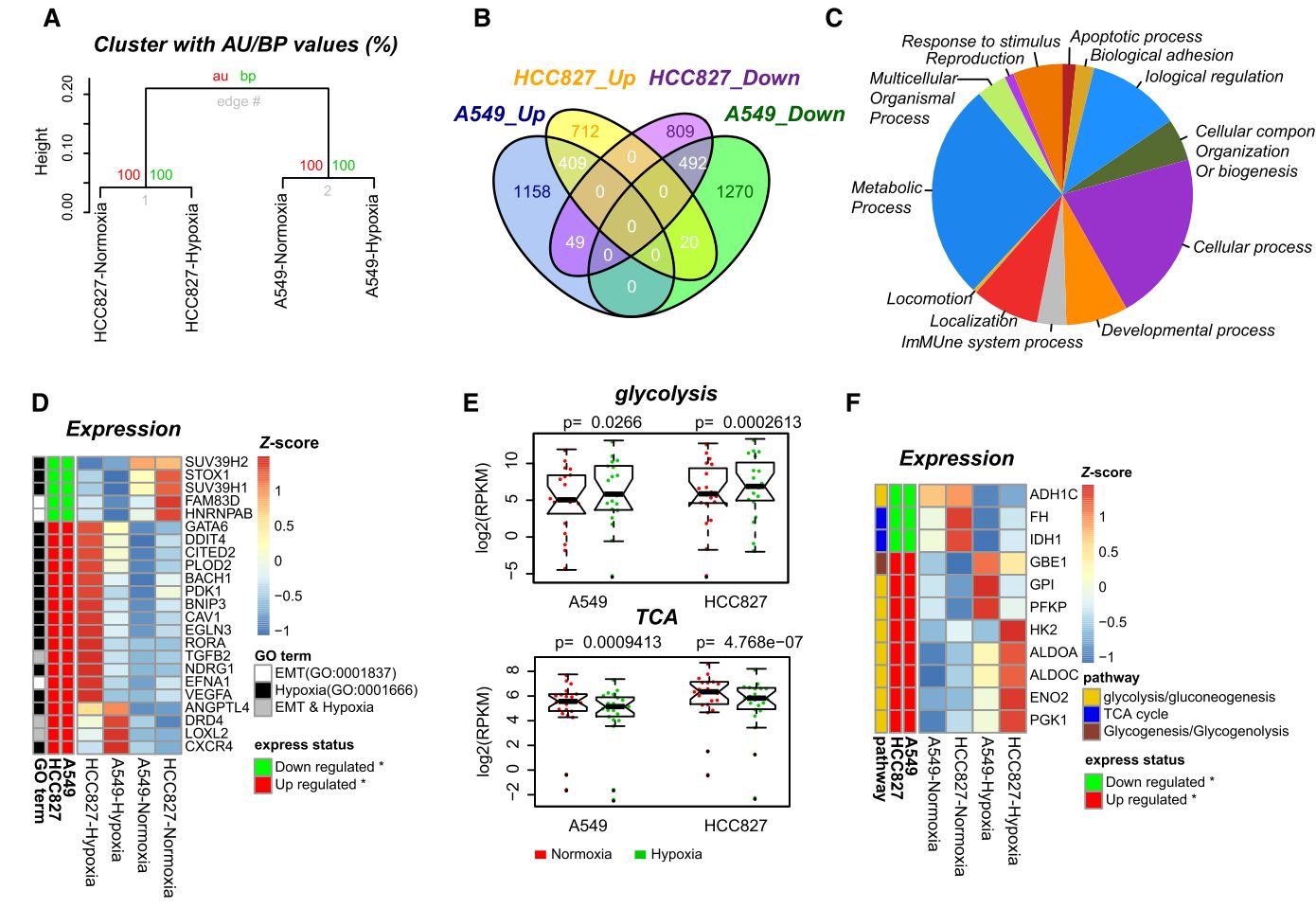
Figure 2:
(A)RNA-seq表达谱聚类分析
(B)上下调DEG重叠分析
(C)GO分析
(D)注释到EMT和缺氧功能的DEG的表达热图
(E)糖酵解和TCA循环通路基因表达差异
(F)糖代谢DEG表达热图
(3)NSCLC细胞中与基因表达相关的表观遗传修饰的差异
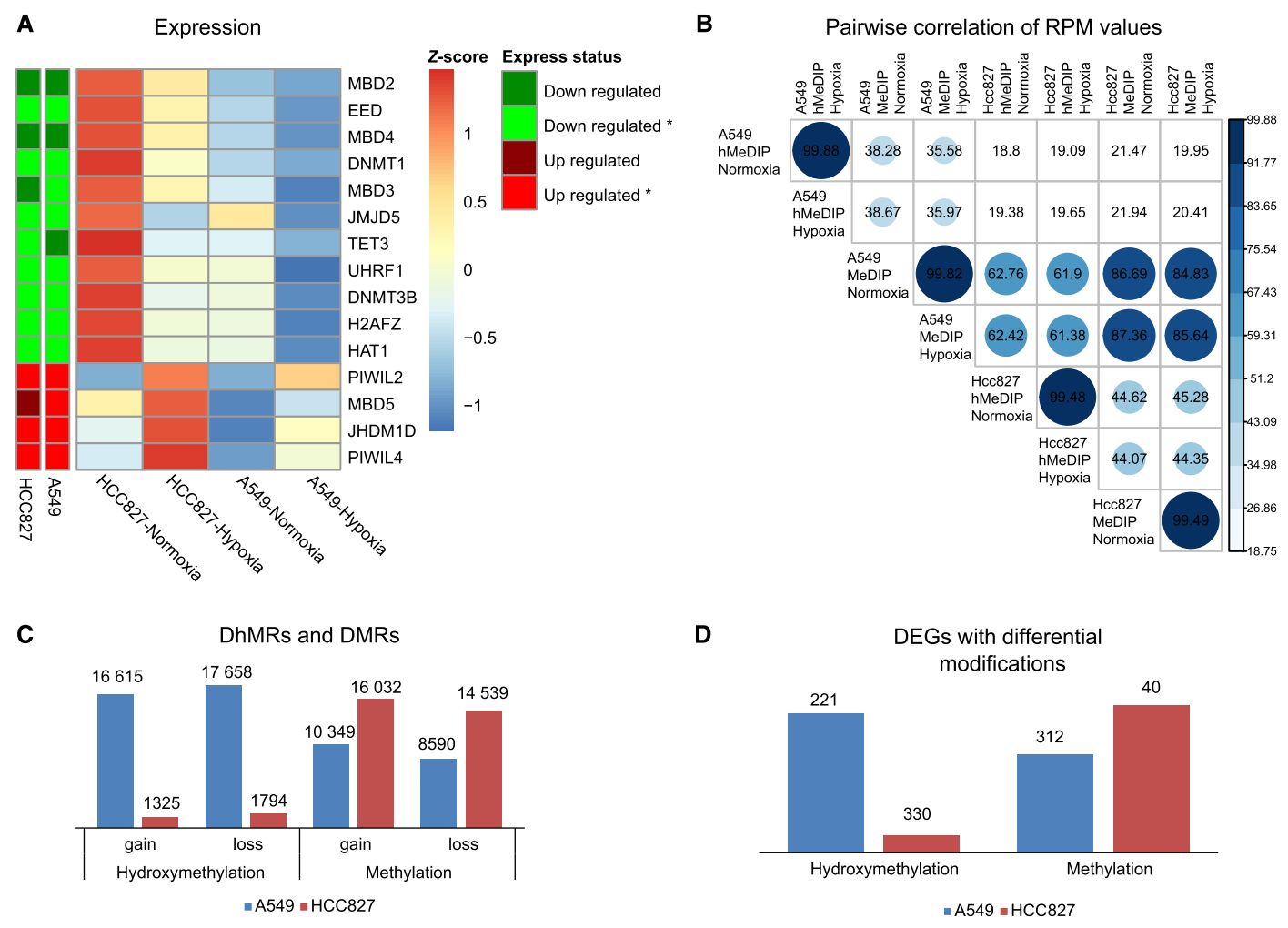
Figure 3:
(A)表观调控基因的差异表达情况
(B)DNA甲基化谱和羟甲基化谱的相关性分析(MeDIP和hMeDIP)
(C)DMR和DhMR鉴定
(D)DMR/DhMR与DEG分重叠分析
(4)HIF结合位点和靶基因表明多种细胞对缺氧的反应
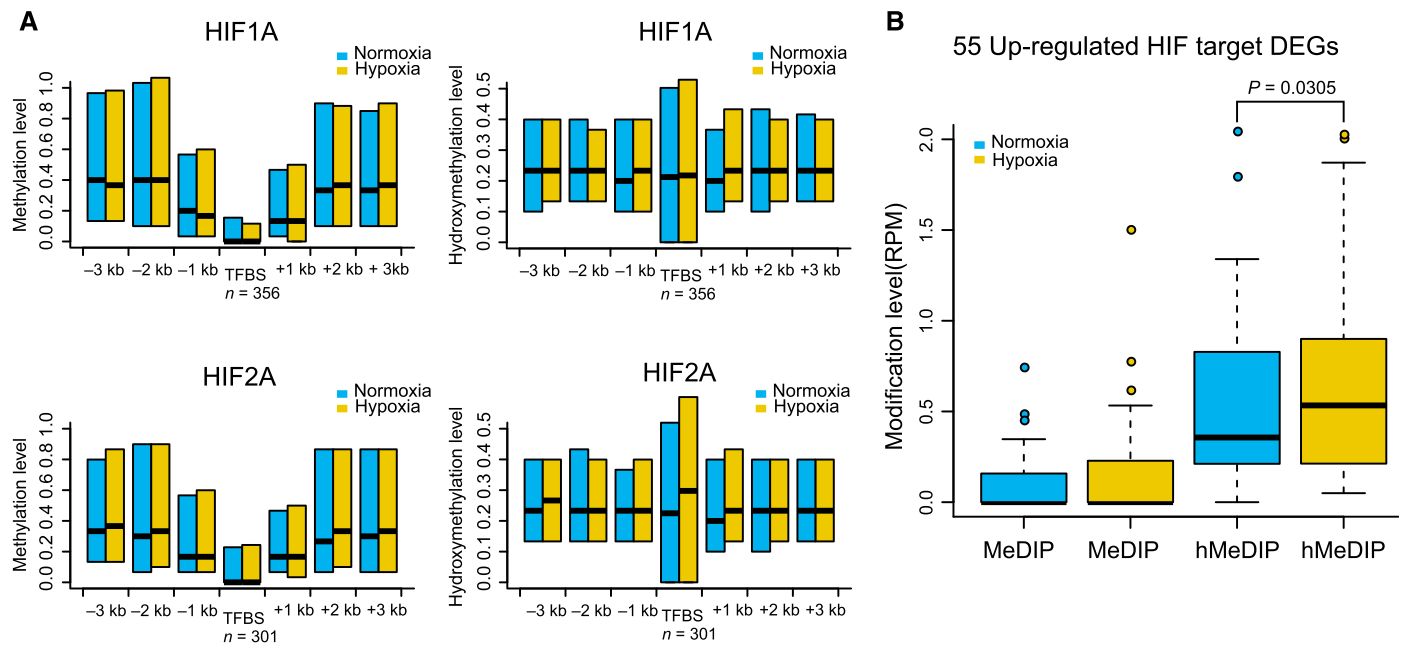
Figure 4:
(A)高氧诱导因子结合位点(HIF-TFBS)附近的基因甲基化和羟甲基化变化
(B)差异表达的HIF靶基因
(5)通过NSCLC患者基因表达数据集对17个基因panel进行训练和验证

Figure 5:
(A)人工神经网络模型预测癌症转移
(B)预测效果评估
(C-F)用其他公开数据集进行预测效果评估
04 结直肠癌 MeDIP-seq、hMeDIP-seq

标题:Integrated analyses of multi-omics reveal global patterns of methylation and hydroxymethylation and screen the tumor suppressive roles of HADHB in colorectal cancer
时间:2018
期刊:Clin Epigenetics
影响因子:IF 6.551
技术平台:MeDIP-seq、hMeDIP-seq、RNA-seq
背景方法:
DNA甲基化是一种重要的与基因表达有关的表观遗传修饰,5mC和5-hmC是维持表观遗传重编程的两个表观遗传标志,基因组甲基化可能致癌。本研究的目的是阐明DNA甲基化和羟甲基化在结直肠癌发生中的表观遗传机制。作者使用MeDIP-seq和hMeDIP-seq绘制了结直肠癌组织和相应正常组织的全基因组DNA甲基化和羟甲基化图谱和对应的转录组基因表达分析(RNA-seq)。鉴定差异甲基化区域(DMR)、差异羟甲基化区域(DhMR)和差异表达基因区域(DEG)。通过DMR、DhMR和DEG分析筛选表观遗传生物标志物,并使用功能分析进行确认。
研究结果:
作者研究了与正常结直肠组织不同的可以用作表观遗传生物标志物的全基因组羟甲基化模式。通过比较结直肠肿瘤组织和正常组织,共鉴定出59249个DMRs,187172个DhMRs和948个DEGs。在将DMR或DhMR的基因与DEGs交叉比对后,筛选出七个在肿瘤中受DNA甲基化异常调控的基因。此外发现HADHB基因的高甲基化与其在结直肠癌(CRC)转录下调相关。肿瘤功能分析表明,HADHB基因降低了癌细胞迁移和侵袭性。结果表明HADHB可能具有肿瘤抑制基因(TSG)的作用。
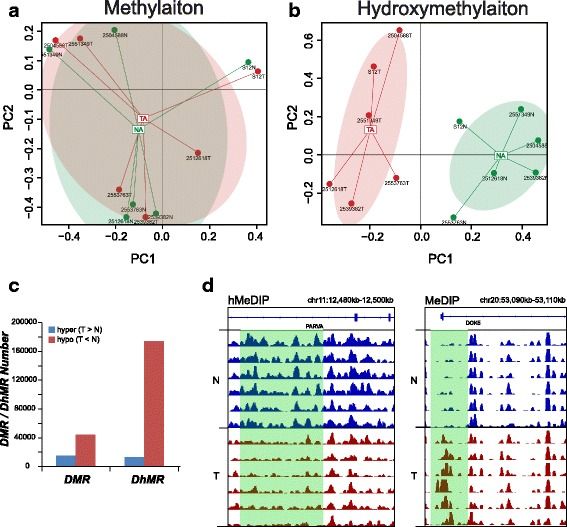
图:甲基化和羟甲基化的不同分布
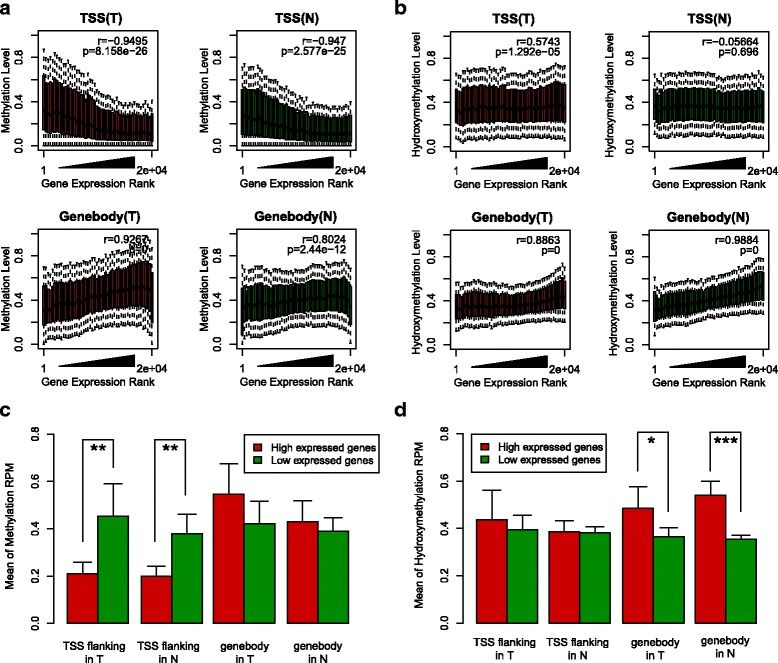
图:表观基因组修饰与基因表达的关系
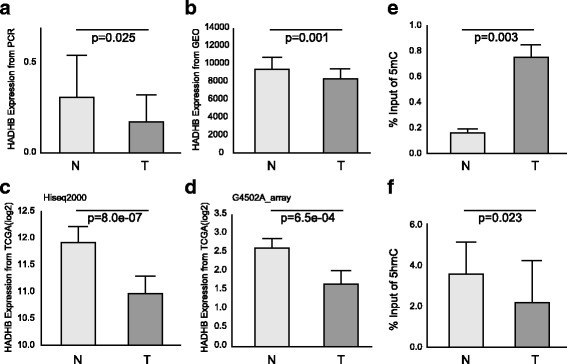
图:HADHB在其他样品和公共功能基因组学数据中的表达和表观遗传修饰验证

图:HADHB在CRC细胞系上的功能实验
结论:
本研究揭示了CRC的全基因组甲基化和羟甲基化模式。通过多组学分析筛选7个CRC相关基因。功能实验支持HADHB是一种潜在的肿瘤抑制基因,可以减少肿瘤细胞的侵袭性和迁移,表明HADHB基因沉默可能促进结直肠癌的发生和进展。
关于DNA羟甲基化测序
DNA羟甲基化是近年发现的一种新的DNA修饰并迅速成为研究热点。随着研究的深入,发现之前被认为是检测DNA甲基化标准的重亚硫酸盐测序并不能区分DNA甲基化(5mC)和DNA羟甲基化(5hmC)。易基因联合剑桥大学建立了化学氧化法结合重亚硫酸盐转化的测序技术(oxidative bisulfite sequencing, oxBS-Seq),该技术不仅可以精确检测DNA甲基化,排除DNA羟甲基化的影响,还可以双文库结合同时单碱基分辨率精确检测DNA羟甲基化。
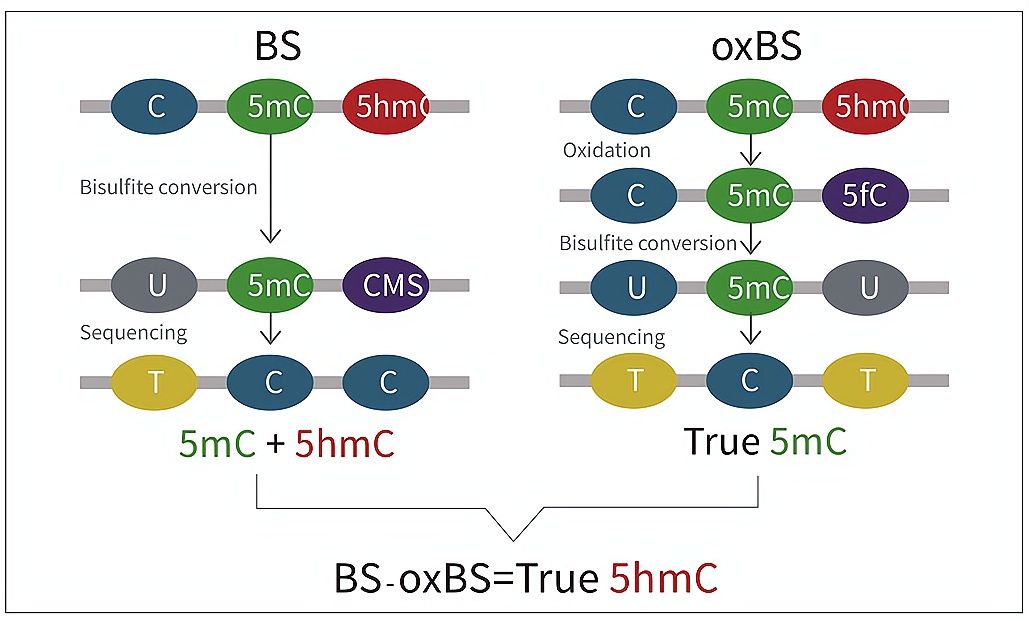
传统BS转化无法区分5mC和5hmC
传统的Bisulfite测序中,5hmC经过Bisulfite处理后变为CMS,CMS在测序中仍然被读作C碱基,因此不能区分5mC和5hmC。
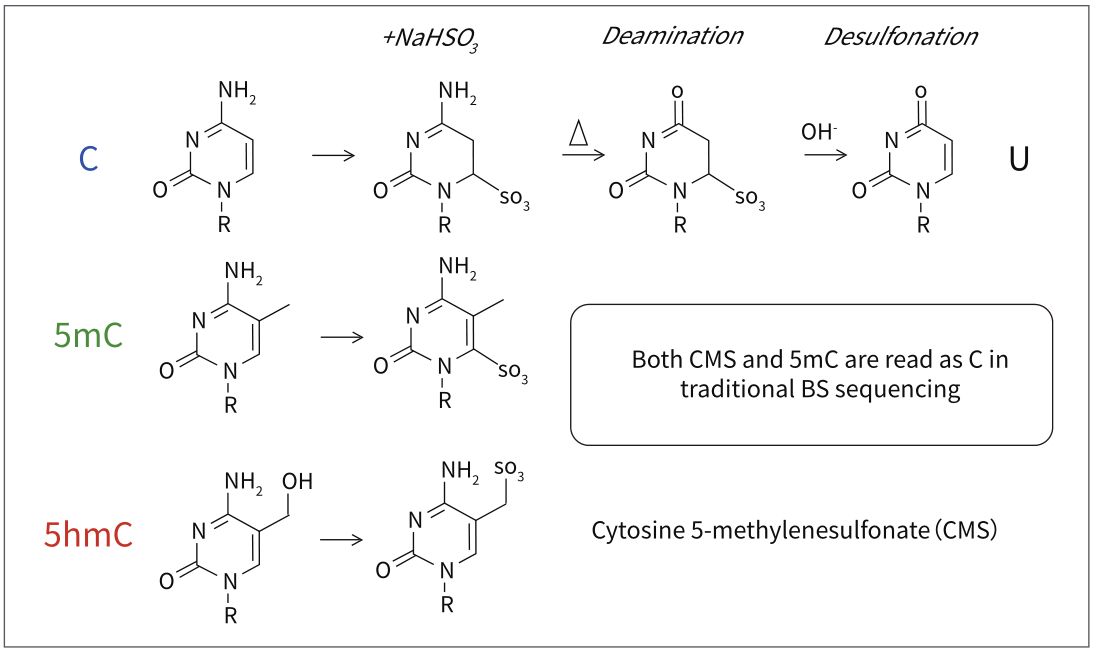
oxBS技术原理
oxBS-Seq将5hmC氧化5fC,后者可以被Bisulfite转为U,从而实现5mC的精准检测;同时,经过与常规Bisulfite结果比较可以实现对5hmC的准确检测。
技术优势
- DNA甲基化检测全新的“标准”;
- 全基因组单碱基检测DNA羟甲基化修饰;
- 多重标准验证高氧化效率和高Bisulfite转换率;
- 实验偏好性低,重复性高(R²>0.98);
- 可满足多种测序应用需求:全基因组氧化甲基化测序(oxWGBS),简化基因组氧化甲基化测序(oxRRBS),目标区域靶基因氧化甲基化测序(Target-oxBS)。
易基因另有免疫沉淀测序(hMeDIP-seq)技术,有羟甲基化测序或组学研究的老师可与我们联系哦~~
参考文献:
- https://doi.org/10.1002/ctm2.498
- https://doi.org/10.3389/fgene.2021.592135
- https://doi.org/10.1002/1878-0261.12491
4、https://doi.org/10.1186/s13148-018-0458-3
相关阅读:


