- 1大模型应用与边缘端推理_大模型在边缘侧的应用
- 2mysql 预编译的好处_Mysql 预处理 PREPARE以及预处理的好处
- 3mysql 锁机制与原理详解_mysql锁实现原理
- 4Qt介绍以及qt_creater的安装和C++项目工程创建_qt如何创建c++项目
- 5dev c++代码提示_git本地还分dev和master?
- 6TF-IDF算法应用介绍_tf-idf算法和应用场景
- 7华为Atlas200dk使用第三步------只用一根网线登录华为开发板_华为atlas怎么使用
- 8极域电子教室6.0全屏变窗口_MAC OS进阶——这10个技巧让你秒变MAC达人
- 9多旋翼无人机小知识_多悬翼m1m2m3m4
- 10AutoGPT 源码搭建与原理分析(四)_gpt源码
ViM-UNet:用于生物医学细分的 Vision Mamba
赞
踩
ViM-UNet: Vision Mamba for Biomedical Segmentation
摘要
卷积神经网络(CNNs),尤其是UNet,是生物医学分割的默认架构。已经提出了基于Transformer的方法,如UNETR,以取代它们,得益于全局视野,但受到更大的运行时间和更高的参数数量的影响。
最近的Vision Mamba架构为Transformer提供了一个有吸引力的替代方案,同样提供了全局视野,但效率更高。
在这里,作者介绍了基于它的ViM-UNet,一种新颖的分割架构,并将其与UNet和UNETR在两个具有挑战性的显微实例分割任务上进行比较。作者发现,它在不同任务上表现得与UNet相似或更好,并且在效率更高的情况下优于UNETR。
代码地址:
https://github.com/constantinpape/torch-em/blob/main/vimunet.md
Introduction
分割是生物医学图像分析中的一个重要任务,应用范围从放射学到显微镜学。大多数现代分割方法都基于卷积神经网络(CNN),其中UNet(Ronneberger等人,2015年)最受欢迎。在文本和视觉领域(ViT,(Dosovitskiy等人,2021年))的成功之后, Transformer 架构也被提出来用于生物医学分割;尤其是UNETR(Hatamizadeh等人,2021年)和SwinUNETR(Hatamizadeh等人,2022年)。它们具有全局视野,对于需要大上下文的任务来说有望获得更好的质量。然而,它们的运行时间和参数数量也更大。最近,Mamba架构(Gu和Dao,2023年)被提出,该架构扩展了状态空间模型(SSM)(Gu等人,2022年),旨在克服这些计算效率低的问题,同时保持全局视野。它已经被Vision Mamba(ViM)(Zhu等人,2024年)适应用于计算机视觉。
在这里,作者介绍了基于 ViM的 ViM-UNet,用于生物医学分割,并将其与 UNet 和 UNETR进行了比较,用于显微镜实例分割,这是生物学的一个重要分析任务。这项任务的大多数方法,例如CellPose(Stringer等人,2021年),StarDist(Schmidt等人,2018年),都是基于UNet架构,而最近的方法也采用了 Transformer ,例如(Archit等人,2023年),并且当前的基准测试(Ma等人,2024年)显示了 Transformer 有利的成果。作者使用了两个具有不同特点的数据集,见图1,发现ViM-UNet的性能与UNet相当或更好(取决于任务),而UNETR表现不佳。作者对结果进行了外部方法的验证,nnUNet(Isensee等人,2021年),这是一个经过良好测试的UNet框架,以及U-Mamba(Ma等人,2024年),它也基于Mamba,但缺少ViM的视觉特定优化。作者的结果显示了ViM在生物医学图像分析中的潜力。作者认为它特别有希望用于依赖于大上下文的任务,例如3D分割或细胞追踪。
Method and Experiments
作者比较了三种不同的架构:
UNet、UNETR和作者贡献的ViM-UNet,这些架构都在torch-em(Pape)中实现。UNet有4个层级,初始特征为64,每提升一个层级特征数量翻倍。对于UNETR,作者使用了Segment Anything(Kirillov等人,2023年)的ViT和与UNet相同的解码器,每个层级由两个卷积层和一个转置卷积层组成;输入来自前一个解码器层和ViT的输出。
作者选择了这种简单的实现方式,而不是像UNet和原始UNETR中的跳跃连接,后者将编码器和解码器中的相应层级连接起来。
作者发现这种设计没有负面影响。对于ViM-UNet,作者使用带有双向SSM层的ViM编码器。与ViT类似,这个模型在图像块上操作;作者使用的图像块大小为16。解码器设计与UNETR相同。对于UNETR和ViM-UNet,作者比较了不同大小的编码器;ViT有Base、Large、Huge三种尺寸,而ViM有Tiny、Small两种尺寸。
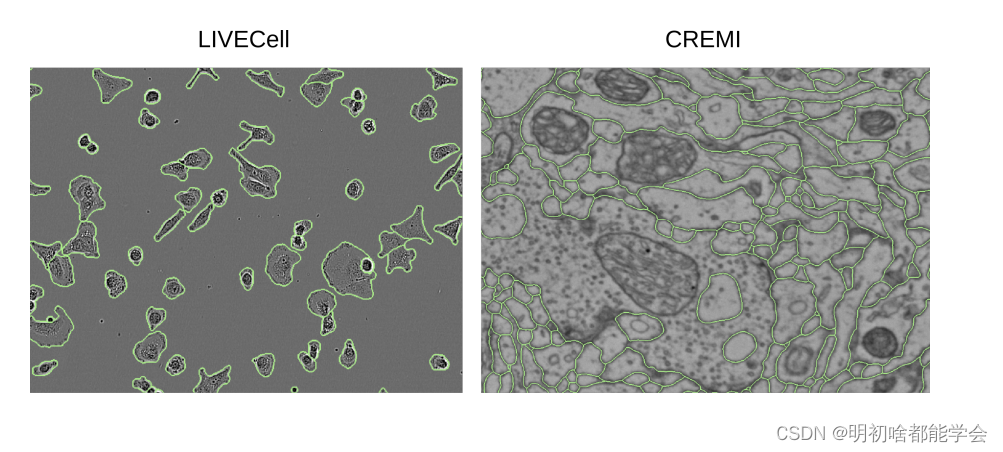
图1:使用ViM-UNetSmall进行分割的示例图像。
作者对两个数据集进行了比较:相位对比显微镜下的细胞分割(LIVECell (Edlund et al., 2021))以及体积电子显微镜下的神经纤维分割(CREMI (Funke et al., 2016))。LIVECell 包含了形态各异的微小细胞,而CREMI 包含了大小不一的神经纤维,详见图1。作者将CREMI的分割限制为2D。对于LIVECell,作者使用了给定的训练、验证和测试划分;对于CREMI,作者使用每个 Voxel 的前75个切片进行训练,接下来的25个进行验证,最后的25个进行测试。在LIVECell实例分割中,作者预测(i)前景和边界概率,这之后通过水波算法进行后处理,以及(ii)前景概率以及细胞中心和边界的距离,同样也用水波算法进行后处理。作者选择(i)与其他实现(见下文)进行比较,其中作者无法实现距离预测,而(ii)因为这种方法更适合这里。对于CREMI作者使用(i)。边界预测是这个任务的常规步骤,通常接下来会进行图聚集(Beier et al., 2017),这在2D中是不需要的。网络使用Adam进行训练,共10万次迭代,初始学习率为,并在平台期进行减少。作者与带有边界分割的nn-UNet (Isensee et al., 2021) 和 U-Mamba (Ma et al., 2024) 进行比较。这两种方法都通过超参数搜索进行配置,作者使用默认设置。作者使用平均分割准确度(Everingham et al., 2010)进行评估。
结果与讨论
图2展示了结果。对于LIVECell数据集,采用距离分割的UNet表现最佳,紧随其后的是ViM-UNet。UNETR的表现显著较差。对于边界分割,UNet明显表现最好,其次是ViM-UNet和外部方法。UNETR再次表现不佳。对于CREMI数据集,ViM-UNet表现最佳,其次是外部方法和UNet,UNETR的结果较弱。作者假设全局视野对于小结构(如LIVECell)并没有带来任何优势,但ViM-UNet可以利用它来处理大结构(如CREMI)。UNETR表现不佳,这很可能是由于参数数量较多(见表1)且缺乏预训练;请注意,通过预训练可以实现更好的性能(Horst等人,2023年)。与外部方法的比较验证了作者的实现并没有表现不佳,但由于训练和推理的差异,无法进行完全客观的比较。作者还研究了推理时间和训练所需的内存,见表1。UNet是最有效的架构,其次是ViM-UNet和UNETR。
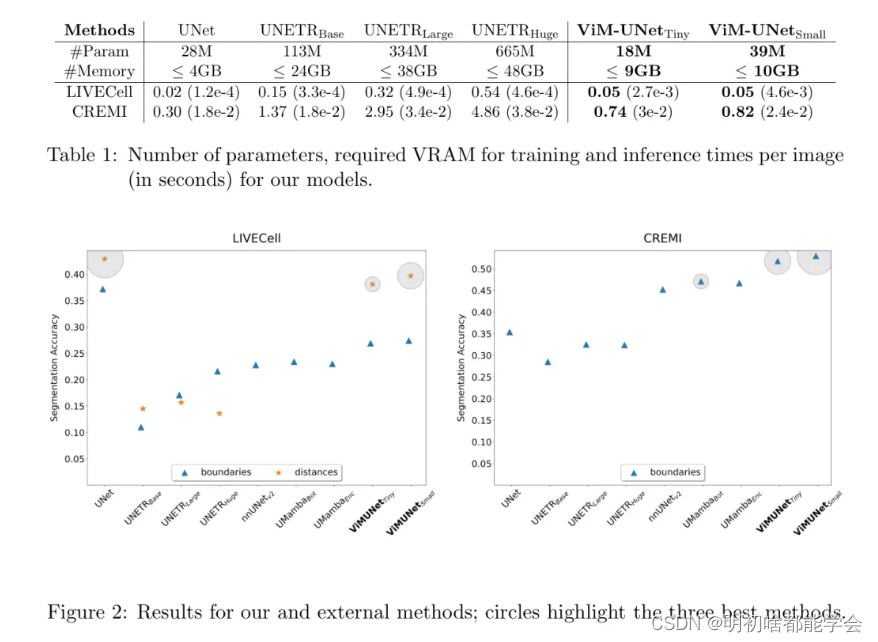
表1:作者模型的参数数量、训练所需的VRAM以及每张图像的推理时间(以秒为单位)。
图2:作者的方法和外部方法的对比结果;圆圈突出了三种最佳方法。
总的来说,ViM-UNet在生物医学图像分析方面很有前景。作者认为,它可能取代基于 Transformer 的方法,在需要大上下文的领域中应用,因为它同样具有全局视野,但效率更高。其较低的参数数量使得可以在较小的数据集上进行应用,且无需大量预训练。作者计划将其扩展到3D分割和跟踪,在这些领域大上下文通常至关重要。


