- 1C语言自定义数据类型(C语言十)_c语言类型是在哪里定义的
- 2ECharts 点击非图表区域的点击事件不触发问题_chart.getzr()不起作用
- 3Springboot+thymeleaf动态修改权限菜单_thymeleaf动态导航栏
- 4Python_文本分析入门_SnowNLP(1)_snownlp函数
- 5Huggingface中Transformer模型使用_huggingface transformer库
- 6FPGA_ZYNQ_XADC_fpga自带adc是多少位的
- 7adb查看设备信息_adb查看设备制造商
- 8Ubuntu配置DNS服务器--bind_ubuntu bind
- 9【华为OD机试】芯片资源限制(贪心算法—Java&Python&C++&JS实现)
- 10xtrabackup全量增量备份+全量增量恢复+binlog增量恢复
R语言进行孟德尔随机化+meta分析(2)----基于R和stata_孟德尔随机化 meta分析
赞
踩
目前不少文章用到了孟德尔随机化+meta分析,在上一章咱们简单介绍了一下meta分析的基础知识。咱们今天来介绍一篇11分文章,由文章看看孟德尔随机化+meta分析如何进行,文章的题目是:Appraising the causal role of smoking in multiple diseases: A systematic review and meta-analysis of Mendelian randomization studies(评估吸烟在多种疾病中的因果作用:孟德尔随机研究的系统回顾和荟萃分析)

吸烟其实没什么创意,唯一的新意就是加入了孟德尔随机化和meta分析。我们可以看到文章的类型就是meta分析,说明孟德尔随机化+meta分析的本质就是个meta分析。作者先是介绍说吸烟与多种疾病之间的因果关系仍然不明确,目的是通过总结孟德尔随机化 (MR) 研究的证据来评估吸烟在多种疾病中的因果作用。
咱们看下它的方法学是怎么做的:

完全是meta分析的套路,分两步提取数据,
第一步:是搜索各大数据库“孟德尔随机化”与“吸烟”的相关文章,把文章中吸烟和疾病的关系的数据提取出来。纳入标准:原始全文文章,介绍了吸烟或终身吸烟的遗传易感性与一种或多种循环、消化、神经和肌肉骨骼系统疾病、内分泌、代谢和眼部疾病或肿瘤风险的关联结果。一共纳入了385篇文章。排除标准:基于相同或重叠研究样本的重复出版物,以及仅使用单一或少数(<10)尼古丁依赖或吸烟行为或数量的工具变量的研究。作者这里提取了年份,样本量,关系的比值比OR。经过排除后适合分析的有29篇。
第二步:有一部分数据就是芬兰基因研究(FinnGen)作者通过检索没有检索到资料,他就自己来做,他使用了 R6版本中的数据进行孟德尔随机化分析,其中包括 260 405 名芬兰人,但剔除了性别不明确、非芬兰血统、基因型缺失率超过 5%、或杂合度过高(±4 个标准差)的数据。此外,作者还利用 GWAS meta 分析中公开的汇总统计数据,对骨关节炎、痛风和原发性开角型青光眼进行了从头开始的 MR 分析。第二部分提取到的数据应该是27篇,因为最后供56篇文章。
下面是他的流程图:
流程图:
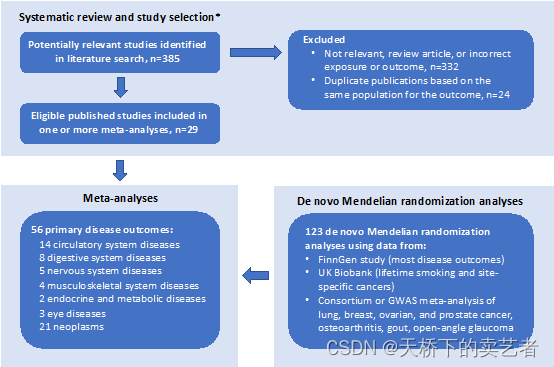
通过流程图咱们可以知道最后作者得到14篇循环疾病的文章,消化疾病8篇,神经系统疾病5篇,肌肉骨骼系统4篇,2篇内分泌,3篇眼科疾病,21篇关于肿瘤的文章。整个过程处理起来还是挺花时间的,毕竟要一篇篇的读,提取数据。
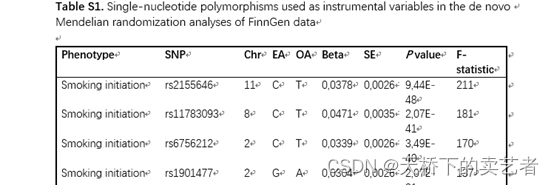
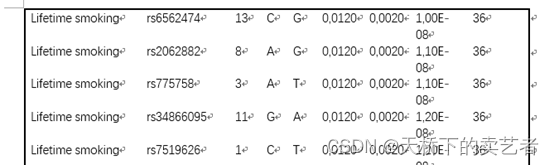
接下来咱们看看作者提供的数据,附表1是作者自己做的孟德尔随机化的结果,它的结果有两个,一个是刚开始吸烟,还有一个是终生吸烟。作者也是根据这两个结果进行meta分析的。
接下来就是作者的两个主表了,表2是开始吸烟的人的疾病分析,表3是终生吸烟的人的疾病分析,作者就是根据这两个表来做meta分析的,下面我把数据提取出来跑一下。
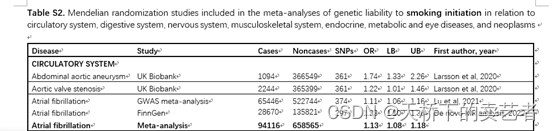
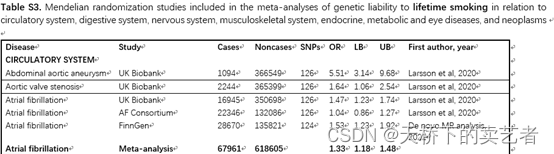
下面我把数据提取出来跑一下,数据量挺大的我就提取刚开始吸烟的患者循环疾病这部分举个例子,其他疾病的都是一样的。这里我们要注意一下,循环系统是有很多疾病的,就拿心房颤动这个疾病来说,作者的数据很多很大,他是把GWAS meta-analysis、FinnGen这2个数据库的结果进行相加,再来做meta分析,如果你的数据没有这么大,你把每个数据库先分别做,然后再汇总也是可以的。
bc<-read.csv("E:/r/test/smokemeta.csv",sep=',',header=TRUE)
names(bc)
- 1
- 2
数据提取出来后下图这个样子,这个数据可以按作者的方法进行提取,如果你想偷懒一点,使用我提取好的数据,公众号回复:代码,可以得到。
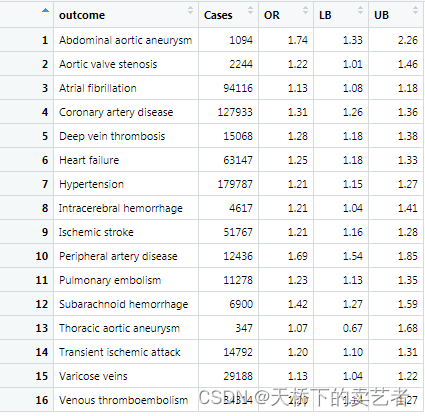
既往咱们已经介绍了《R语言forestploter包优雅的绘制孟德尔随机化研究森林图》,需要的可以自己看一下。今天咱们来介绍一下forestplot包绘制这个森林图,这个包相对简单一点,容易上手。
library(forestplot)
- 1
咱们先生成个可信区间
bc$`OR (95% CI)` <- sprintf("%.2f (%.2f to %.2f)", bc$OR, bc$LB, bc$UB)
- 1
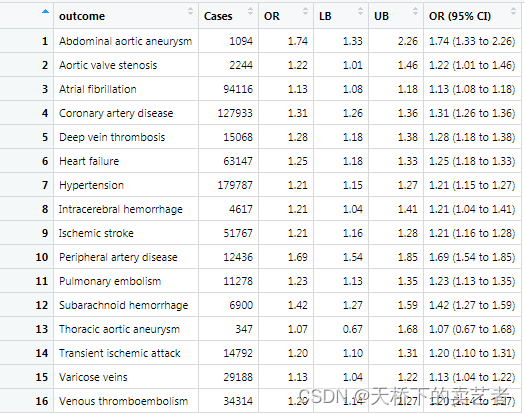
生成可信区间后我们需要生成一个绘图区间,选择你需要的变量就可以了,我这里选1,2,6
dt1<-as.matrix(bc[,c(1,2,6)])
- 1
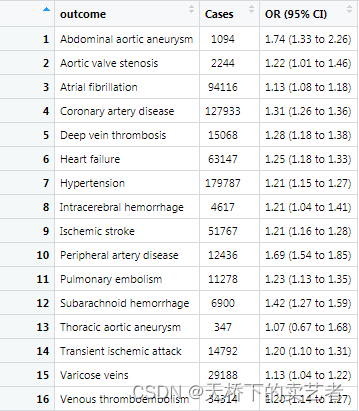
这里注意一下,dt这个数据是矩阵没有列名,我们还要生成一个列名
dt1 <- rbind(c("outcome","Cases","OR (95% CI)"),dt1)
- 1
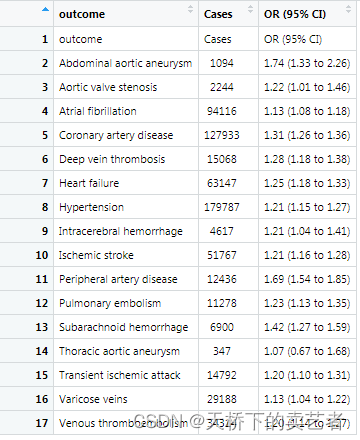
这样的数据就可以绘图了
forestplot(labeltext=dt1,graph.pos="right",
mean=c(NA,bc$OR),
lower=c(NA,bc$LB),
upper=c(NA,bc$UB),
graphwidth = unit(60,"mm"),#设置图片位置和宽度
boxsize =0.2,line.margin = unit(5,"mm"),#对散点和线条进行设置
lineheight = unit(5,"mm"),#设置图形行距
col=fpColors(box = "grey0",lines = "grey0",summary = "grey0"),
colgap = unit(1,"mm"),#图形列间距
zero = 1,#参照值
xticks = c(0,1,2))#X轴的定义标签
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
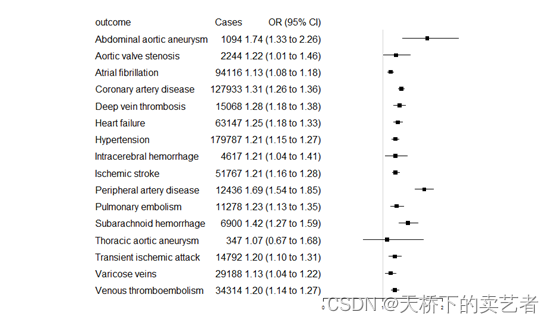
咱们可以看到和作者做的几乎一模一样
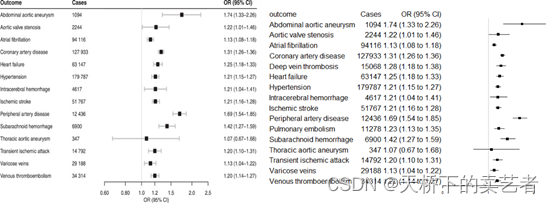
为什么我说几乎一模一样,因为还是有点不一样的,作者没有11278肺栓塞这个数据,但是它的原数据是有的,估计绘图时忘记加进去了
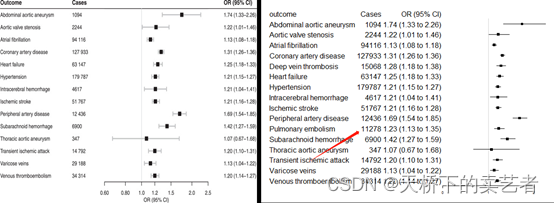
绘图出来了,还有一个东西没有解决。有些论文是有报meta分析的I(异质性)和P值的,这个怎么求出来呢?


这个两个值作者文章推荐使用stata来计算,stata做meta分析简单了许多, 使用metan函数就行
metan or lb ub
- 1
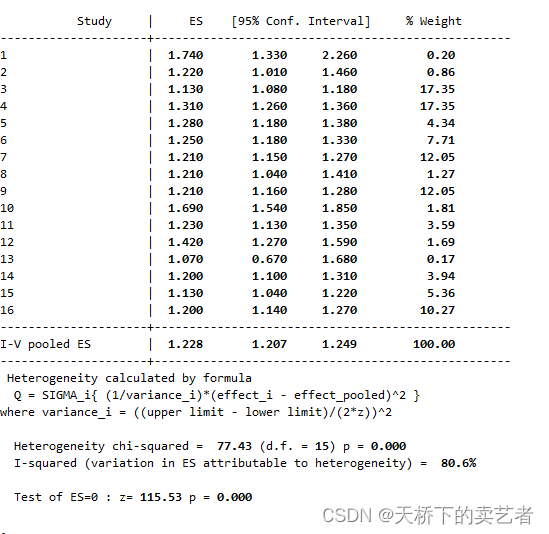
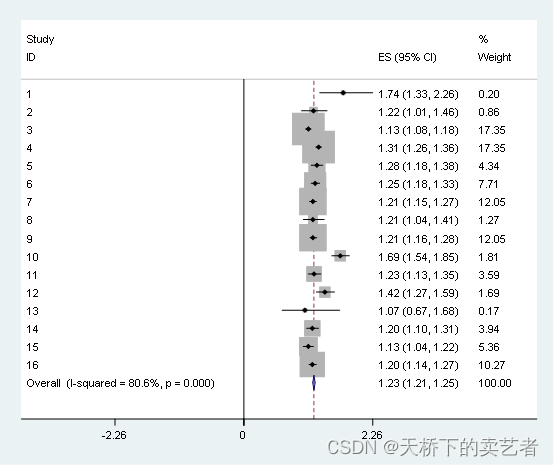
图片还可以修改,我这里不弄了。最终算得I为80.6%,P为0.00,可能和作者有点不同,因为他少加了一个研究。最后作者还做了敏感性分析,是通过孟德尔随机化来做的,不是所有的文章都做,下面这篇文章就没做敏感性分析。

我这里就不弄了,有兴趣看我既往的文章。


