- 1蓝牙技术|蓝牙远距离遥控,伦茨科技ST17H66可以帮到你_st17h66汽车无钥匙进入蓝牙
- 2RabbitMQ面试要点
- 3select * 与 count(*)数量不一致_技术分享 | MySQL:count(*)、count(字段) 实现上区别
- 4鸿蒙星河版笔记来袭!界面开发、组件的属性和方法_鸿蒙下列组件类型中,容器组件是
- 5算法与数据结构复习 第四章 字符串(详解)_设主串 t = abaabaabcabaabc,模式串 s = abaabc,采用 kmp 算法进行
- 6Vue项目遇到的问题解决及优化,如提示框和页码显示_vue3 的 防止catch错误返回出现在页面上
- 7PTA L1-002:打印沙漏(python)_l1-002打印沙漏python
- 8浅谈AI人体姿态识别技术的先进性及安防视频监控应用场景_ai 识别视频中人物的动作
- 9阿里云服务器端口授权对象设置自定ip访问_授权对象请输入ipv4 cidr地址
- 10【EI会议】2024年电网、电气与电子工程国际会议(PCEEE 2024)_2024 international conference on power grid, elect
具有 SAM2 分段的 NDVI 无人机_sam2教程
赞
踩
在我们之前的博客文章《OAK相机扩展NDVI功能检测植物健康情况》中,我们探讨了 NDVI 方法以及如何使用多光谱相机计算它。
今天,我们通过使用带有多光谱相机的无人机并使用 SAM2 模型进行场分割和健康比较,将 NDVI 感知提升到一个新的水平。
具有 SAM2 分段的 NDVI 无人机
1. 硬件
我们使用了 OAK-D-SR 的 PCBA 并更换了一个 CCM(紧凑型相机模块),因此一个传感器感知可见光波段 (380-750nm),而另一个传感器感知近红外波段 (>750nm)。

OAK-D-SR 连接到 RPi Zero 2W,RPi Zero 2W 连接到 OAK-D-SR 并每秒保存两帧。这两款设备都由移动电源供电,再加上DJI Mini 2 SE无人机(249克),所有设备的重量总共386克。

2. SAM2 分段
录制左/右帧后,使用 SAM2 Dmo 应用程序,上传彩色视频,并选择(一次仅 3 个)我们感兴趣的字段。选择字段并运行模型后,您可以在“网络”选项卡的“propagate_in_video”请求下检查分段结果。将这些结果保存在一个文件中,然后对其进行解码和可视化。
SAM2 结果是 RLE 编码的,因此需要对其进行解码才能获得掩码。您可以使用 pycocotools 来解码掩码。
- from pycocotools import mask as mask_utils
- mask = mask_utils.decode(annotation["segmentation"])
3. NDVI比较
在演示的底部,您可以看到字段之间的 NDVI 比较。因为 NDVI 是相对的(不是绝对的),所以我们只能用它来比较字段的健康状况。
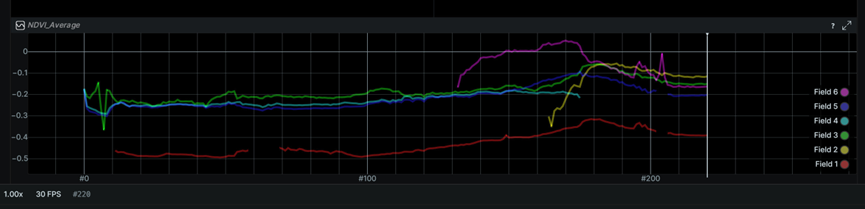
字段 6 具有最高的 NDVI 值,这从彩色 NDVI 图像中也很明显 - 它比其他字段更绿。

4. 可视化和代码
我们使用 Rerun 进行整个可视化,使用 OpenCV 进行图像处理和轮廓计算(以获得更好的可视化效果)。
以下是演示背后的主要逻辑。完整的代码可以在这里找到,完整的演示可以在这里找到(包括SAM结果和视频)。
- # Run & initialize ReRun viewer
- rr.init('NDVI /w SAM2', spawn=True)
-
- # Prepare rerun visualization
- annotationContext = [(0, "Background", (0, 0, 0, 0))]
- for i, color in enumerate(colors):
- annotationContext.append((i + 1, f"Field {i + 1}", color))
- rr.log(f"NDVI_Average/Field{i+1}",
- rr.SeriesLine(color=color, name=f"Field {i+1}")
- )
- rr.log("Color", rr.AnnotationContext(annotationContext), timeless=True)
-
- t = 0
- size = (800, 1280)
- for frame_idx in range(len(sam_data[0])):
- rr.set_time_sequence("step", t)
- t += 1
-
- frames = get_all_frames()
- ndvi = calc_ndvi(frames['color'], frames['ir'])
-
- segmentations = np.zeros(size)
- for i, data in enumerate(get_sam_output(frame_idx)):
- for result in data.get("results", []):
- field_num = result['object_id']+i*3 # 3 segmentations per file
- # Decode the RLE mask
- mask = np.array(maskUtils.decode(result["mask"]), dtype=np.uint8)
- # Set full_mask to num where mask
- segmentations[mask == 1] = field_num + 1 # as 0 is Background
-
- line_strips = get_contours(mask)
- rr.log(
- f"Color/Contours{field_num + 1}",
- rr.LineStrips2D(line_strips, colors=colors[field_num])
- )
- rr.log(
- f"NDVI/Color/Contours{field_num + 1}",
- rr.LineStrips2D(line_strips, colors=colors[field_num]
- )
-
- mean_ndvi = np.mean(ndvi[mask == 1])
- rr.log(f"NDVI_Average/Field{field_num + 1}", rr.Scalar(mean_ndvi))
-
- rr.log("Color/Image", rr.Image(frames['color'][..., ::-1]))
- rr.log("NDVI/Color", rr.Image(frames['ndvi_colorized'][..., ::-1]))
- rr.log("Color/Mask", rr.SegmentationImage(segmentations))

5. 可以进行的改进
需要注意的一点是,NDVI 是按图像视图计算的,而不是全局计算的。这就是为什么该字段的中位数 NDVI 会发生变化,而不是一个常数。为了改善这一点,我们可以使用整个视频(例如进行图像拼接)并计算整个区域的NDVI。
6. 结论
我们已经展示了如何使用带有多光谱相机的无人机来捕获 NDVI 图像,并使用 SAM2 模型进行现场分割和健康状况比较。这种方法可用于各种农业任务,例如监测作物健康、检测疾病等。
如果您有任何意见或建议,欢迎在评论中告诉我!


