- 1使用Axure RP9关于鼠标模式问题_axure切换鼠标样式
- 2spring boot中集成jedis操作redis_spring.redis.pool.testonborrow
- 3由于系统缓冲区空间不足或队列已满 mysql,zabbix 由于系统缓冲区空间不足或队列已满,不能执行套接字上的操作...
- 4基于 Spring boot + MyBatis 的在线音乐播放系统_springboot mp3文件怎么上传
- 5Nightingale 夜莺监控系统 - 部署篇(1)_夜莺v6.7.2部署
- 6关于使用core文件进行故障定位分析_core文件怎么分析
- 7Linux-Shell编程
- 8用云服务器windows环境下来搭建一个Minecraft服务器教程以及客户端使用教程java版_winserver2012安装mc服务器
- 9基于django的图片推荐系统-可视化大屏分析_django图片推荐系统
- 10如何使用ChatGPT构建一个Web应用程序?_chatgpt如何开发网站
【转】医学影像处理相关知识整理(一)_pixel spacing (0028,0030) 与 sid sod d的关系
赞
踩
转自:医学影像处理相关知识整理(一) - 知乎
Segmentation to RT structure
近日做医疗影像处理相关的内容,感慨于这方面资料不全、散碎,遂记录自己获得的一些资料以供日后查阅。
DICOM
DICOM 是医学图像和相关信息的国际标准,它定义了满足临床需要的可用于数据交换的医学图像格式,被广泛用于放射、成像的诊疗诊断设备。也就是说,在医院放射科使用的设备上读取的影像基本都是DICOM格式,包括CT、MRI、超声等。
DICOM格式的图像,每一张都带有除像素(体素)信息之外的大量信息。其信息组成主要有以下几部分:
- Patient:病人信息
- Study:诊疗信息,如检查时间、检查部位、ID等
- Series:序列号、图像坐标信息等。图像坐标信息主要关注:
- SliceThickness:层厚,即每张切片之间的间距
- ImagePositionPatient:该张切片坐标原点(图像左上角,二维图中向下为y轴,向右为x轴)在空间中的坐标(x,y,z)
- ImageOritentationPatient:六元组,当前切片的x、y轴与解剖学坐标系间的关系。
此处涉及矢状面、冠状面、横断面
简单的记忆方式是:矢状面即一支箭射中人所形成的面,正中二分人体。
冠状面可想象古代官帽,从头顶向地面的切面。
横断面即腰斩平面。
在下图中,红色切面为矢状面,紫色切面为冠状面,绿色切面为横断面。
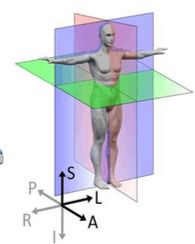
六元组所表示的,即是与这三个面的夹角关系,以余弦值呈现。
影像上,正方向为L、S、P。
用(x1,x2,x3,y1,y2,y3)来表示的话,x1、x2、x3分别表示x轴与L、P、S间的夹角余弦值,y1、y2、y3则表示y轴与L、P、S间的夹角余弦值。
使用这个六元组可以判断图像区域是否为矩形(两向量点乘为0),以及与解剖学坐标的关系。当这个六元组只含有0、1、-1时,代表切片一定与某解剖学平面平行。
更多内容可以参见此博客,讲解得很详细。
https://blog.csdn.net/zssureqh/article/details/61636150
- Image:图像信息。主要关注:
- PixelSpacing:二元组。用来表示当前二维图像坐标上,xy轴的单位长度,在实际坐标系中所占据的长度。
- Rows、Columns:行列信息。
- Bits Allocated、Bits Stored:每个像素分配的位数、存储位数
- Window Center、Window Width:窗位、窗宽
- Rescale Slope、Rescale Intercept :斜率、截距。主要用在图像可视化中。
除了上述四个部分 tag 信息外,就是 PixelData 信息。此处存储的既是该张切片的像素信息。
DICOM 文件可以使用 pydicom 库来进行读写,该库提供了非常全面的操作。
在 vscode 中搜索 dicom 有一个插件可以用来看 dicom 文件信息。
segmentation
segmentatiom 是对 DICOM 序列文件进行器官分割、靶区勾画后的结果,以 nifti 格式给出(也可以为其他格式,但存储形式基本都是裸数据,直接给出分割结果的数组)。其中的信息为三维数组形式,每个体素点存储该点对应图像的分割结果:
- 0:代表此点为background,或此次标注中不关注的区域
- 1~n:代表此点为对应的区域。
- 其中 1~n 所代表的器官、区域由分割程序定义
RT structure
RT structure 文件是导入到放疗系统中所需的 DICOM 文件,其中存储了每个结构区域的组成,以及相对应的原始 DICOM 序列标识和部分病人信息。其主要结构分为三个部分:
- 首先为头部信息,其中存储了诸如 patientname、patientid 一类的病人信息,以及 DICOM 信息。
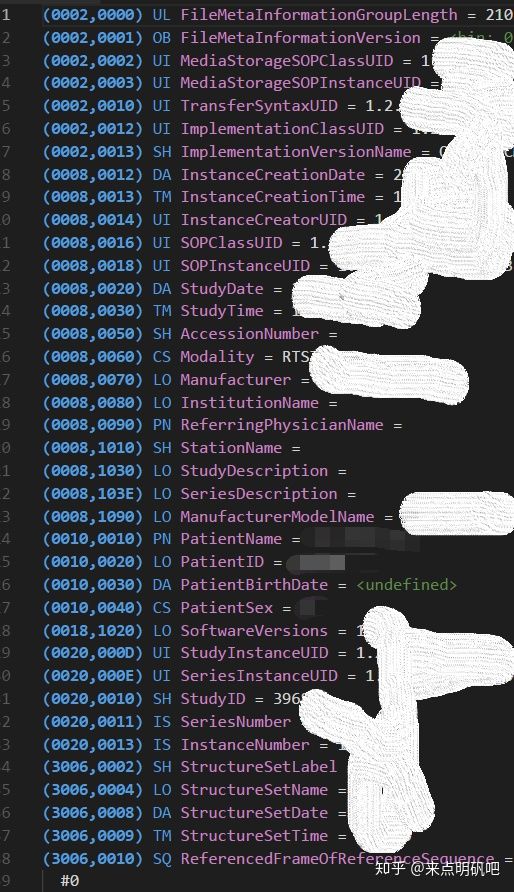
- 第二部分为 reference 信息,即该 RT structure 文件参考的 dicom 序列文件。
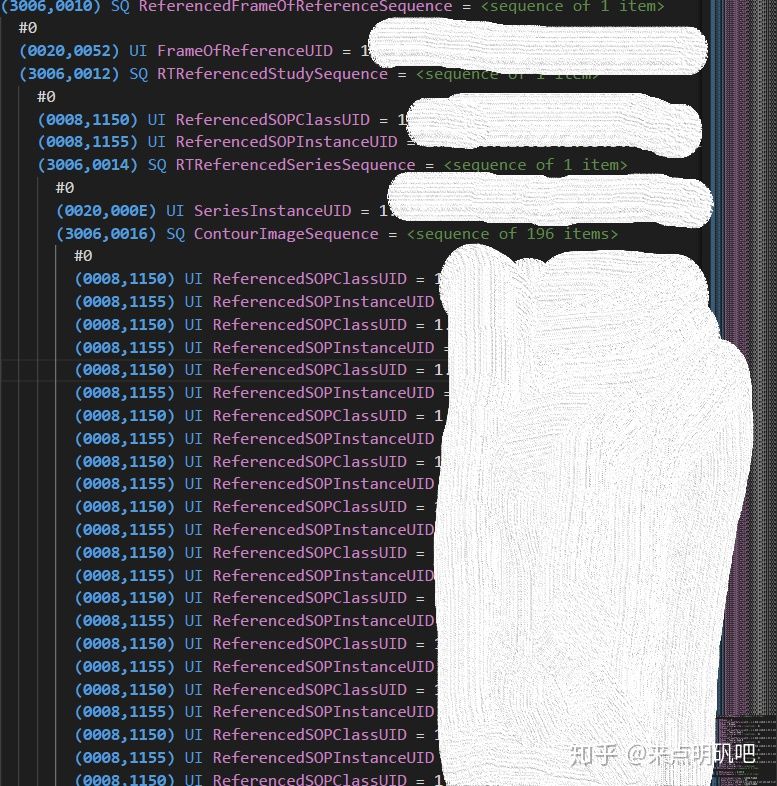
- 第三部分即是结构信息,存储各个区域的构成点、参考dicom文件等。
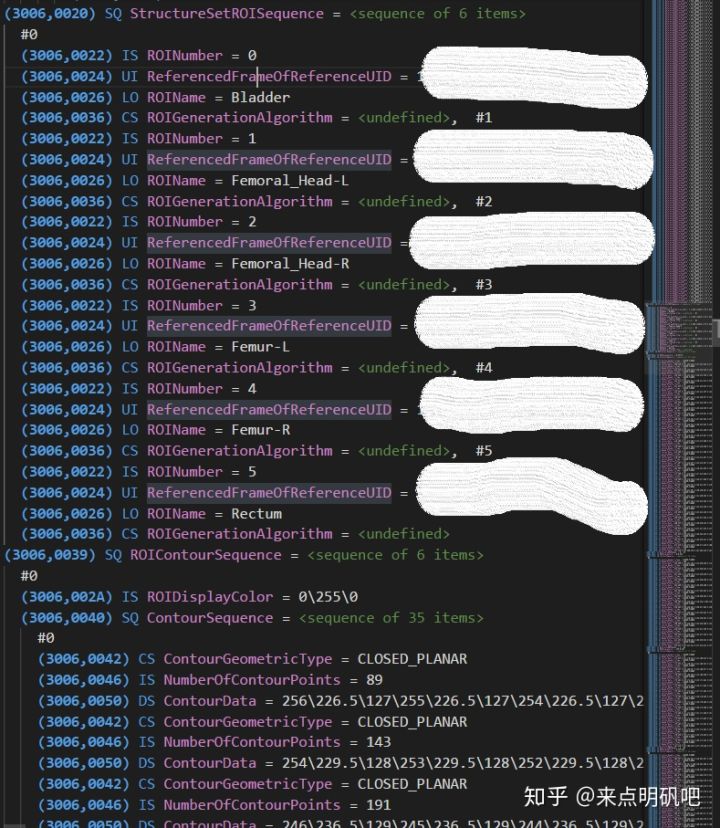
若要解析 RT structure 文件可以从以上三部分入手
segmentation to RT structure
近期所做的工作主要是将已经生成的分割、勾画信息结合原始的 DICOM 序列文件,生成 RT structure 文件(后简称 RTs ),以供医生后续使用。
主要思路为:nifti->mha->RTs
nifti->mha
mha 是一种 metadata 格式,其中存储了头部信息和像素信息。与 mha 格式类似的是 mhd 格式,区别在于 mhd 将头部信息存储于一个单独的 .raw 文件中,本身只存储数据信息。
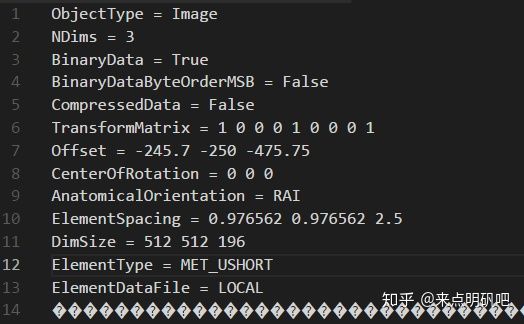
上图即为一个 mha 文件所存储的头部信息,可以看到,它存储了维度、原点、方向、像素间距等信息,这部分信息在后续构建 RTs 时是非常重要的。
那么,在初步了解 mha 文件的构成后,要将 nii 文件转换为 mha 文件就比较简单了。
思路为:
- 使用 SimpleITK 库读取 nii 文件中储存的体素信息。
- 使用 pydicom 库读取参考的 DICOM 序列文件信息。然后使用这里得到的信息用于构建 mha 头部信息。
- 最后再使用 SimpleITK 库写 mha 文件
一个简单的可执行示例代码如下:
- import SimpleITK as sitk
- import pydicom
- reader=sitk.ReadImage("labels.nii")
- referencect=pydicom.read_file("file_path.dcm")##输入参考的 dicom 文件,一个即可
-
- ConstPixelSpacing =(float(referencect.PixelSpacing[0]), float(referencect.PixelSpacing[1]), float(referencect.SliceThickness))
-
- Origin = referencect.ImagePositionPatient
-
- img=sitk.GetArrayFromImage(reader) #z y x
- img2=sitk.GetImageFromArray(img) #z y x
- img2.SetSpacing(ConstPixelSpacing)
- img2.SetOrigin(Origin)
- sitk.WriteImage(img2,"testmha.mha")
上例代码仅显示如何构建一个 mha 文件,在 nii 转化 mha 中还有很多细节需要主要:
- 在一个 DICOM 序列中,你最终参考的 DICOM 文件将决定你构建的 mha 文件头部信息中
offset项,该项将影响到构建 RTs 时切片的走向。例如若你选择的 DICOM 文件为切片中最顶部的一张,则你的整个结构由于层厚大于0,将向上生长,与真实结构相反。因此选择哪张切片作为参考,要结合层厚来确定。通常层厚大于0,因此选择最底部的切片作为参考。 - SimpleITK 库在读入 nii 文件生成数组时,将以 (z,y,x) 形式生成数组,如果同样以 SimpleITK 库写文件,可以不用注意,但若还有其他诸如可视化等需求,应注意坐标对应。
- 写 mha 文件时,可能出现 PixelSpacing、Origin 由于浮点数精度而不完全等于元数据的问题。如下图所示,此问题我暂未想到解决方案,但由于在后续生成 RTs 并无影响,因此暂时忽略此问题。

mha->RTs
将 mha 转换为 RTs 文件是一个比较棘手的问题,一开始我预备在了解 RTs 文件的结构后,手动构造 RTs 文件,但考虑到可靠性(有可能信息转换不完全或兼容性不够好,毕竟医学的东西谨慎一点更好),最终没有采用手动构造的方法,而是使用了 plastimatch 这个比较成熟的软件来进行。但不排除后续使用自己写的代码来完成这个步骤的可能性。
plastimatch 提供了 DICOM RT 导出功能,支持从 mha 文件导出 RTs,同时也支持 nii 导出 RTs。
在我成功从 mha 导出 RTs 后,试图简化步骤直接从 nii 到 RTs,但经过尝试后发现 nii 到 RTs 的结果与 mha 到 RTs 并不相同(很奇怪),且并不正确,同时,从 nii 到 RTs 所需的时间非常久,在五分钟以上,而使用 nii->mha->RTs 耗时不到 30s
这个结果令人费解,mha 文件的信息都是从 nii 中解析得到,导出 RTs 所使用的参考 dicom 序列也都相同,却导向了不同的结果。我最初猜测是由于 nii 中数组方向可能有问题,但在我修改方向重写 nii 后结果依然不对。
那么在安装 plastimatch 后,使用如下三条指令即可使用 mha 生成 RTs:
- plastimatch adjust --input testmha.mha --pw-linear "0,0,1,1,2,2,3,4,4,8,5,16" --output TEMP-1.mha
- plastimatch convert --input TEMP-1.mha --output-img TEMP-2.mha --output-type uint32
- plastimatch convert --input-ss-img TEMP-2.mha input-ss-list structure_ref.txt --referenced-ct renference_files/ --output-dicom DICOM-OUT
接下来解释一下上述三条指令:
- 第一条指令的目的是将 mha 文件存储的体素的信息做一个变换,遵循 0->0,1->1,2->2,3->4,4->8,依次类推。如果你的勾画结果并非是以位图方式存储(即第 0bit 为 1 表示该点为第 0 种结构,第 1bit 为 1 表示该点为第 1 种结构),那么你就需要这个操作将其转换为该格式,以方便后续处理。
- 第二条指令的目的是转换 mha 文件中的信息为 uint32 存储。
- 第三条指令生成 RTs 文件,其中
input-ss-list structure_ref.txt后续阐述。referenced-ct renference_files/指定要参考的 DICOM 序列所在的文件夹,最终将在DICOM-OUT目录下生成 RTs 文件。
structure_ref.txt
该文件遵循下述格式:
- 0|0 255 0|Bladder
- 1|0 0 255|Femoral_Head-L
- 2|255 0 255|Femoral_Head-R
- 3|0 255 255|Femur-L
- 4|255 255 0|Femur-R
- 5|122 122 122|Rectum
在未指定该文件时,生成的 RTs 文件中结构名是没有指定的,将以 unknown structure 的名字呈现,这对后续医生的操作是不利的,因此需要指定上述文件,以形成 value-color-structure_name 的对应关系。此处的 value,即 0、1、2...等值,是指对应 bit 位为 1。因此前述第一条指令的转换操作就是不可缺少的,将普通的值转换为对应的 bit 位为 1 的值,才能正确标识结构名字。
others
此处列出一些可能有用的库:
SimpleITK:支持较多格式的文件,提供了一些处理图像内容的操作。
pydicom:提供对 DICOM 文件读写、修改的操作。
itk、vtk:前者提供处理,后者提供显示,是医学影像处理中常用的库。
dicom2nifti:dicom 文件转 nii 文件等操作。
gdcm2vtk:提供 vtk 支持的格式文件转换 dicom 文件等操作。
此外还有 gdcm、dcmtk 等库尚未详看。
plastimatch 这个软件也提供了很丰富的功能,最重要的是在它的 mail list 中,开发者会回复使用相关问题。
后续可能会调研图像显示相关的资料并更新。
不过没搞明白一通操作下来是什么目的,既然已有勾画信息,再弄rts有什么其他用途
因为勾画信息不是医生做的,是用AI做的。AI做的勾画信息不是RTs结构的,医生没办法在现在的放疗系统上使用,所以要转一下。
mha可以转回nii吗?要怎么转?
从mha文件里面提取像素信息,然后保存为nii就可以了,nii本质上就是裸数据


