- 1人脸识别--SeetaFace2介绍
- 2硬核!2023版Android面试指南,涵盖Android所有核心技能_android面试项目亮点
- 3org.apache.flink.table.api.ValidationException: Expected LocalReferenceExpression. Got: timestamp_expected localreferenceexpression. got: duration
- 4字节跳动2024前端技术岗发布+最新内部面试题_字节前端定级
- 5Android Studio上传至GitHub_failed to create repository
- 6数据仓库与数据挖掘实验练习题
- 7机器学习与自然语言处理
- 8Python基础:lambda函数详解_lamda函数python
- 9CSS 元素垂直居中的 6种方法_ie的css垂直居中
- 10原来阿里P8的工资这么低啊。。。
如何在linux系统中用conda安装R环境及R包_在conda安装r 包install.packages("pmcmrplus")
赞
踩
一、miniconda3的安装不再赘述
二、安装R环境
1. 提前准备好conda的R单独环境
conda env list #查看已有环境
conda create -n R4.1.2
conda activate R4.1.2
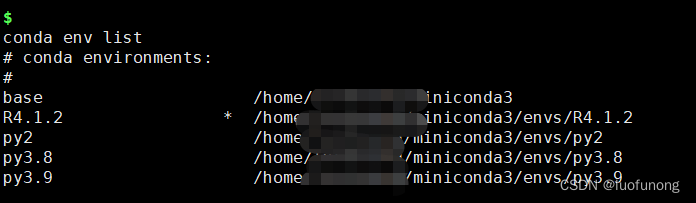
2. 进入环境后,多添加一些conda channel
- conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free
- conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forge
- conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/bioconda
- conda config --set show_channel_urls yes
- conda config --show
channel少的话,r-base种类少
conda search r-base
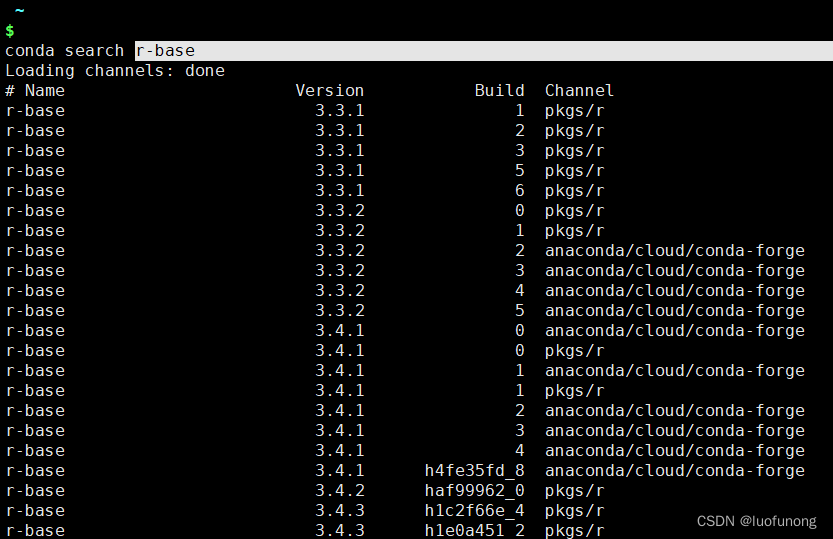
选择一款适合的版本
- conda install r-base=4.1.2
- which -a R
将自己刚才安装的R绝对路径添加到.bashrc中的环境变量中$PATH,也可以加alias。
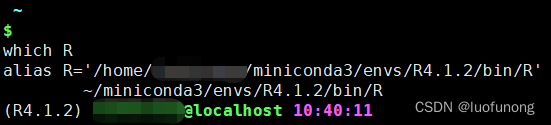
OK, 现在R的环境已经安装好了。
三、安装R包
1. 镜像设置
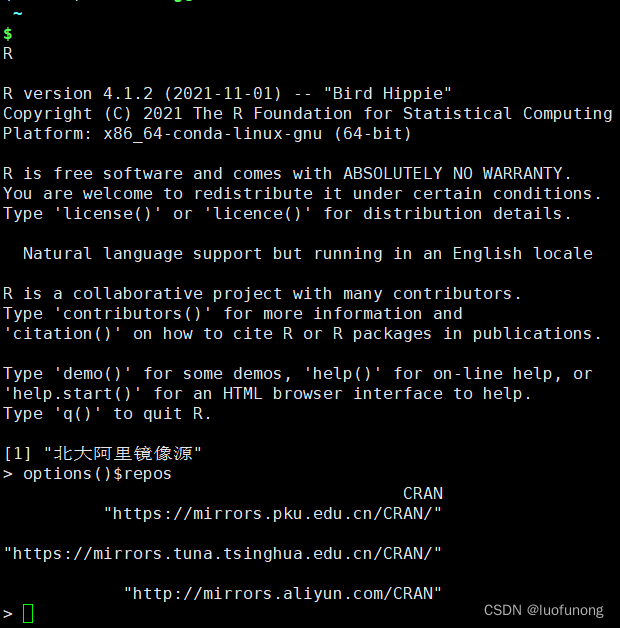
进入R
options()$repos #查看现有镜像
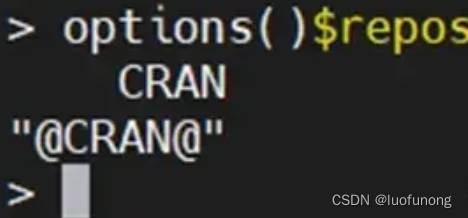
OK,没有任何镜像
file.edit(file.path("~",".Rprofile")) #打开一个文件
在文件中写入下面的内容 #多写几个镜像
options("repos"=c(CRAN="https://mirrors.pku.edu.cn/CRAN/","The Comprehensive R Archive Network","http://mirrors.aliyun.com/CRAN"))
print("已设置北大阿里云镜像")
写完后,退出,重新进入R
options()$repos #查看现有镜像
2. 安装BiocManager ##官网Bioconductor - Install
R4.1.2对应的BiocManager版本是3.14
在R中输入
if (!require("BiocManager",quietly = TRUE))
install.packages("BiocManager")
BiocManager::install(version ="3.14")
3. 安装R包
方法一:>install.packages("package_name")
方法二:
> library(BiocManager)
> BiocManager::install("ggplot2")
OK,此时你可以安装自己想要的R包了。
#附些常见的命令,查看已安装的R包“library()”。加载包“library(packagename)”。查看已加载的包“(.packages())”。取消已加载的包“detach("package: packagename")”。(参考知乎-阿辉的科研)


