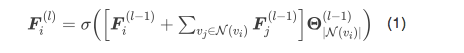- 1Java简单计算器界面及功能的实现_java实现计算器界面及功能
- 2电气领域相关数据集(目标检测,分类图像数据及负荷预测),输电线路图像数据_电网数据集
- 3sql中使用“where 1=1 and ....“ 到底影响效率吗,回答不会也永远不会_sqlserver用where 1=1影响查询速度吗
- 4二叉树的四种遍历方式
- 5Android正方教务系统课程表+查成绩+查考试安排_android 成绩查询
- 6Altium Designer画PCB详细教程_ad pcb
- 7npm i 长时间卡住问题_npm i 卡死
- 8arm交叉编译
- 9时间序列分析:使用Pandas探索能源数据集_德国能源数据时间序列分析
- 10Android Studio项目 通过implementation 添加依赖时下载的jar包放在哪里了_implementation 如何添加
赠书 | GNN 模型在生物化学和医疗健康中的典型应用
赞
踩

文中有数据派THU福利哦
计算生物化学和医疗健康的数据常常通过图来表示。
例如,分子和化合物可以自然地表示为以原子为节点、以键为边的图。蛋白质相互作用(Protein-ProteinInteractions,PPI)记录了两个或多个蛋白质之间的物理联系, 这种联系可以很自然地用图的形式表示。
此外,在制药行业中,药物相互作用(Drug-Drug Interactions,DDI)描述了在使用不同药物组合治疗复杂疾病时的不良结果,这种相互作用也可以用图来表示。
图神经网络模型具有强大的图表示学习能力,已被应用于许多生物化学和医疗健康应用中,包括药物开发与发现、药物相似性整合、复方药物副作用预测、药物推荐和疾病预测。
下面将讨论GNN 模型在生物化学和医疗健康中的一些典型应用。
图神经网络已经被用来推动药物开发和发现中的许多重要任务。
这些任务的实例包括:
1)分子表示学习,该任务可以用于辅助分子属性预测等下游任务,从而有助于将候选分子的搜索范围缩小到具有合适性质的分子上;
2)分子图生成,旨在生成具有某种期望性质的分子;
3)药物–靶标结合亲和力预测,即预测药物–靶标的相互作用强度,以便于新药开发和药物再利用;
4)蛋白质相互作用界面预测,其目的在于预测蛋白质相互作用界面,以便于理解分子相互作用界面,进而理解分子机制。
接下来介绍图神经网络在分子表示学习、药物–靶标结合亲和力预测以及蛋白质相互作用界面预测等方面的应用。
01 分子表示学习
预测新型分子的性质对于材料设计和药物发现具有重要意义。
深度学习方法已经被用于预测分子性质。
通常来说,分子可以是任意大小和形状的,所以前馈网络和卷积神经网络等深度学习方法不能直接应用于分子数据。
预测过程通常包括两个阶段:特征提取,提取分子指纹,即编码分子结构信息的向量表示;性质预测,将提取的分子指纹作为输入,利用深度学习方法预测。
在传统方法中,可以使用一些现成的指纹软件提取分子指纹,而这样缺乏来自下游任务的指导。因此,提取出来的表示对于下游任务来说可能并不是最佳的。
文献[1]提出了一种端到端的预测框架,它采用图神经网络以一种可微的方式学习分子指纹。具体而言,一个分子可以表示为一个图
,其中节点表示原子,边表示这些原子之间的键。因此,分子性质预测的任务可以看作图分类或图回归问题,这就需要学习图级表示。注意,在描述分子的背景下,这些表示称为分子指纹。应用于该任务的图神经网络模型由图滤波层和图池化层组成。具体而言,文献[1]采用了全局池化方法。
本文首先介绍其图滤波层,再介绍获取分子指纹的全局池化层。对于节点
,第 层中的图滤波操作可表述为:

02 蛋白质相互作用界面预测
如图1 所示,蛋白质是具有生化功能的氨基酸链。如图2 所示,氨基酸是一种有机化合物,它含有氨基(–NH2)、羧基(–COOH)官能团和每个氨基酸特有的侧链(R 基)。


图1 蛋白质由一串氨基酸组成

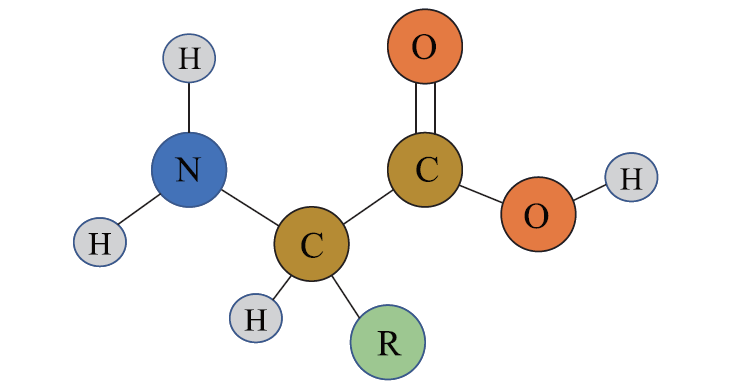
图2 氨基酸的一个说明性示例
蛋白质为了实现它们的功能,需要与其他蛋白质相互作用。预测这些相互作用发生的界面是一个具有挑战性的任务,同时这也在药物发现和设计中有着重要的应用。蛋白质相互作用界面由相互作用的蛋白质中相互作用的氨基酸残基和附近的氨基酸残基组成。
具体来讲,文献[3]考虑来自不同蛋白质的两个氨基酸残基,如果其中一个氨基酸残基中的任何一个非氢原子在另一个氨基酸残基中任何一个非氢原子的6Å 内,则认为它们是界面的一部分。因此,蛋白质相互作用界面预测问题可以建模为以来自不同蛋白质的一对氨基酸残基作为输入的二分类问题。
在文献[2]中,蛋白质被建模为图。在图中,蛋白质中的氨基酸残基被视为节点,这些节点之间的关系被定义为边,然后使用图神经网络模型学习节点表示,并利用这些表示进行分类。
接下来介绍如何将蛋白质表示为图,并介绍蛋白质相互作用界面预测的方法。



药物-靶标结合亲和力预测
开发一种新药通常既耗时又昂贵。
在药物开发的早期阶段,药物–靶标相互作用(Drug-Target Interactions,DTI)的识别对于缩小候选药物的搜索范围至关重要。它还可用于药物再利用,旨在识别现有或废弃药物的新靶标。
药物–靶标结合亲和力预测任务是推断给定的药物对与靶标之间的结合强度,可以将其视为一项回归任务。在药物–靶标亲和力预测任务中,经常涉及的靶标主要有4 种,即蛋白质、疾病、基因和副作用。本节以蛋白质为例说明如何在这项任务中使用图神经网络模型。

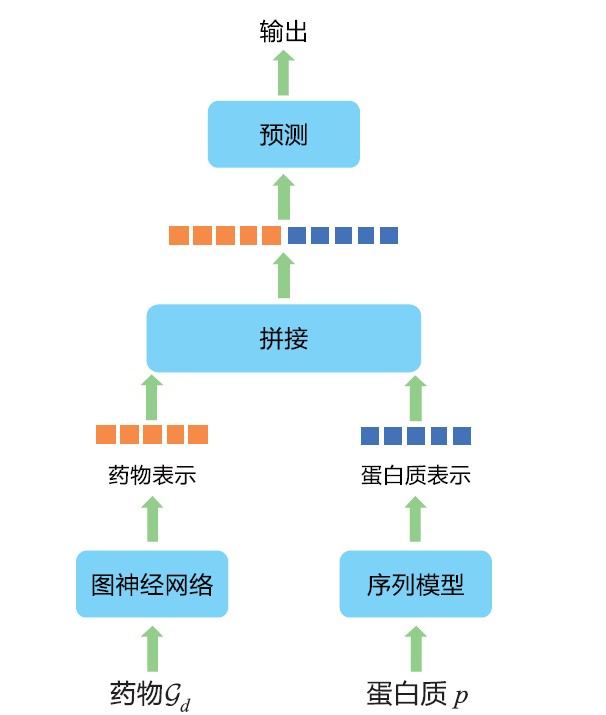
图3 药物–靶结合亲和力预测的一般框架
参考文献:
[1] DUVENAUD D K, MACLAURIN D, IPARRAGUIRRE J, et al. Convolutional networks on graphs for learning molecular fingerprints[C]. Advances in neural information processing systems, 2015: 2224–2232.
[2] FOUT A, BYRD J, SHARIAT B, et al. Protein interface prediction using graph convolutional networks[C]. Advances in Neural Information Processing Systems, 2017:6530–6539.
[3] AFSAR MINHAS F U A, GEISS B J, BEN-HUR A. Pairpred: Partner-specific prediction of interacting residues from sequence and structure[J]. Proteins: Structure,Function, and Bioinformatics, 2014, 82(7): 1142–1155.
[4] NGUYEN T, LE H, VENKATESH S. GraphDTA: prediction of drug–target binding affinity using graph convolutional networks[J]. BioRxiv, 2019.
以上内容节选自《图深度学习》一书!


▊《图深度学习》
马耀,汤继良 著
王怡琦,金卫 译
揭秘图深度学习的研究和学习路线图
全面覆盖图深度学习的基础理论、模型方法、实际应用及前沿进展
获俞士纶等十余位人工智能国际专家赞誉!
本书全面介绍了图深度学习的理论基础、模型方法及实际应用,既适合对数据挖掘、机器学习和社交网络分析感兴趣的本科生和研究生阅读,也适合企业开发者和项目经理阅读。对于没有计算机科学背景,但想要应用图神经网络来推进其所在学科发展的研究人员,本书同样是一本值得参考的读物。
 数据派THU独家福利!
数据派THU独家福利!
点点为数据派THU的粉丝们争取了3本赠书福利!欢迎小伙伴儿在下方留言区说出想要获得赠书的理由,我们将为点赞数最高(截止到2021年6月28日早8点)的3位读者免费送上此书~小编会联系你们哦!