热门标签
热门文章
- 1c 添加多语言resx,C# 多语言利器 – ResX Manager
- 2AI面试必备/深度学习100问1-50题答案解析
- 3idea上传代码到github的3种方式_idea上传项目到github
- 4【Java多线程】【线程状态和安全】_java多线程安全
- 5scrapy pipeline 管道 (图片,文件)_files_result_field
- 6总是做不好需求管理?这6款必备工具送给你_pingcode和禅道哪个好
- 7java针对http远程调用工具类
- 8去掉鼠标系列之一: 语雀快捷键使用指南
- 9iOS runtime 详解和使用场景(最详细的使用教程)
- 10Spark Kubernetes 的源码分析系列 - submit
当前位置: article > 正文
使用PLINK做GWAS(2)_plink进行gwas
作者:你好赵伟 | 2024-04-21 14:29:39
赞
踩
plink进行gwas
利用PLINK对单基因控制的性状进行定位,之前一篇文章粗滤的记录了一下使用PLINK的命令,本次博客主要记录一下如何根据SNP芯片的数据制作PLINK的格式,以及分析的步骤,最后数据的可视化。
首先,有的公司给的新片的数据就是一个excel表格,列表示的是样品,行表示的是基因型,基因型用ATCG构成,当然有的基因型是用数字0或者1表示的。下面我拿到的一个数据是15个样品构成的基因型,有13个样品是突变体,2个样品是野生型。 格式如下:
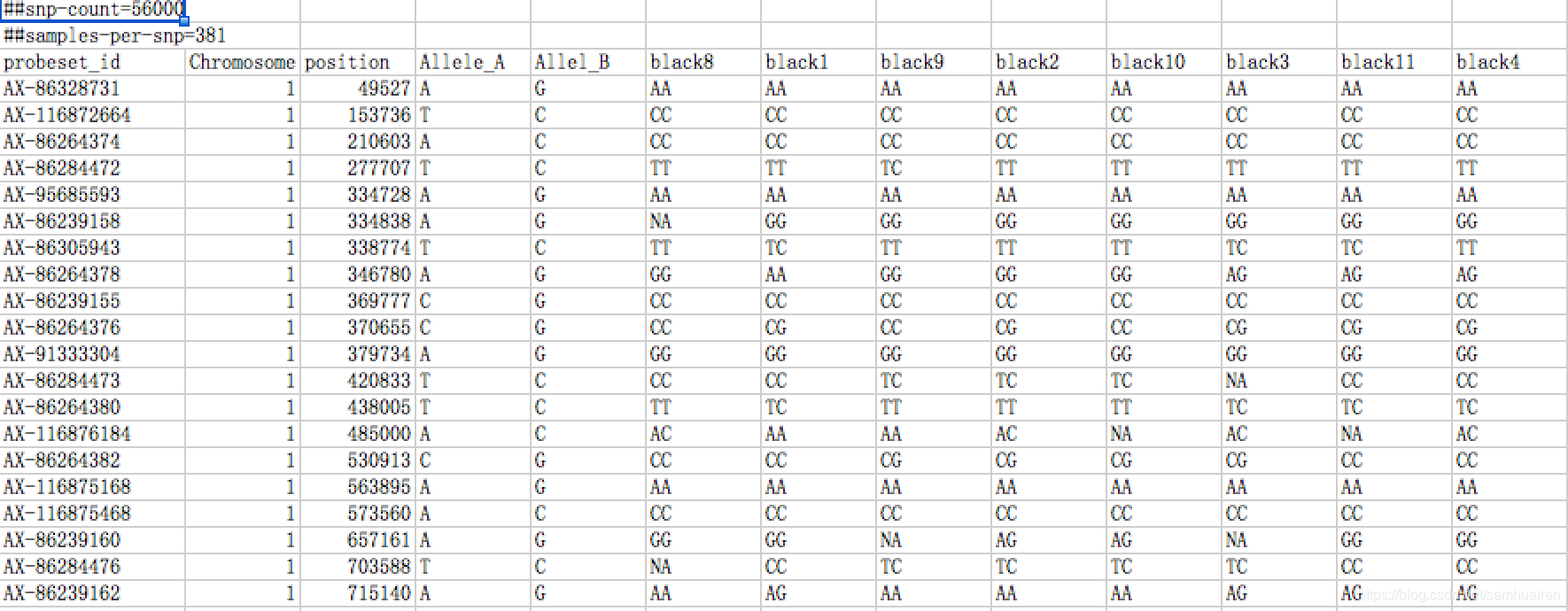
第二步:将此基因型存成csv文件,使用R进行读取,在R中制作plink所需要的两个文件,plink.map 和plink.ped. 其中map文件包含了四列,分别是染色体,SNP名词,遗传位置,SNP物理位置。没有遗传距离的可以全部写成0. ped 文件的包涵了family ID, individual ID, Paternal ID,Maternal ID,Sex (1=male; 2=female; other=unknown),phenotype, 以及每个材料的基因型,每个基因型需要将2个allele 分开写。 对于植物,family ID,Paternal ID,Maternal ID,Sex (1=male; 2=female;
声明:本文内容由网友自发贡献,不代表【wpsshop博客】立场,版权归原作者所有,本站不承担相应法律责任。如您发现有侵权的内容,请联系我们。转载请注明出处:https://www.wpsshop.cn/w/你好赵伟/article/detail/463489
推荐阅读
相关标签


