- 1【Quart 框架——来源于Flask的强大且灵活的异步Web框架】
- 2IDEA支付宝小程序开发流程——授权登录_支付宝小程序 minidev identitykeypath
- 3【计算机网络】域名劫持无处遁形:基于HTTPDNS打造可靠且安全的域名解析体系_域名解析接口
- 4什么是同步整流和异步整流_同步整流和异步整流的区别
- 525K Stars! Open WebUI + Ollama + Llama3搭建本地私人ChatGPT
- 6Postman 接口测试神器_在线postman
- 7RENISHAW雷尼绍双读数头系统应用分享
- 8亚马逊云科技 EC2服务搭配SD Webui开箱即用的AIGC文生图/图生图平台_aws sd-webui 配额
- 9elasticsearch配置文件详解_cluster.name
- 10MyBatis查询数据库之四(动态SQL -- if、trim、where、set、foreach 标签)_insert into和if标签
聚类分析|基于密度的聚类方法DBSCAN及其Python实现
赞
踩
0. 基于密度的聚类方法
之前介绍过基于划分的聚类算法,如k-means和k-medoids,也介绍过基于层次聚类的方法。这两种方法本质上都是基于距离的算法,只能发现类圆形的聚类。
基于密度的方法(Density-based Methods)是基于密度的,可以克服基于距离的算法只能发现“类圆形”聚类的缺点。
主要思想:只要在给定半径邻近区域的密度超过某个阈值,就把它添加到与之相近的簇类中去。
这样的方法可以用来过滤噪声数据,并且可以发现任意形状的聚类。
基于密度的方法中,代表算法有:
- DBSCAN算法
- OPTICS算法
- DENCLUE算法
以下对DBSCAN(Density Based Spatial Clustering of Applications with Noise,DBSCAN)聚类算法进行介绍。
1. DBSCAN聚类算法的相关概念
DBSCAN是基于密度聚类中的经典算法,突出特色在于:
- 第一,利用小类的密度可达性(或称连通性),可发现任意形状的小类
- 第二,聚类同时可以发现数据中的噪声,也即离群点
DBSCAN聚类中有两个重要参数:
- 邻域半径 ε \varepsilon ε;
- 邻域半径 ε \varepsilon ε范围内包含的最少观测点个数,记为 m i n P t s minPts minPts。
基于这两个参数,DBSCAN聚类将样本观测点分成以下4类:
- 核心点(核心样本) P P P:若任意样本观测点 O O O的邻域半径 ε \varepsilon ε内的邻居个数不少于 m i n P t s minPts minPts,则称 O O O为核心点,记作 P P P
- 核心点 P P P的直接密度可达点 Q Q Q:若任意样本观测点 Q Q Q在核心点 P P P的邻域半径 ε \varepsilon ε范围内,则称点 Q Q Q为核心点 P P P的直接密度可达点。也称从点 P P P直接密度可达点 Q Q Q
- 核心点 P P P的密度可达点 Q Q Q:若存在一系列样本观测点 O 1 , O 2 , … , O n O_1, O_2, …, O_n O1,O2,…,On,且 O i + 1 ( i = 1 , 2 , . . . , n − 1 ) O_{i+1}(i=1,2,...,n-1) Oi+1(i=1,2,...,n−1)是 O i O_i Oi的直接密度可达点,且 O i = P , O n = Q O_i=P,O_n=Q Oi=P,On=Q,则称 Q Q Q是点 P P P的密度可达点,也称从点 P P P密度可达点 Q Q Q。
- 噪声点:除上述类型之外的样本观测点,均定义为噪声点。
如下图所示, q q q是一个核心样本, p p p由 q q q直接密度可达。
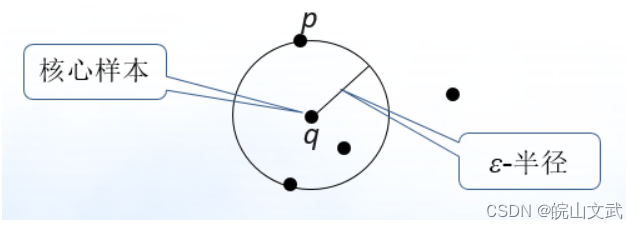
- 直接密度可达的传递性会导致密度可达!
- 若存在任意样本观测点 O O O,同时密度可达点 O 1 O_1 O1和点 O 2 O_2 O2,则称点 O 1 O_1 O1和点 O 2 O_2 O2是密度相连的。
2. DBSCAN聚类过程
设置邻域半径
ε
\varepsilon
ε和邻域半径
ε
\varepsilon
ε范围内包含的最少观测点个数
m
i
n
P
t
s
minPts
minPts。
在参数设定的条件下,DBSCAN聚类过程大致包括形成小类和合并小类两个阶段:
2.1 形成小类
- 从任意一个样本观测点 O i O_i Oi开始,在参数限定的条件下判断 O i O_i Oi是否为核心点。
- 若 O i O_i Oi是核心点,首先标记该点为核心点。然后,找到 O i O_i Oi的所有(如 m m m个)直接密度可达点(包括边缘点),并形成一个以 O i O_i Oi为“核心”的小类,记作 C i C_i Ci。 m m m个直接密度可达点(尚无小类标签)和样本观测点 O i O_i Oi的小类标签均为 C i C_i Ci
- 若 O i O_i Oi不是核心点,那么 O i O_i Oi可能是其他核心点的直接密度可达点,或密度可达点,亦或噪声点。若 O i O_i Oi是直接密度可达点或密度可达点,则一定会在后续的处理中被归到某个小类,带有小类标签 C j C_j Cj。若是噪声点,则不会被归到任何小类中,始终不带有小类标签
2.2 合并小类
- 判断带有核心点标签的所有核心点之间,是否存在密度可达和密度相连关系。若存在,则将相应的小类合并起来,并修改相应样本观测点的小类标签
3. DBSCAN算法的Python实现
在scikit-learn中,包含sklearn.cluster.DBSCAN的算法。DBSCAN()常用形式为:
DBSCAN(eps=0.5,min_samples=5,metric=’euclidean’, algorithm=’auto’)
- 1
参数说明:
- eps:设置半径,用以确定邻域的大小,默认0.5。
- min_samples:int,设置阈值MinPts。
- metric:采用的距离计算方式,默认是欧式距离。
- algorithm:取值{’auto’,’ball_tree’,’kd_tree’,’brute’}。用于计算两点间距离并找出最近邻的点。取值为:’auto’:由算法自动取舍合适的算法;’ball_tree’:用ball树来搜索;’kd_tree’:用kd树搜索;’brute’:暴力搜索。对于稀疏数据,一般取值’brute’。
3.1 DBSCAN算法示例
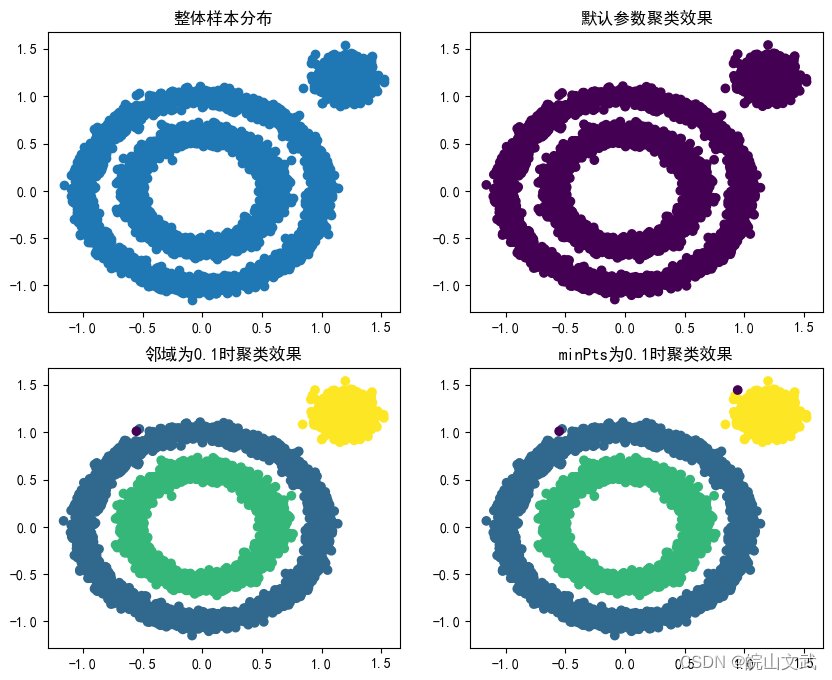
- 首先生成一组随机数据,为了体现DBSCAN在非凸数据的聚类优点,生成三簇数据,两组是非凸的。
- 直接使用DBSCAN默认参数,观看聚类效果
- 对DBSCAN的两个关键的参数eps和min_samples进行调参,当类别数太少,需要增加类别数,那么就可以减少邻域的大小,默认是0.5,下面减到0.1
- 将min_samples从默认的5增加到10,观看效果。
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
x1,y1=datasets.make_circles(n_samples=5000, factor=0.6,noise=0.05)
x2,y2 = datasets.make_blobs(n_samples=1000, n_features=2, centers=[[1.2,1.2]], cluster_std=[[.1]],random_state=9)
x = np.concatenate((x1, x2))
plt.rcParams['font.sans-serif']=['SimHei']
plt.rcParams['axes.unicode_minus']=False
plt.figure(figsize=(10,8))
plt.subplot(2,2,1)
# 观察总体样本分布
plt.title("整体样本分布")
plt.scatter(x[:,0],x[:,1], marker='o')
plt.subplot(2,2,2)
# 观察认参数聚类效果
plt.title("默认参数聚类效果")
x = np.concatenate((x1, x2))
y_pred = DBSCAN().fit_predict(x)
plt.scatter(x[:,0],x[:, 1], c=y_pred)
plt.subplot(2,2,3)
# 观察邻域为0.1时聚类效果
plt.title("邻域为0.1时聚类效果")
y_pred2 = DBSCAN(eps = 0.1).fit_predict(x)
plt.scatter(x[:,0],x[:,1],c=y_pred2)
plt.subplot(2,2,4)
# 观察minPts为10时聚类效果
plt.title("minPts为0.1时聚类效果")
y_pred3 = DBSCAN(eps = 0.1,min_samples =10).fit_predict(x)
plt.scatter(x[:,0],x[:,1],c=y_pred3)
plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
3.2 DBSCAN的异型聚类特点
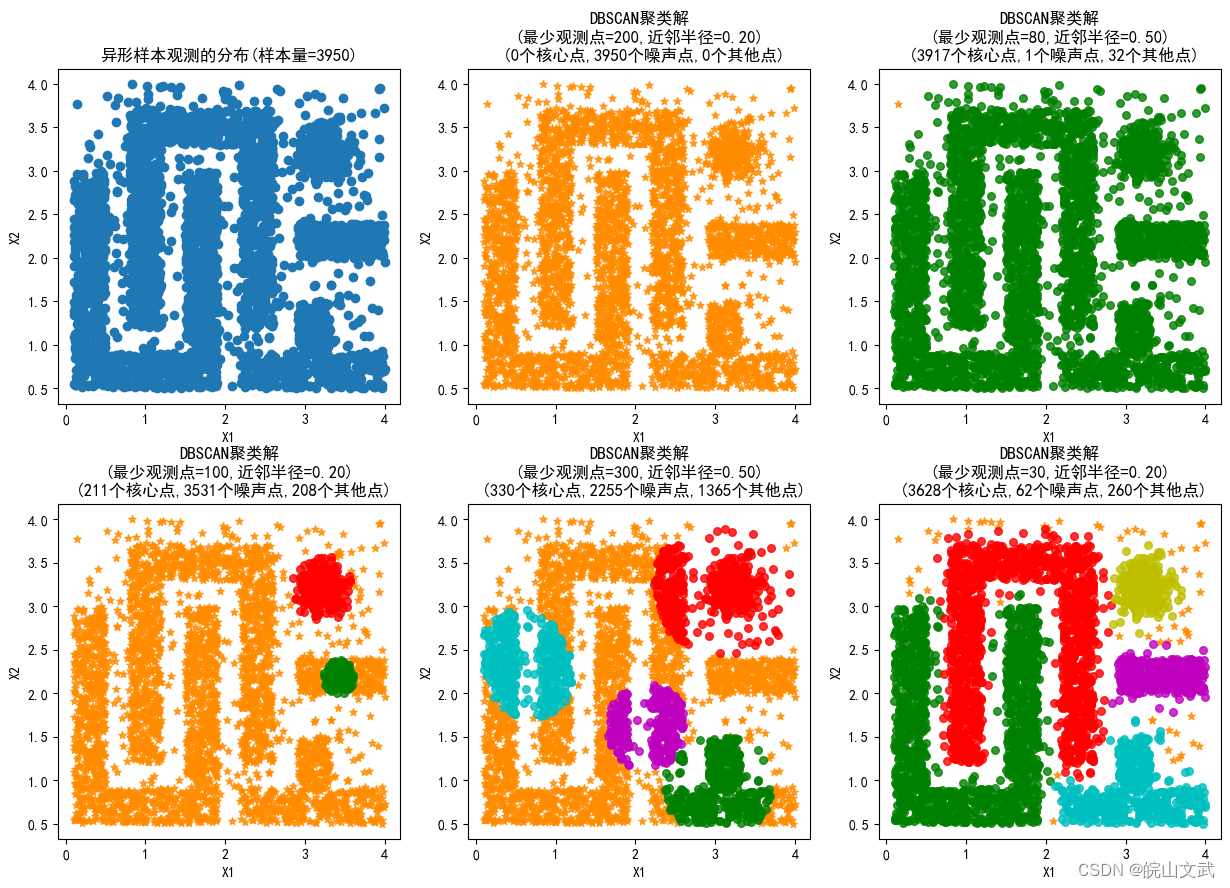
DBSCAN聚类的最大特点是能够发现任意形状的类。
DBSCAN算法对参数
ε
\varepsilon
ε(邻域半径)和
m
i
n
P
t
s
minPts
minPts极为敏感。以下进行相关参数设置试验以说明特性:
- 邻域半径 ε \varepsilon ε较小,且邻域范围内的最少观测点个数 m i n P t s minPts minPts较多
- 邻域半径 ε \varepsilon ε较大,且邻域范围内的最少观测点个数 m i n P t s minPts minPts较少
- 适中的参数设置
试验代码如下:
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from itertools import cycle
import warnings
warnings.filterwarnings(action = 'ignore')
%matplotlib inline
plt.rcParams['font.sans-serif']=['SimHei']
plt.rcParams['axes.unicode_minus']=False
from scipy.stats import norm
from sklearn.datasets import make_moons
from sklearn.cluster import DBSCAN,Birch,KMeans,estimate_bandwidth,MeanShift
X=pd.read_csv('异形聚类数据.txt',header=0)
fig=plt.figure(figsize=(15,10))
plt.subplot(231)
plt.scatter(X['x1'],X['x2'])
plt.title("异形样本观测的分布(样本量=%d)"%len(X))
plt.xlabel("X1")
plt.ylabel("X2")
colors = 'bgrcmyk'
EPS=[0.2,0.5,0.2,0.5,0.2]
MinS=[200,80,100,300,30]
Gid=1
for eps,mins in zip(EPS,MinS):
DBS=DBSCAN(min_samples=mins,eps=eps)
DBS.fit(X)
labels=np.unique(DBS.labels_)
Gid+=1
plt.subplot(2,3,Gid)
for i,k in enumerate(labels):
if k==-1: #噪声点
c='darkorange'
m='*'
else:
c=colors[i]
m='o'
plt.scatter(X.iloc[DBS.labels_==k,0],X.iloc[DBS.labels_==k,1],c=c,s=30,alpha=0.8,marker=m)
plt.title("DBSCAN聚类解\n(最少观测点=%d,近邻半径=%.2f)\n (%d个核心点,%d个噪声点,%d个其他点)"%(mins,eps,len(DBS.components_),
sum(DBS.labels_==-1),len(X)-len(DBS.components_)-sum(DBS.labels_==-1)))
plt.xlabel("X1")
plt.ylabel("X2")
fig.subplots_adjust(hspace=0.3)
fig.subplots_adjust(wspace=0.2)
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
4. DBSCAN算法的缺点
在DBSCAN算法中,有两个初始参数 ε \varepsilon ε(邻域半径)和 m i n P t s minPts minPts( ε \varepsilon ε邻域最小样本数)需要用户手动设置输入,并且聚类的类簇结果对这两个参数的取值非常敏感,不同的取值将产生不同的聚类结果,其实这也是大多数需要初始化参数聚类算法的弊端。


