- 1Win 10 UEFI + Ubuntu 18.04 UEFI 双系统 (by quqi99)_active命令只能用于固定mbr磁盘
- 2ChatGPT 能拯救程序员吗?
- 3【MySQL篇】第五篇——表的约束_mysql建表约束
- 4DDoS的攻击原理与防御方法_ddos攻击防御
- 5动态规划【2020第一版】_动态规划segmentation
- 6MariaDB 10.5,MySQL乱码问题,设置字符编码UTF8
- 7小白如何进入IT行业及如何选择培训机构_想学it怎样筛选培训机构
- 8Springboot计算机毕业设计基于微信小程序民宿预订系统【附源码】开题+论文+mysql+程序+部署
- 9领导暗示你辞职的五个方式,遇到了你是乖乖离开还是死扛到底?_老板暗示主动辞职如何应对
- 10分布式主键?雪花算法?号段模式?一文搞懂目前主流分布式主键解决方法
Nat.Neuro(IF=28+)| eQTL结合GWAS数据,单细胞MR还有多久!!
赞
踩
有何AI与医学:孟德尔随机化准备从大家问题多地方以及简单介绍写起来,然后补全整个分析流程!欢迎转发关注哦!

今天我们给大家带来一篇细胞水平eQTL分析结合GWAS数据揭示精神和神经疾病的新风险基因。那么细胞层面的eQTL数据,是不是可以让我们拿来做孟德尔随机化呢,再来个炫一点的名字,单细胞孟德尔随机化。
全文速览
迄今为止,大多数基因表达数量性状基因座(eQTL)的研究一直都是组织层面,已经探明eQTL不同脑组织中的差异,但是特定类型的细胞类型却没有。所以作者的这项研究中先做了8种来自前额叶皮层,颞叶皮层和白质的脑细胞eQTL,共鉴定了7607个基因,其中相当一部分(46%,3537/7607)显示出细胞类型特异性,在小胶质细胞中的特异性最强。细胞类型水平的eQTL比组织水平的eQTL影响有更强的特异性,并具有更大的效应值。紧接着作者利用脑细胞类型eQTL与全基因组关联研究(GWAS)的整合,揭示了神经精神疾病和神经退行性疾病的表达与疾病风险之间的新关系。对于大多数GWAS基因座,在单个细胞类型中单个基因共定位,为疾病病因学提供了新的线索。研究结果表明,脑细胞类型之间的基因表达的遗传调控存在显著差异,并揭示了疾病风险基因影响脑部疾病的潜在机制。
方法与结果
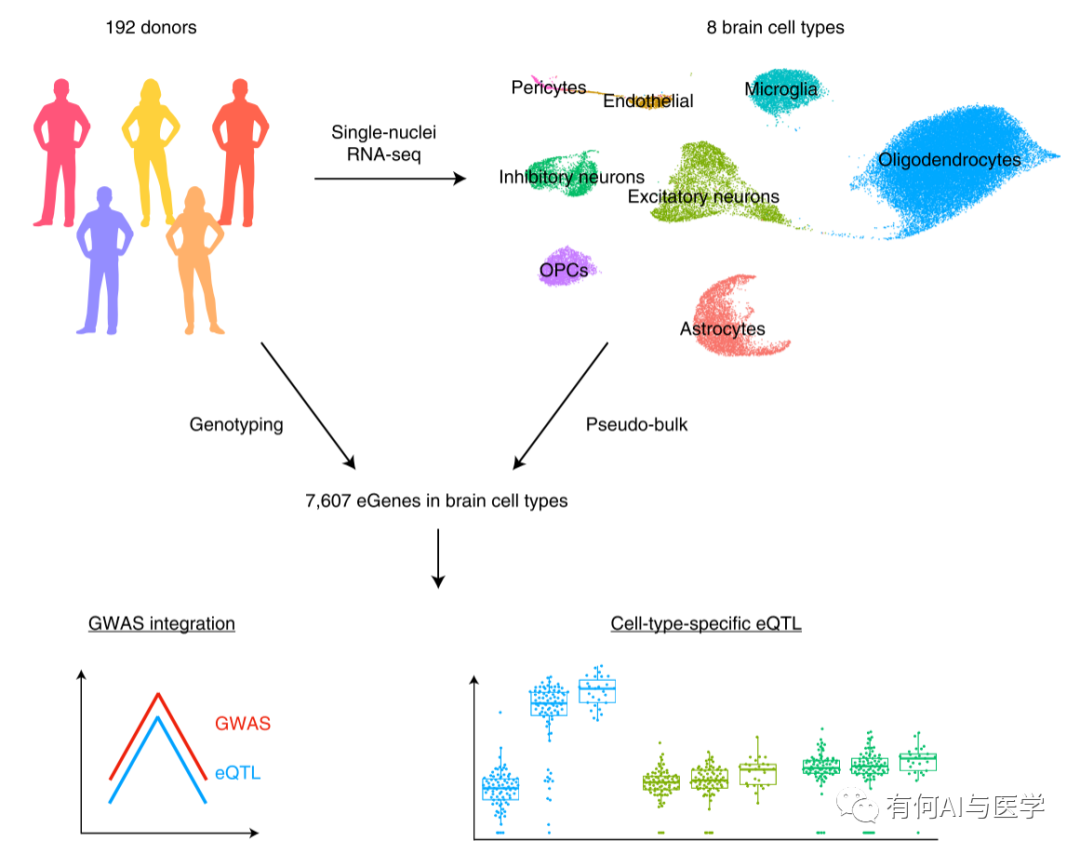
1-作者对来自148个独立个体的270个人脑样品中进行了单核RNA测序(RNA-seq)以及基因分型(DNA),整合了作者自己先前发表的单核转录组学研究,102个人脑样本,这些研究先前已进行了基因分型。总共获得了来自215个个体的373个大脑样本。经过质控,获得了8种细胞(每种细胞样本144-192),每类细胞基因数量在6940 - 14595个,基因分型测得530万个单核苷酸多态性(SNP)。

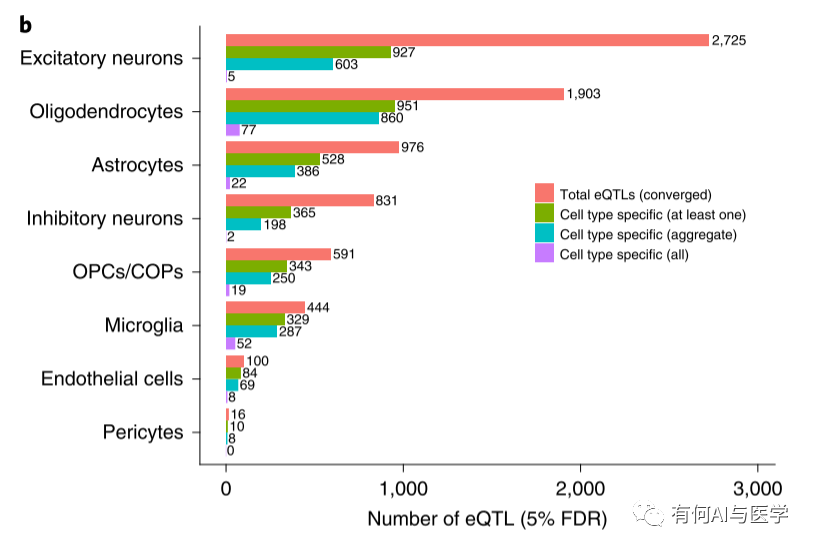
2-作者对上述得到的数据进行了eQTL分析,挑选每个表达基因的转录起始位点(TSS)周围的1Mb窗口内所有SNP,同时调整协变量,来鉴定顺势eQTL。8种不同脑细胞类型共鉴定7607个顺式eQTL基因(FDR<0.05),部分基因在不同类型细胞中均显著,去重后的基因数量为4799(如图a和b)。图b中我们也可以看出,当细胞数量越多,鉴定的出显著的基因越多,其成线性变化,也让我们可以猜测数据量增加还有鉴定出许多的基因位点。
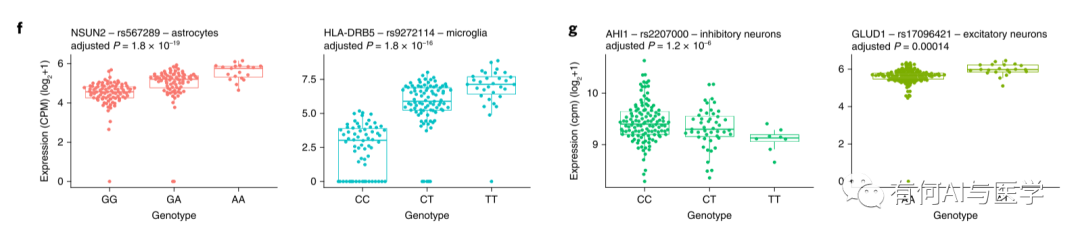
3-Fine-mapping分析去鉴定SNP对于顺势eQTL的因果作用。共定位了591个cis-eQTL(因果概率>50%),具有高因果概率的精细映射SNP包括rs2207000,作为抑制性神经元AHI1-eQTL的可能因果SNP(概率= 0.87),和rs17096421作为兴奋性神经元GLUD1-eQTL的可能的因果SNP(概率= 0.53)。

4-之后作者做了scATAC-seq(单细胞染色质转座酶可接近性分析),scATAC-seq能够提供染色质可接近性的信息,并揭示单个细胞中基因转录活跃的区域。结果看出ATAC-seq峰位于胶质细胞类型(星形胶质细胞、小胶质细胞、少突胶质细胞和少突胶质细胞前体细胞),在之前发现的顺式eQTL周围富集。这表明所发现的神经胶质eQTL,位于基因组中与细胞类型特异性基因调控功能相关的区域。
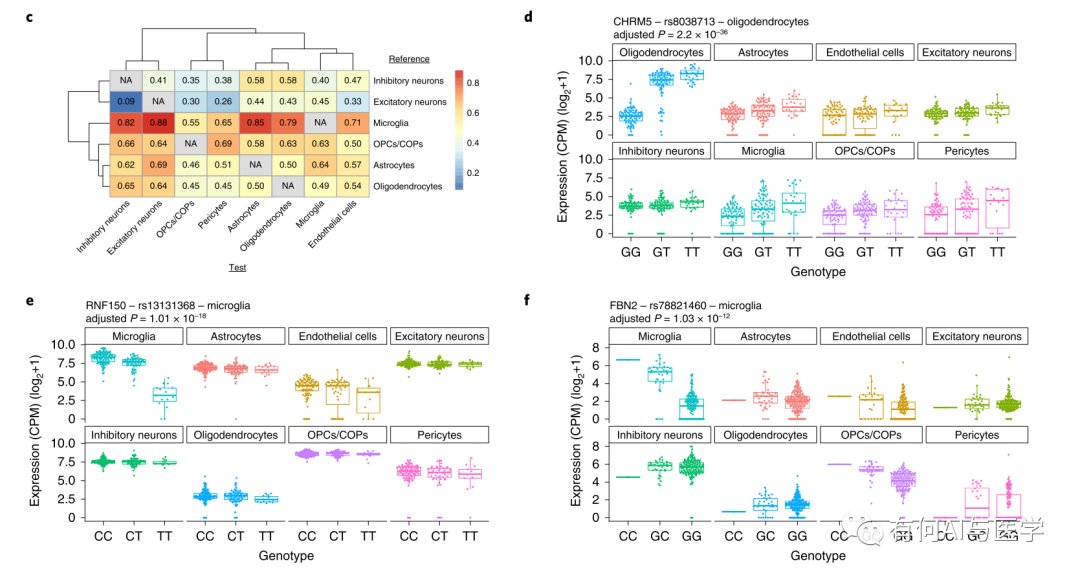
5-细胞类型特异性顺式eQTL效应值。这里作者对之前做的进一步探索,主要想表达不同基因eQTL在细胞类型与组织类型中的效应值有差别,在小胶质细胞顺式eQTL基因的特异性证据最强。例如:少突胶质细胞中的CHRM5、AVEN和TMPRSS5,以及小胶质细胞中的RNF150和FBN2等。之后又计算了这些细胞特异性的遗传效应在9%至88%。如上图C。
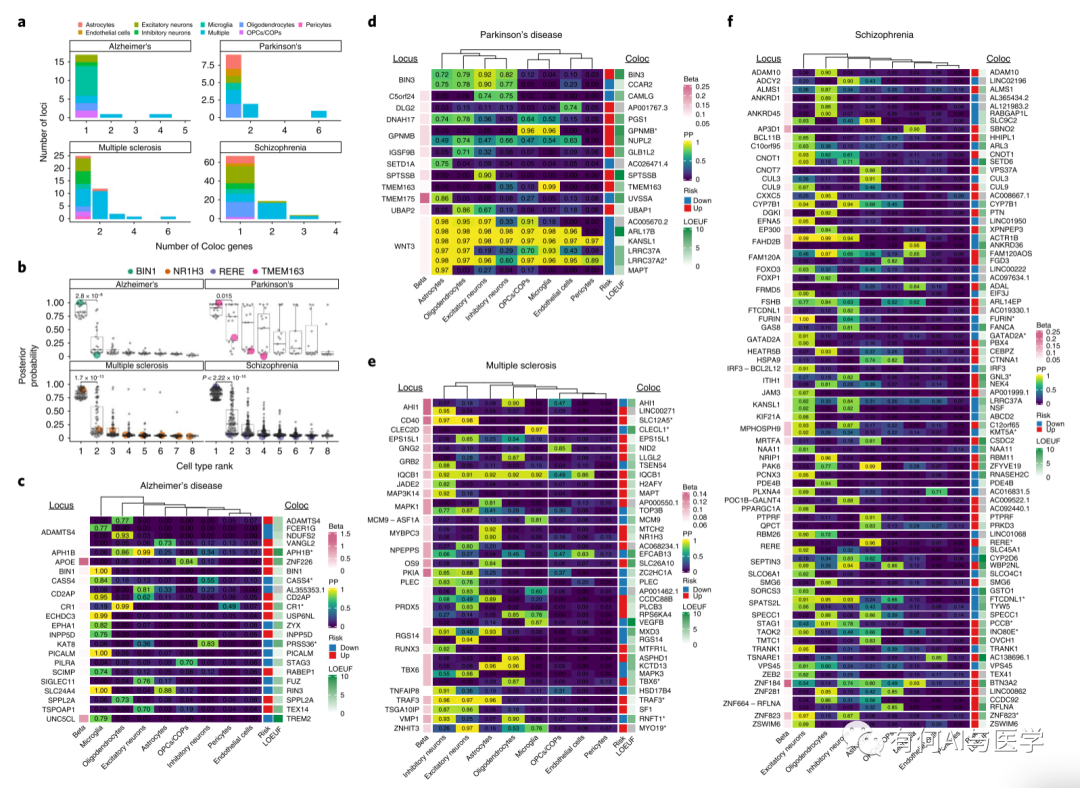
6-脑细胞类型eQTL与脑疾病的介导关联。作者这一部分作用使用共定位分析(Coloc),对8种脑细胞eQTL数据阿尔茨海默病(AD)、帕金森病(PD)、精神分裂症(SCZ)和多发性硬化症(MS)做共定位分析,结果如上。分别有15-46%eQTL在四种不同的病症GWAS基因座共定位的后验概率(PP)> 0.7。具体基因信息如图d-f。

7-风险变异与细胞类型调控元件的映射。这一步主要对上一步的分析进行探索注释,通过ChIP测序(PLAC-seq),两个AD相关GWAS SNP(rs10792832和rs3851179)关联到小胶质细胞PICALM的特异性增强子,而另一个AD相关GWAS SNP(rs10933431)关联到小胶质细胞INPP5D特异性增强子中。之后使用SNP2TFBS工具鉴定可以破坏特异性转录因子的结合的精细定位的SNP。
总结借鉴
读完文章我们应该对单细胞层面的eQTL有了解,那么单细胞层面的孟德尔随机化是不是就应运而生呢。其次本文的数据都给出了具体地址,可以用来学习。文章中的共定位方法,Fine-mapping,SNP2TFBS都是很值得借鉴的方法与软件,感兴趣的朋可以详读一下。
原文链接:



