- 1Kafka - Kafka 为啥抛弃 Zookeeper?
- 2EduCoder-Linux 与 Python编程2021-Python面向对象编程实训-第2关:三种贷款方式建模_本关的编程任务是,补全 7-2.py文件中 begin-end 区间的代码,实现用三种贷款方式建
- 3GitLab 查看版本信息
- 4SQL难点对比分析:IN 和 EXISTS 的用法对比_sql in 和 exsit的差别
- 5jmeter连接mysql数据库驱动_Jmeter连接Mysql数据库
- 6Oracle数据库分页解释小记
- 7【软考经验分享】软考-中级-嵌入式备考
- 8Android Studio设置字体大小_android studio 字体大小
- 9Gradle构建脚本基础(三)_settings.gradle
- 10基于ESP32的透明电视网络相册(可网页配置WIFI)_esp32电子相册
VM-UNet: Vision Mamba UNet for Medical Image Segmentation
赞
踩
VM-UNet: Vision Mamba UNet for Medical Image Segmentation
VM-UNet:基于视觉Mamba UNet架构的医学图像分割
论文链接:http://arxiv.org/abs/2402.02491
代码链接:https://github.com/JCruan519/VM-UNet
1、摘要
文中利用状态空间模型SSMs,提出了一种针对医学图像分割的U形架构模型,称为Vision Mamba UNet(VM-UNet)。具体地,引入了视觉状态空间(VSS)块作为基础模块,以捕捉丰富的上下文信息,并构建了一个不对称的编码器-解码器结构。在ISIC17、ISIC18和Synapse数据集上进行了全面实验,结果显示VM-UNet在医学图像分割任务中表现出良好的性能。
2、创新点
受VMamba[20]在图像分类任务中取得成功的影响,本文首次提出Vision Mamba UNet(VM-UNet),这是一种纯基于SSM的模型,旨在展示在医疗图像分割任务中的潜力。VM-UNet主要由编码器、解码器和跳跃连接三部分组成。编码器使用VMamba的VSS块进行特征提取,并通过拼接操作进行下采样。解码器包含VSS块和扩展操作,以恢复分割结果的大小。跳跃连接部分,为了突出最原始纯SSM模型的分割性能,采用了最简单的相加操作。
在器官分割和皮肤病变分割任务上进行了广泛实验,以展示纯SSM模型在医疗图像分割中的潜力。VM-UNet代表了纯SSM分割模型的最基本形式,没有包含任何特殊设计的模块。
本文的主要贡献可以总结如下:
1)提出了VM-UNet,这是首次探索纯SSM模型在医疗图像分割中的应用潜力;
2)在三个数据集上进行了全面实验,结果显示VM-UNet具有显著的竞争力;
3)为纯SSM模型在医疗图像分割任务中建立了基准,提供了有价值的见解,为开发更高效和有效的SSM分割方法铺平了道路。
3、原理
Preliminaries
在现代基于状态空间模型(SSM)的模型中,如结构化状态空间序列模型(S4)和Mamba,它们都依赖于经典的连续系统,该系统通过映射一维输入函数或序列 x ( t ) ∈ R x(t) ∈ R x(t)∈R,通过中间隐状态 h ( t ) ∈ R N h(t) ∈ R^{N} h(t)∈RN到输出 y ( t ) ∈ R y(t) ∈ R y(t)∈R。这个过程可以表示为线性常微分方程(ODE):
h
′
(
t
)
=
A
h
(
t
)
+
B
x
(
t
)
,
h^{'}(t) = Ah(t) + Bx(t),
h′(t)=Ah(t)+Bx(t),
y
(
t
)
=
C
h
(
t
)
,
(
1
)
y(t) = Ch(t), (1)
y(t)=Ch(t),(1)
其中 A ∈ R N × N A ∈ R^{N \times N} A∈RN×N表示状态矩阵, B ∈ R N × 1 B ∈ R^{N \times 1} B∈RN×1和 C ∈ R N × 1 C ∈ R^{N \times 1} C∈RN×1分别表示投影参数。
S4和Mamba将这个连续系统离散化,使其更适合深度学习环境。它们引入一个时间尺度参数 Δ \Delta Δ,并通过固定的离散化规则(如零阶保持法,ZOH)将 A A A和 B B B转换为离散参数 A ˉ \bar A Aˉ和 B ˉ \bar B Bˉ:
A
=
e
x
p
(
Δ
A
)
,
A = exp(\Delta A),
A=exp(ΔA),
B
=
(
Δ
A
)
(
−
1
)
∗
(
e
x
p
(
Δ
A
)
−
I
)
⋅
Δ
B
.
(
2
)
B = (\Delta A)^{(-1)} * (exp(\Delta A) - I) \cdot \Delta B. \ (2)
B=(ΔA)(−1)∗(exp(ΔA)−I)⋅ΔB. (2)
离散化后,SSM模型可以通过线性递归或全局卷积两种方式计算。线性递归和全局卷积分别定义为:
h
′
(
t
)
=
A
ˉ
h
(
t
)
+
B
ˉ
x
(
t
)
,
h^{'}(t) = \bar{A} h(t) + \bar{B} x(t),
h′(t)=Aˉh(t)+Bˉx(t),
h
′
(
t
)
=
C
h
(
t
)
.
(
3
)
h^{'}(t) = C h(t). \ (3)
h′(t)=Ch(t). (3)
K
=
(
C
B
ˉ
,
C
A
ˉ
B
ˉ
,
.
.
.
,
C
A
ˉ
L
−
1
B
ˉ
)
,
K = (C\bar{B}, C\bar{A} \bar{B}, ..., C \bar{A}^{L-1} \bar{B}),
K=(CBˉ,CAˉBˉ,...,CAˉL−1Bˉ),
y
=
x
∗
K
ˉ
,
(
4
)
y = x * \bar{K}, \ (4)
y=x∗Kˉ, (4)
其中 K ˉ ∈ R L \bar{K} ∈ R^{L} Kˉ∈RL表示结构化卷积核, L L L表示输入序列 x x x的长度。
Vision Mamba UNet (VM-UNet)
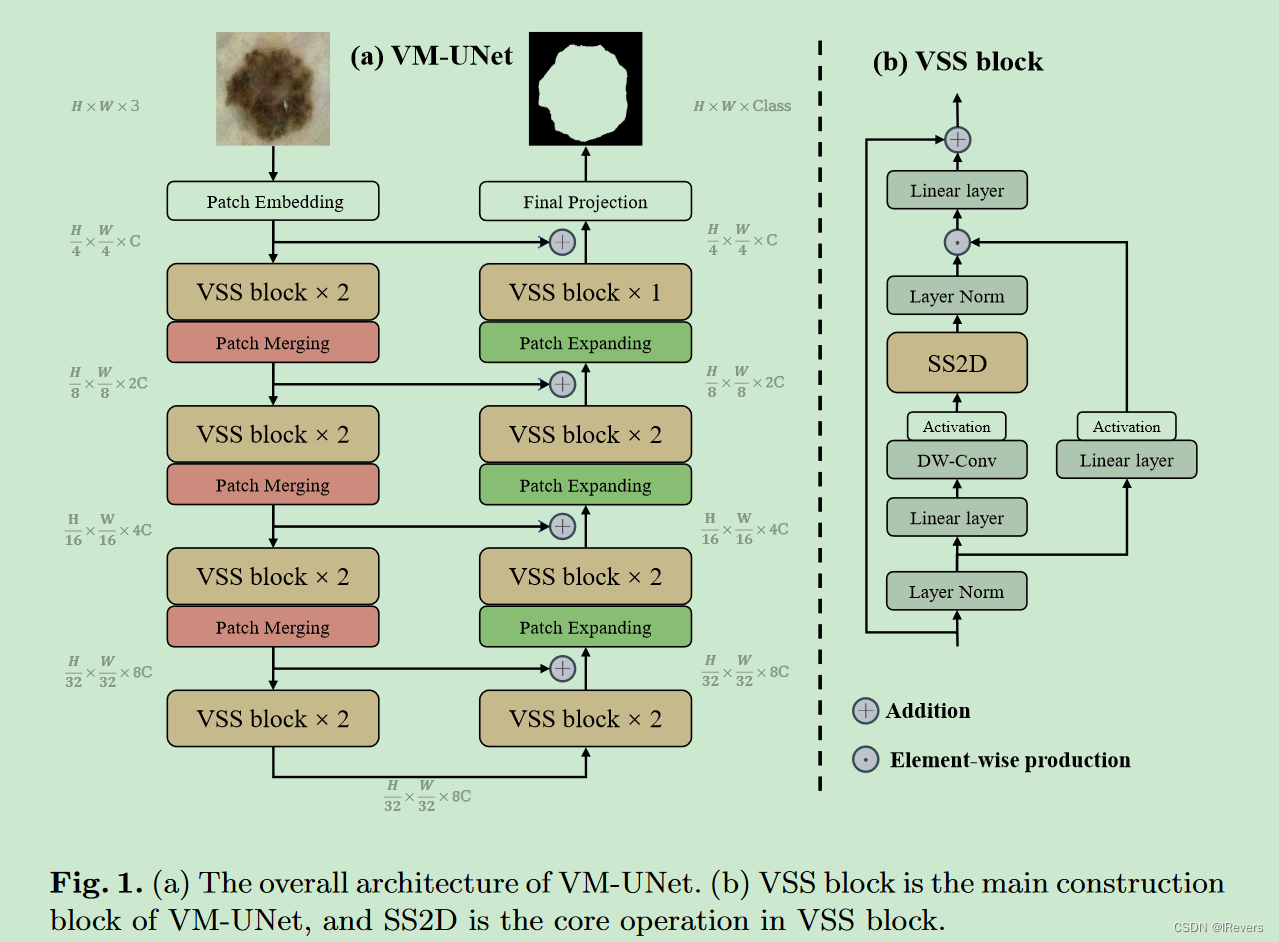
VM-UNet的整体架构如图1(a)所示。VM-UNet包括一个Patch Embedding层、一个编码器、一个解码器、一个Final Projection层以及跳跃连接。与先前的方法[9]不同,采用了非对称设计,而非传统的对称结构。
Patch Embedding层将输入图像 x ∈ R H × W × 3 x ∈ R^{H \times W \times 3} x∈RH×W×3分割成不重叠的 4 × 4 4 \times 4 4×4大小的patch,然后将图像维度映射到 C C C, C C C默认为96。这过程产生嵌入后的图像 x ′ ∈ R H 4 × W 4 × C x^{'} ∈ R^{\frac{H}{4} \times \frac{W}{4} \times C} x′∈R4H×4W×C。最后,使用Layer Normalization [7] 对 x ′ x^{'} x′进行标准化,然后将其输入编码器进行特征提取。编码器由四个阶段组成,每个阶段末尾进行patch合并操作,以减小输入特征的高宽同时增加通道数。四个阶段使用了[2, 2, 2, 2]个VSS块,每个阶段的通道数分别为[C, 2C, 4C, 8C]。
解码器同样分为四个阶段,前三个阶段开始时使用patch expanding操作,以减少特征通道数并增加高宽。四个阶段使用了[2, 2, 2, 1]个VSS块,每个阶段的通道数为[8C, 4C, 2C, C]。解码器之后,使用Final Projection层恢复特征的大小,以匹配分割目标。具体来说,通过4倍的ppatch expanding进行上采样,恢复特征的高宽,然后通过投影层恢复通道数。
对于跳跃连接,采用简单的相加操作,没有额外的复杂性,不会引入额外参数。
VSS block
VSS块源自于VMamba模型[20],是VM-UNet的核心模块,如图1(b)所示。输入首先经过层归一化,然后分为两个分支。在第一个分支中,输入通过一个线性层后接一个激活函数处理。在第二个分支中,输入经过线性层、深度可分离卷积DWConv和激活函数处理,然后送入2D选择性扫描(SS2D)模块以进一步提取特征。接着,特征通过层归一化进行规范化,然后与第一个分支的输出进行逐元素相乘,以融合两条路径。最后,特征通过一个线性层混合,并与残差连接相结合,形成VSS块的输出。本文默认使用SiLU[14]作为激活函数。

SS2D模块由三个部分组成:scan expanding操作、S6块和scan merging操作。如图2所示,这四个方向的扫描能够将输出图像恢复到与输入相同的大小。S6块源自Mamba[16],在S4[17]的基础上引入了选择性机制,通过调整SSM的参数来适应输入。这样,模型能够区分并保留重要的信息,同时过滤掉无关的细节。S6块的伪代码在算法1中给出。

Loss function
VM-UNet的引入旨在验证纯基于状态空间模型(SSM)在医学图像分割任务中的应用潜力。因此,仅采用基础的二元交叉熵损失(BceDice损失)和Dice损失(CeDice损失),分别针对二分类和多分类任务,如公式5和6所示。
BceDice损失函数为:
L
B
c
e
D
i
c
e
=
λ
1
L
B
c
e
+
λ
2
L
D
i
c
e
.
(5)
L_{BceDice} = \lambda_1 L_{Bce} + \lambda_2 L_{Dice}. \quad \text{(5)}
LBceDice=λ1LBce+λ2LDice.(5)
CeDice损失函数为:
L
C
e
D
i
c
e
=
λ
1
L
C
e
+
λ
2
L
D
i
c
e
,
(6)
L_{CeDice} = \lambda_1 L_{Ce} + \lambda_2 L_{Dice}, \quad \text{(6)}
LCeDice=λ1LCe+λ2LDice,(6)
其中, N N N表示样本总数, C C C表示类别总数。 y i y_{i} yi和 y ^ i \hat{y}_i y^i分别代表真实标签和预测值。 y i , c y_{i,c} yi,c是一个指示符,如果样本 i i i属于类别 c c c则为1,否则为0。 y ^ i , c \hat{y}_{i,c} y^i,c是模型预测样本 i i i属于类别 c c c的概率。 ∣ X ∣ |X| ∣X∣和 ∣ Y ∣ |Y| ∣Y∣分别代表真值和预测结果。 l a m b d a 1 lambda_1 lambda1和 l a m b d a 2 lambda_2 lambda2是损失函数的权重,通常默认设置为1。
具体地,BCE损失计算如下:
L
B
c
e
=
−
1
N
∑
i
=
1
N
[
y
i
log
(
y
^
i
)
+
(
1
−
y
i
)
log
(
1
−
y
^
i
)
]
.
(7)
L_{Bce} = -\frac{1}{N} \sum_{i=1}^{N} [y_i \log(\hat{y}_i) + (1 - y_{i}) \log(1 - \hat{y}_i)]. \quad \text{(7)}
LBce=−N1i=1∑N[yilog(y^i)+(1−yi)log(1−y^i)].(7)
而Cross-Entropy损失计算为:
L
C
e
=
−
1
N
∑
i
=
1
N
∑
c
=
1
C
y
i
,
c
log
(
y
^
i
,
c
)
.
(7)
L_{Ce} = -\frac{1}{N} \sum_{i=1}^{N} \sum_{c=1}^{C} y_{i,c} \log(\hat{y}_{i,c}). \quad \text{(7)}
LCe=−N1i=1∑Nc=1∑Cyi,clog(y^i,c).(7)
公式(7)中的最后一项是Dice损失,它衡量了预测和真值的交集与两者并集的比例。
4、实验
Datasets
ISIC17和ISIC18数据集:国际皮肤图像协作组织2017年和2018年的挑战数据集(ISIC17和ISIC18)[8,1,12,2]是两个公开的皮肤病变分割数据集,分别包含2,150和2,694张带分割标签的皮肤镜像。作者遵循先前工作[28],以7:3的比例划分数据集作为训练和测试集。具体来说,ISIC17数据集中,训练集包含1,500张图像,测试集包含650张图像。ISIC18数据集中,训练集包括1,886张图像,而测试集包含808张图像。对于这两个数据集,我们详细评估了包括平均交并比(mIoU)、Dice相似系数(DSC)、准确率(Acc)、敏感性(Sen)和特异性(Spe)在内的多个指标。
Synapse多器官分割数据集(Synapse):Synapse[19,3]是一个公开的多器官分割数据集,包含30个腹部CT病例,共有3,779张轴向腹部临床CT图像,涵盖了8种腹部器官(主动脉、胆囊、左肾、右肾、肝脏、胰腺、脾脏和胃)。按照先前研究[10,9]的设置,我们使用18个病例进行训练,12个病例进行测试。对于这个数据集,我们报告Dice相似系数(DSC)和95% Hausdorff距离(HD95)作为评估指标。
Implementation details
在实施细节部分,作者遵循先前研究[28,9]的做法,将ISIC17和ISIC18数据集的图像调整为 256 × 256 256 \times 256 256×256尺寸,而Synapse数据集的图像调整为 224 × 224 224 \times 224 224×224。为了防止过拟合,我们采用了随机翻转和随机旋转等数据增强技术。在ISIC17和ISIC18数据集上,我们使用BceDice损失函数,而在Synapse数据集上采用CeDice损失函数。我们设置批次大小为32,并使用AdamW优化器[23],初始学习率为 1 e − 3 1e-3 1e−3。CosineAnnealingLR调度器[22]被选用,最大迭代次数为50,最小学习率为 1 e − 5 1e-5 1e−5。训练轮数设置为300。对于VM-UNet,使用预训练在ImageNet-1k上的VMamba-S的权重初始化编码器和解码器。所有实验都在单个NVIDIA RTX A6000 GPU上进行。
Main results
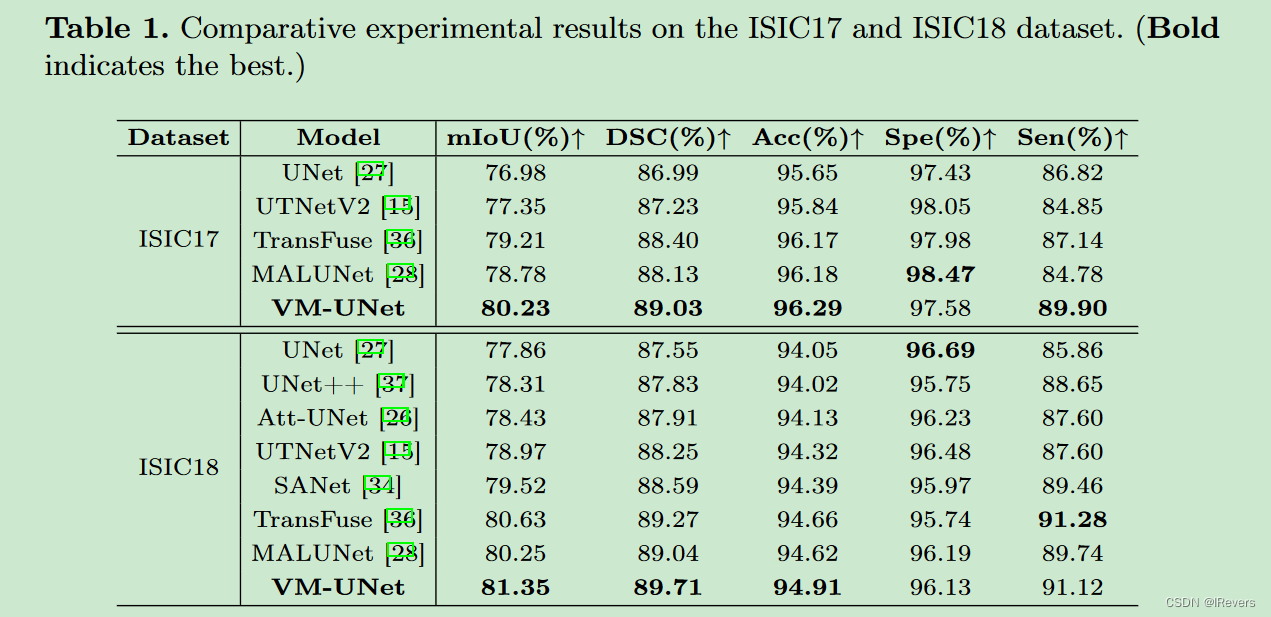
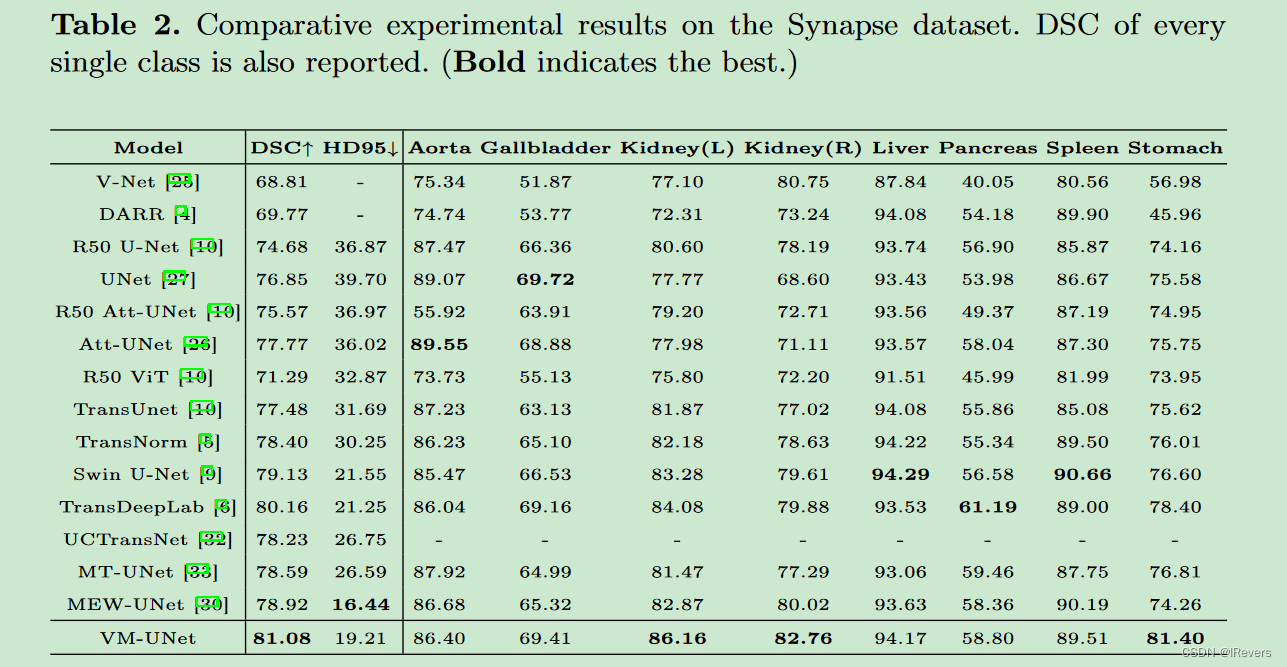
作者对VM-UNet与当前最先进的模型进行了比较,并在表1中展示了实验结果。实验评估了模型在ISIC17、ISIC18和Synapse等常用医疗图像分割数据集上的性能,包括 Dice 约束系数(Dice Score)、Intersection over Union (IoU) 和平均 Hausdorff 距离(Mean Hausdorff Distance,MHD)等关键指标。VM-UNet在这些指标上显示出与同类模型相当甚至优于的结果,证明了其在处理医学图像时的有效性和竞争力。这些对比实验不仅验证了文中所提的设计策略,也为基于状态空间模型的医疗图像分割提供了新的参考点。
Ablation studies
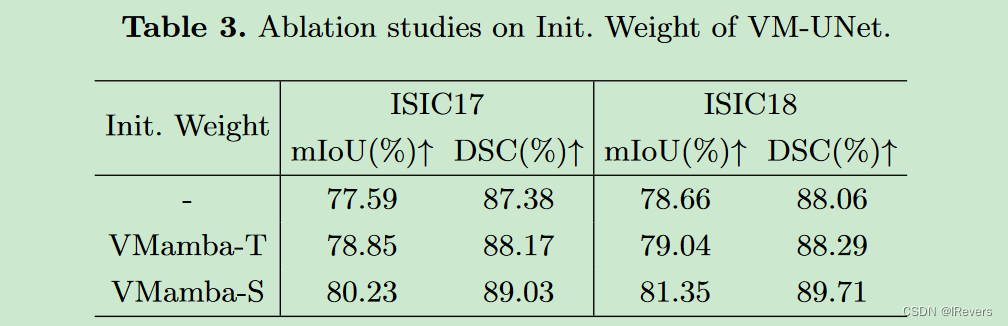
本节针对ISIC17和ISIC18数据集对VM-UNet的初始化进行了消融实验。我们分别使用预训练的VMamba-T和VMamba-S权重初始化VM-UNet。实验结果显示(见表3),更强大的预训练权重显著提升了VM-UNet的下游性能,这表明VM-UNet在很大程度上受到预训练权重的影响。
4、总结
文中引入了一种基于状态空间模型的纯模型VM-UNet,作为医学图像分割的基线。通过VSS块构建VM-UNet,并使用预训练的VMamba-S初始化权重。在皮肤病变和多器官分割数据集上的全面实验表明,纯基于状态空间模型的模型在医学图像分割任务中表现出强大的竞争力。


