- 1Xray-基础详细使用_xray使用教程
- 2数据结构单链表实现通讯录_前插法写通讯录
- 33W字吃透:SpringCloud gateway底层原理和实操_技术自由圈
- 4Caddi Programming Contest 2021(AtCoder Beginner Contest 193)-B - Play Snuke-题解_atcoder 193
- 5单片机产品低功耗设计_单片机低功耗
- 6flink checkpoint配置整理_flinkcdc checkpoint
- 7线性代数-行列式-p1 矩阵的秩
- 8【C++】双指针算法:和为s的两个数字
- 9鸿蒙南向开发环境的搭建(OpenHarmony)
- 10MCU低功耗设计(二)实践_iar 功耗 工具
开源分子对接程序rDock使用方法(1)-Docking in 3 steps_rdock每次对接结果不同
赞
踩
前言
rDock是一个快速、多功能的开源对接程序,可用于将小分子配体与蛋白质或核酸受体的对接;选用不同的对接模式可以完成考虑受体结合水的分子对接(Docking with explicit waters)以及药效团限制性对接(Docking with pharmacophore restraints),也可以用来做高通量虚拟筛选(HTVS)。
本文介绍 rDock用于受体-配体的标准对接(Docking in 3 steps),为研究其他模式下的分子对接做准备。
一、Docking in 3 steps 标准对接
rDock官网
rDock的介绍、Linux系统上本地安装请参考系列博文:开源分子对接程序rDock的安装及使用流程
rDock 的基本对接步骤及注意事项
包括3步:定义对接体系、产生对接位点和分子对接。
NOTES:
- 为受体(蛋白质或核酸)准备MOL2文件:需要考虑到原子类型问题,建议准备一个全原子MOL2文件,rDock会自动删除非极性氢。确保在$RBT ROOT/data/sf/RbtionicToms.rm中定义了任何非标准原子名称和子结构名称,以便正确分配分布式形式电荷。确保Tripos原子类型设置正确。rDock使用Tripos类型来推导其他关键的原子性质,如原子序数和杂化态。rDock MOL2解析器是为读取CCDC/Astex protein.MOL2文件而开发的,因此此验证集是事实上的标准参考。如果您怀疑某个特定的MOL2文件是否适合rDock,则应将其与CCDC/Astex MOL2文件的格式进行比较。关于Mol2格式,可以参考化学分子Mol2文件格式与使用注意事项。
- 准备定义系统的.prm。必须定义得受体参数包括SECTION MAPPER和SECTION CAVITY。如果您希望激活对接位点附近末端OH和NH3+基团的采样,请确保您定义了RECEPTOR_FLEX参数。
- 使用rbcavity生成对接站点(.as)文件;如果你想使用参考配体腔定义对接位点的方法,需要受体结合位点中的配体结构。
- 准备您想要对接的配体SD文件,需要注意SD文件解析的原子类型问题。特别是,要确保形式电荷和形式键级是一致的,这样文件中就不会有价键错误。rDock将报告任何感知到的价态误差,但仍然会完成。请注意,rDock在对接过程中从不采样键长、键角、环构象或不可旋转键,因此用户需要确保初始构象应该是合理的。
- 事先运行一个小的测试计算,以检查系统是否定义正确。例如,使用一个小的配体SD文件,仅使用打分选项(-p score.prm)和-T 2选项运行rbdock以生成详细输出用于检查。输出将包括受体原子特性、配体原子特性、灵活性参数、评分功能参数和对接协议参数。满足要求后,进行较大规模的计算。
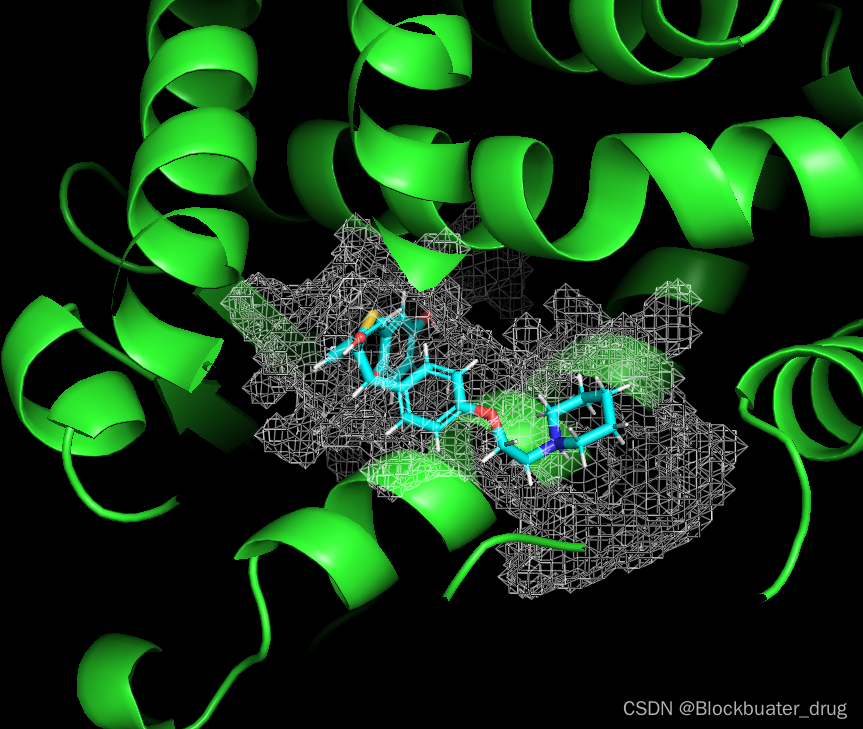
二、 三步对接案例
rDock对接案例输入文件来源:
人雌激素受体α配体结合结构域与拮抗剂配体4-D的复合物,RCSB下载 pdb id 1SJ0 。
receptor文件:下载1SJ0,加氢,加电荷,删除水分子,选中受体结构,保存为1sj0_rec.mol2
ligand文件:选中配体文件,保存为1sj0_ligand.sd
Step 1. 结构文件准备
mkdir 1sj0_workdir
cd 1sj0_workdir
- 1
- 2
通过prm文件定义对接体系。
prm 文件是 rDock所特有的文件格式,有以下作用:系统定义文件,评分函数定义文件,搜索协议定义文件
以下是ASTEX数据集的.prm文件示例:
RBT_PARAMETER_FILE_V1.00 TITLE 1sj0_DUD RECEPTOR_FILE 1sj0_rdock.mol2 RECEPTOR_FLEX 3.0 SECTION MAPPER SITE_MAPPER RbtLigandSiteMapper REF_MOL 1sj0_ligand.sd RADIUS 6.0 SMALL_SPHERE 1.0 MIN_VOLUME 100 MAX_CAVITIES 1 VOL_INCR 0.0 GRIDSTEP 0.5 END_SECTION SECTION CAVITY SCORING_FUNCTION RbtCavityGridSF WEIGHT 1.0 END_SECTION
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
将以上内容保存为cdk2_rdock.prm,受体结构mol 2文件为cdk2_rdock.mol2,位于结合位点的已知配体结合pose的文件为xtal-lig.sd。
使用的时候我们只需要修改以上内容即可。关于.prm文件的注意事项可以参考如下:
- 文件的第一行必须是RBT PARAMETER FILE V1.00,前面不能有空格;
- 注释行:注释行应在第一列中以#字符开头,前面不能有空格;
- 对于关键字TITLE, SECTION或END_SECTION:
· 关键字必须从第一列开始,前面不能有空格
·关键字TITLE 应该在文件中只出现一次,用于提供标题字符串,通过各种脚本显示,如运行rbscreen.pl,关键字后面应该跟一个空格字符,然后是标题字符串,其中可能包含空格。如果标题行出现的次数一次以上,使用最后一次的记录。
·关键字SECTION可以出现多次,并且应该始终与结束END_SECTION配对;关键字后面应该跟一个空格字符,然后是节名,它本身可能不包含空格。在.prm文件中,所有节名称都必须是唯一的。所有SECTION / END_SECTION节中的参数名/值对属于该节。
·在TITLE和SECTION关键字之后需要有一个空格字符,否则该部分的后续参数将被忽略。 - 参数名/值对:参数名称/值对被读取为自由格式的文本,并且可以有前缀、后缀,并由任意空格分隔。这意味着参数名称和值字符串本身不允许包含任何空格。值字符串被解释为适合该参数的数值、字符串或布尔值。布尔值应输入为TRUE或NULL大写字符串。
- prm文件不允许TAB出现。
Step 2. 产生对接位点

以上文件准备就绪,进入到以上文件的目录,用rbcavity命令生成对接空间:
rbcavity -was -d -r 1sj0_rdock.prm
- 1
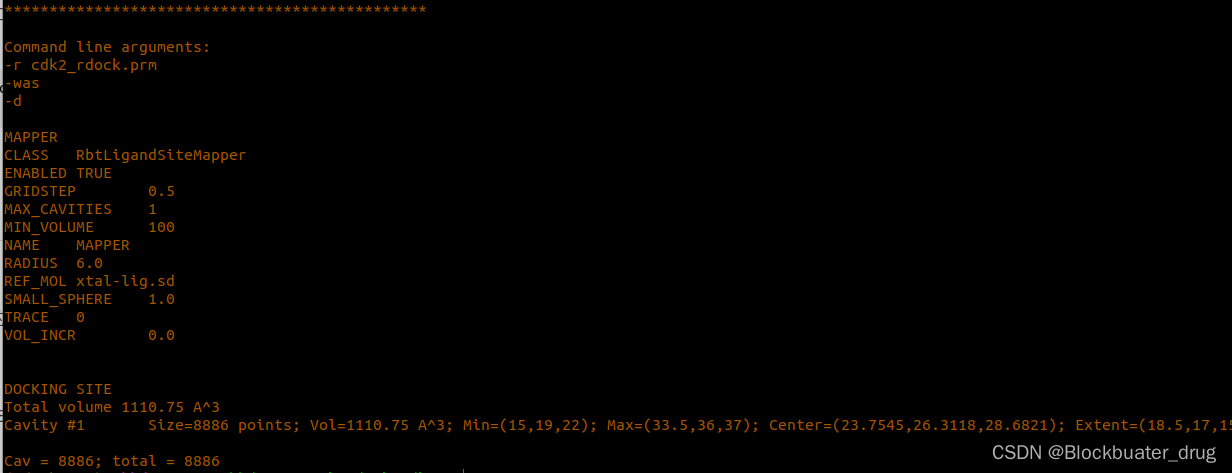 使用-d参数将生成网格“.grd”文件。该文件可以在pymol中查看:
使用-d参数将生成网格“.grd”文件。该文件可以在pymol中查看:
pymol 1sj0_rdock.mol2 1sj0_ligand.sd 1sj0_rdock_cav1.grd
- 1
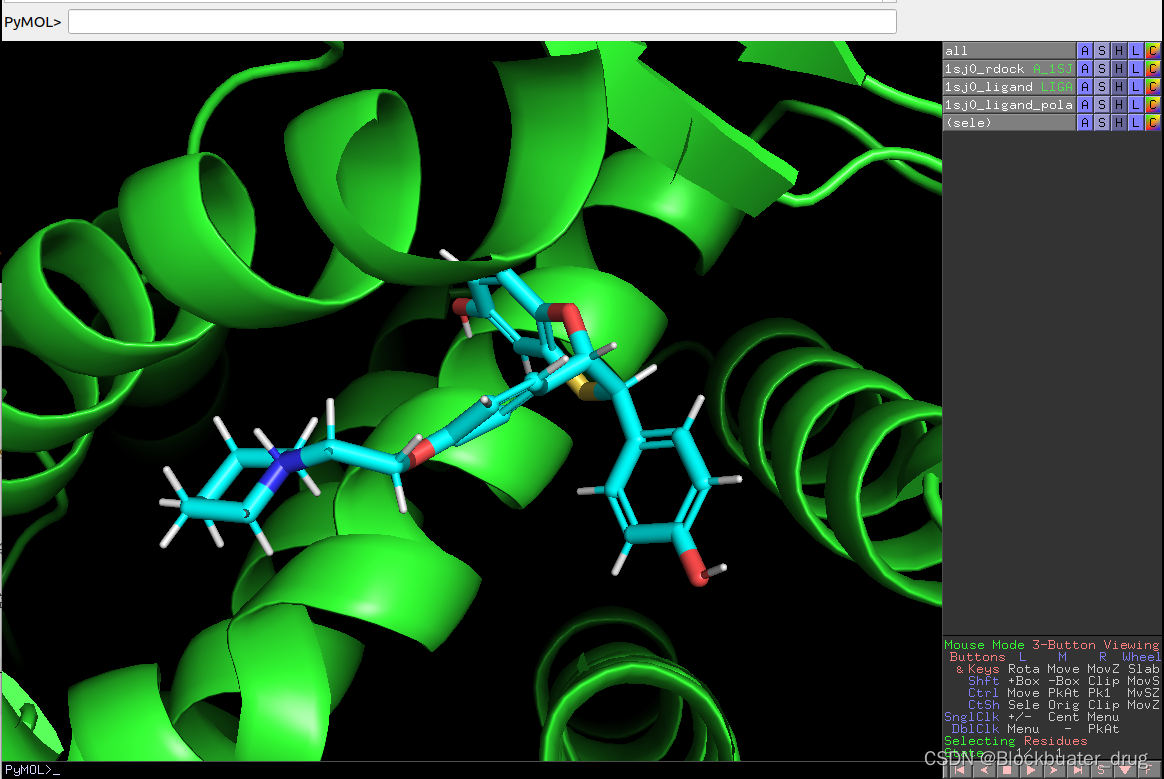
在pymol命令行输入以下:
isomesh cavity, 1sj0_rdock_cav1, 0.99
- 1
便于查看,调整了透明度。cavity基本覆盖了配体的空间,就是在这个区域进行对接。如果不合适,可以调整 .prm文件中的参数MIN_VOLUME,GRIDSTEP和MAX_CAVITIES。
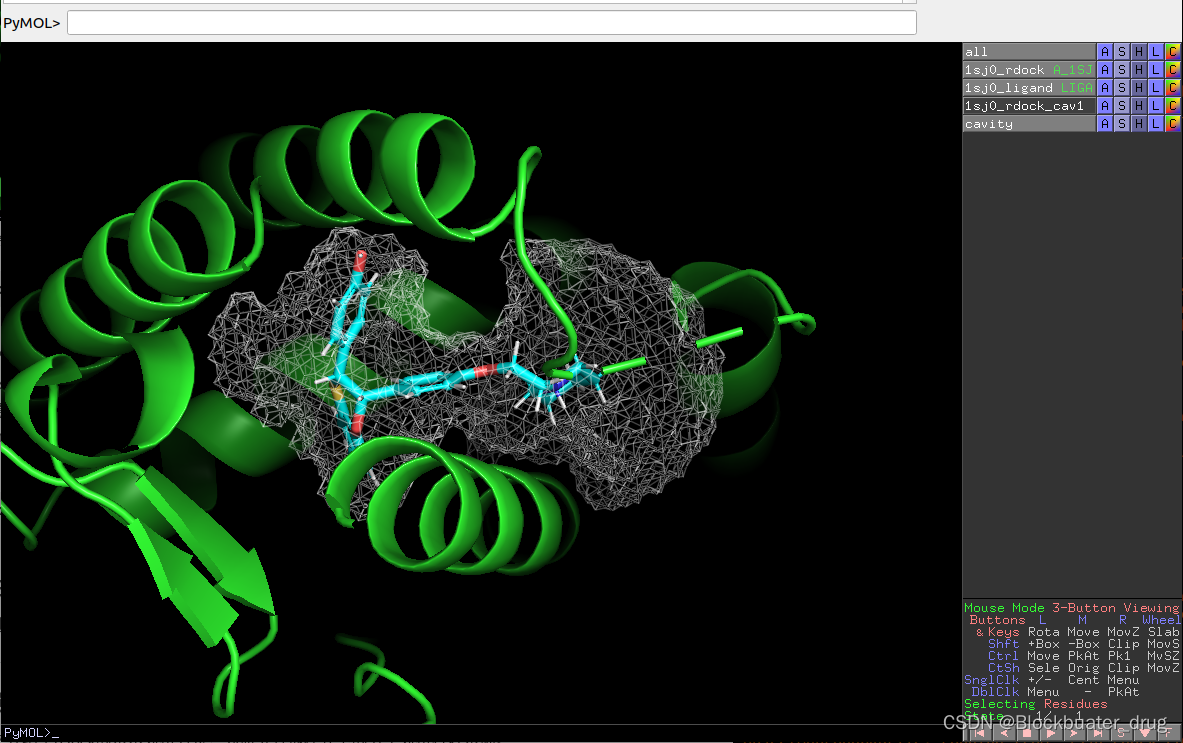
Step 3. 运行分子对接
3.1 检查输入文件
将以上生成文件置于1sj0_workdir文件夹中, 文件内容如下:

3.2 测试-只进行打分
rbdock -i 1sj0_ligand.sd -o output-score -r 1sj0_rdock.prm -p score.prm -T 2
- 1
结果为output-score.sd 文件。输出部分如下:
3.3 运行rdock
运行配体1sj0_ligand.sd的重对接,可以使用以下命令,对每个配体运行50次:
rbdock -i 1sj0_ligand.sd -o output-rdock -r 1sj0_rdock.prm -p dock.prm -n 50
- 1
运行片刻,结果为output-rdock.sd 文件。最后显示“END OF RUN”输出部分如下:
三、 结果查看
在MOE中查看,可以通过SCORE排序。
moe 1sj0_rdock.mol2 output-dock.sd
- 1
rdock的结果:
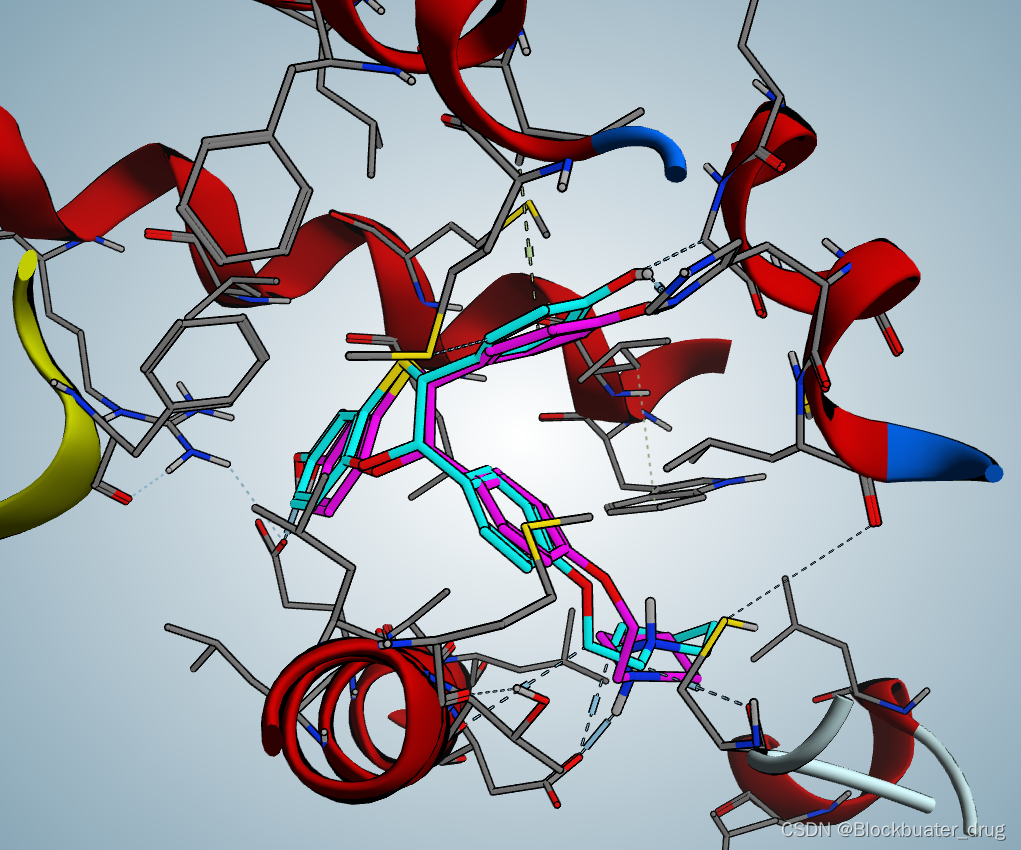
总结
本文介绍了rDock的基本对接方法,包括结构文件准备、产生对接位点,运行分子对接三个部分,为研究其他模式下的分子对接打好基础。
参考资料
- https://bbdrug.blog.csdn.net/article/details/136050880
- https://rdock.github.io/



