SNP的rsID与位置信息的相互匹配 rsID/ chr:pos conversion_b37 snp138
赞
踩
SNP的rsID与位置信息的相互匹配 rsID/ chr:pos conversion
SNP的rsID与位置信息的相互匹配 rsID/ chr:pos conversion – GWASLab – GWAS实验室
本文内容:
| 1 2 3 4 5 6 7 8 9 |
|
rsID与染色体位置信息的匹配或转换是生物信息学研究中必不可少的,但却又是十分繁琐的一项步骤,很多同学都在纠结这个问题,本文将总结常用的转换方法,以供参考:
转换前需要考虑的问题:
- 变异的标准化 :Variant Normalization 变异的标准化
- 参考基因组的版本 :人类参考基因组 Human reference genome hg19/hg38/GRCh37/GRCh38/b37/hs375d

1.个别SNP的转换:
直接使用dbsnp(详见:SNP数据库 – dbSNP)查询,查询个别SNP时方便快捷,可以直接搜rsID
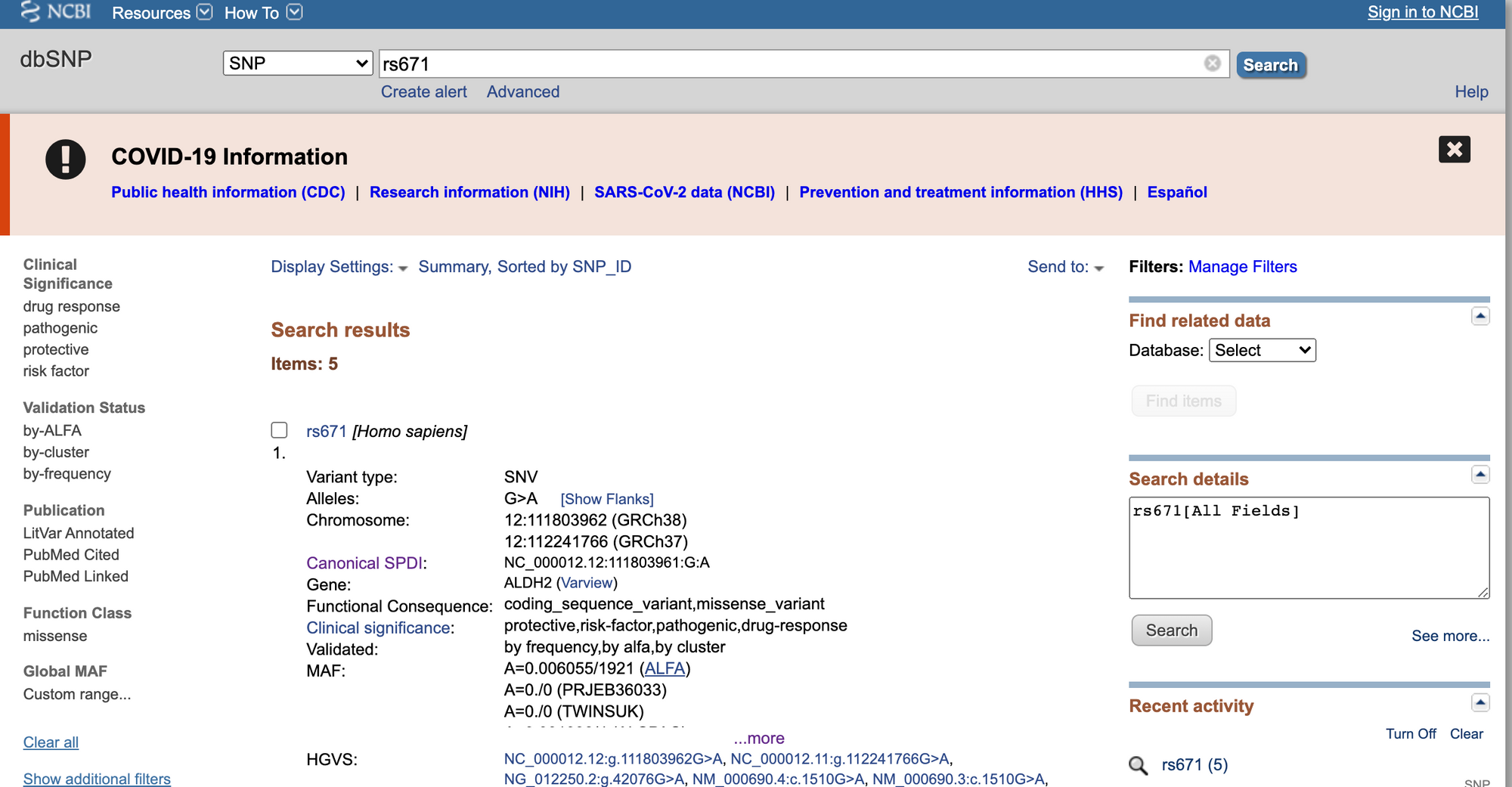
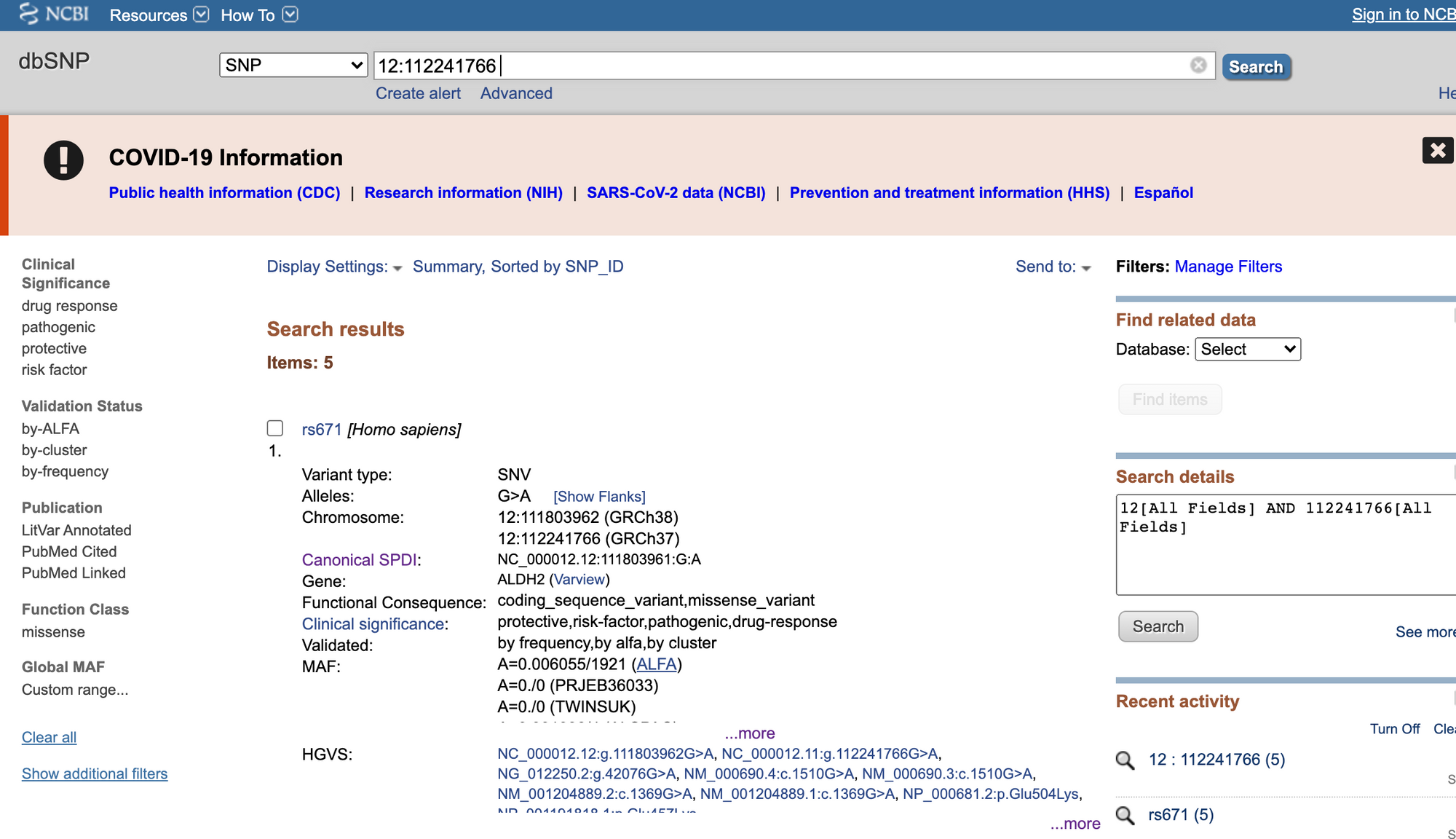
也可以以chr:pos的形式搜rsID
2.少量转换可用的网页工具 (100,000 snp以内)
2.1 SNPnexus : SNP Annotation Tool
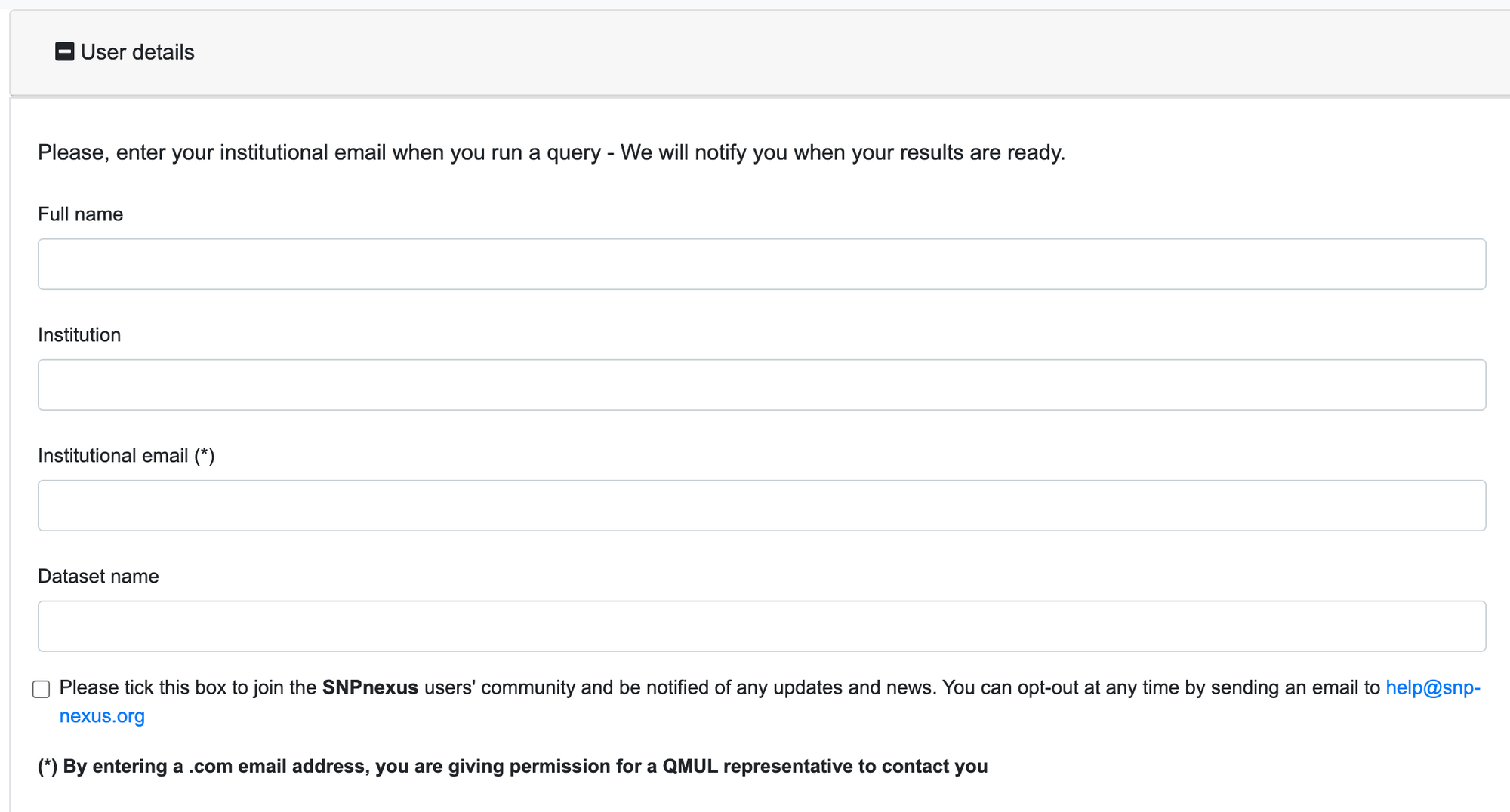
首先输入用户信息,学术用途是免费的,使用自己的edu邮箱即可
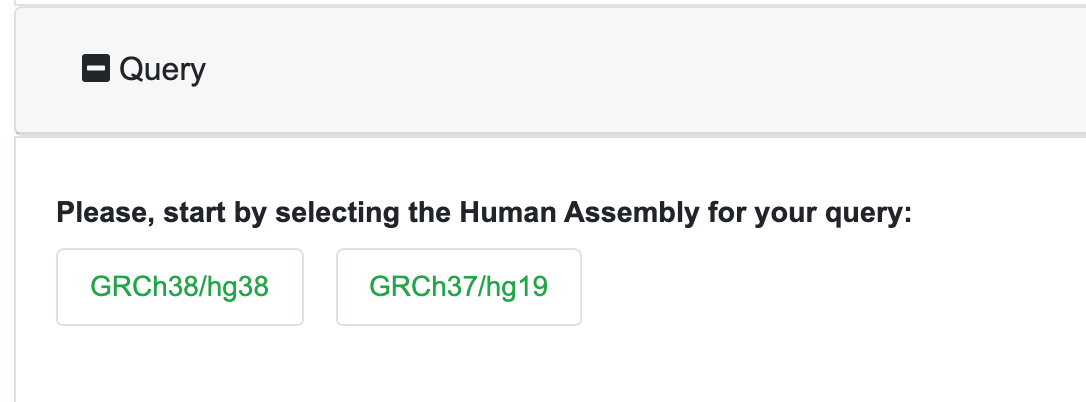
之后选择assembly的版本
选择后可以通过多种方式提交自己要查询的SNP
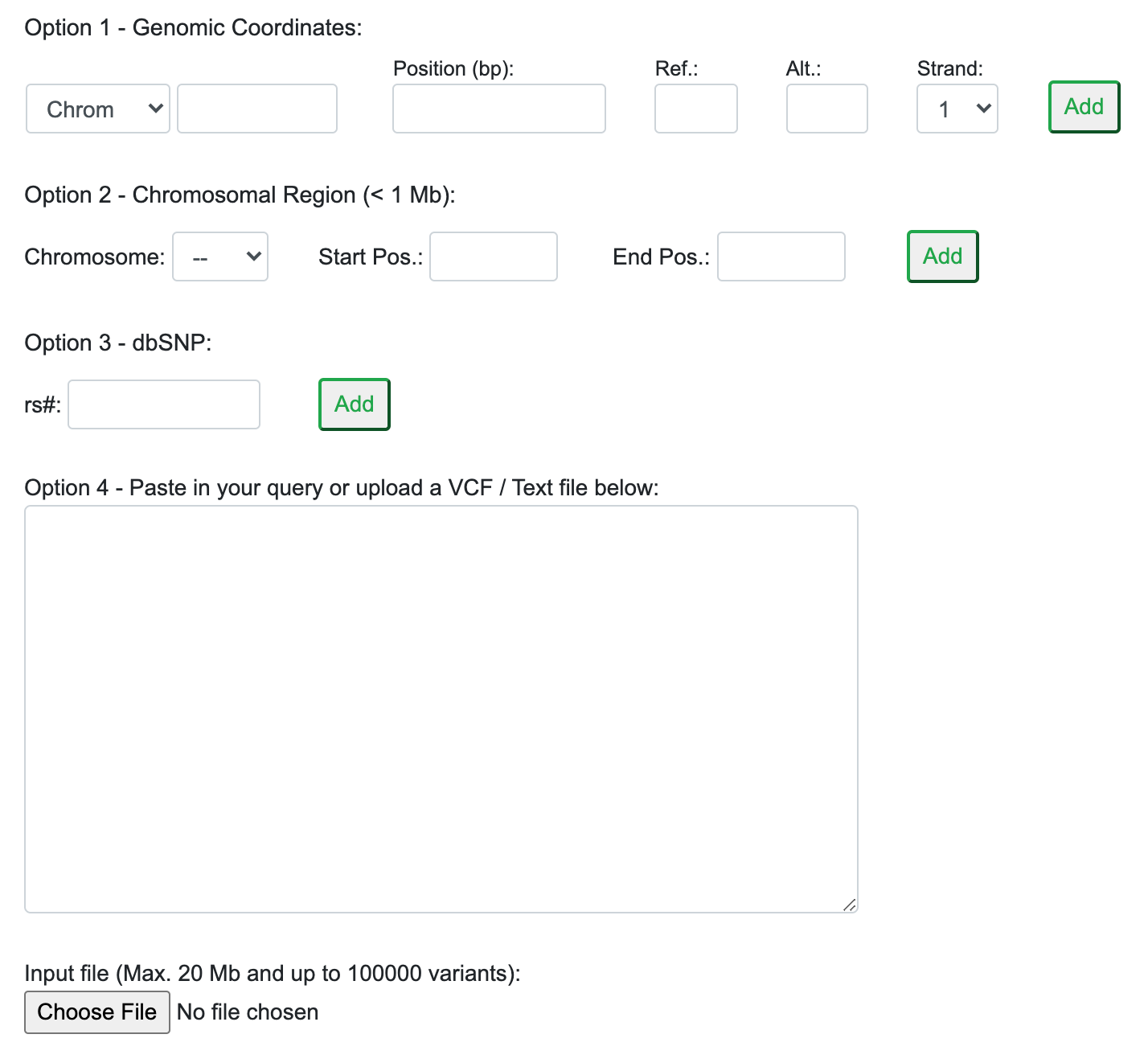
2.1.1 这里我们通过上传文件的方式对rsID进行注释
输入文件 snp.txt 格式如下 (其他格式的具体描述详见:SNP Annotation Tool - User Guide)
| 1 2 3 4 5 6 7 8 9 10 |
|
然后是注释用的数据选择,各数据库版本见:SNP Annotation Tool - User Guide
因为我们只查询位置信息,就不勾选其他数据库,只使用默认的Ensembl

点击submit query后,稍作等待,结果就会显示出来,可以导出为VCF或txt文本格式。

2.1.2 位点转换为rsID时,输入文件如下:
格式为:< Type Name Position Allele1 Allele2 Strand > # Genomic position data for novel SNPs
| 1 2 3 4 5 |
|
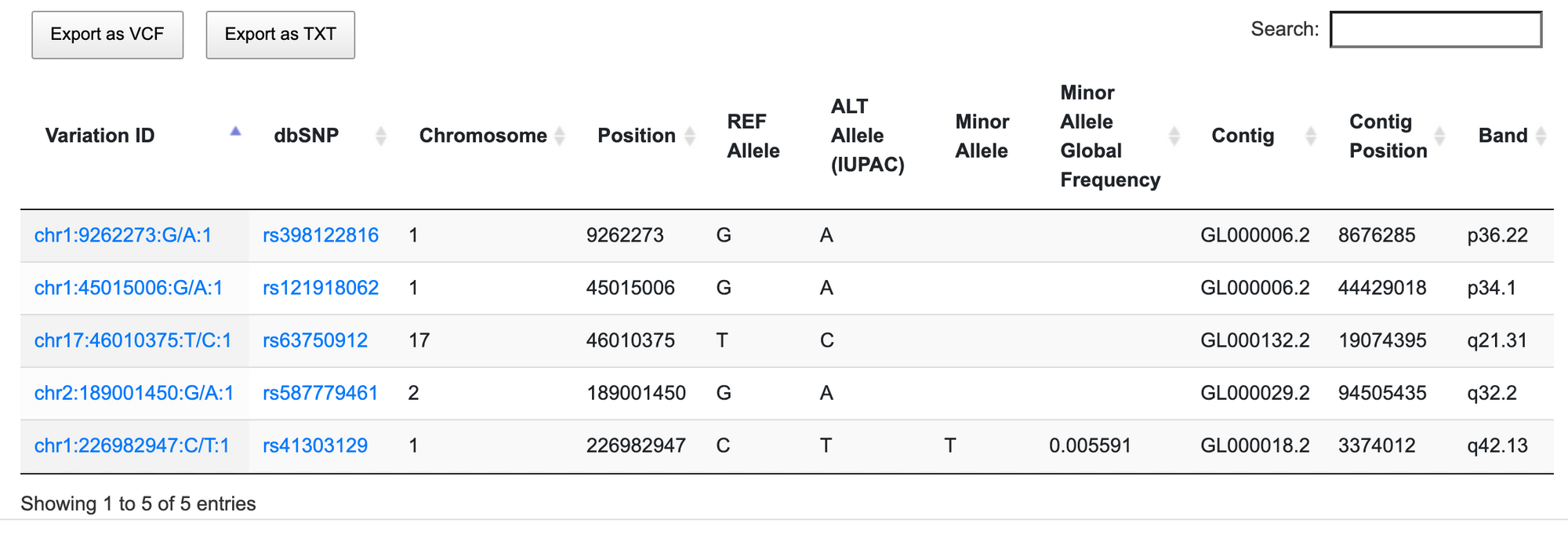
提交后即可查询,结果如下所示
2.2 VEP网页版
VEP注释工具使用详见(预留链接)
VEP网页版:VEP web interface
点击launch VEP

2.2.1 可以在input data处直接粘贴要查询的rsID,或是上传文件
点击输入框下方的example,可以查看可用的输入格式
这里以Variant identifiers 为例:
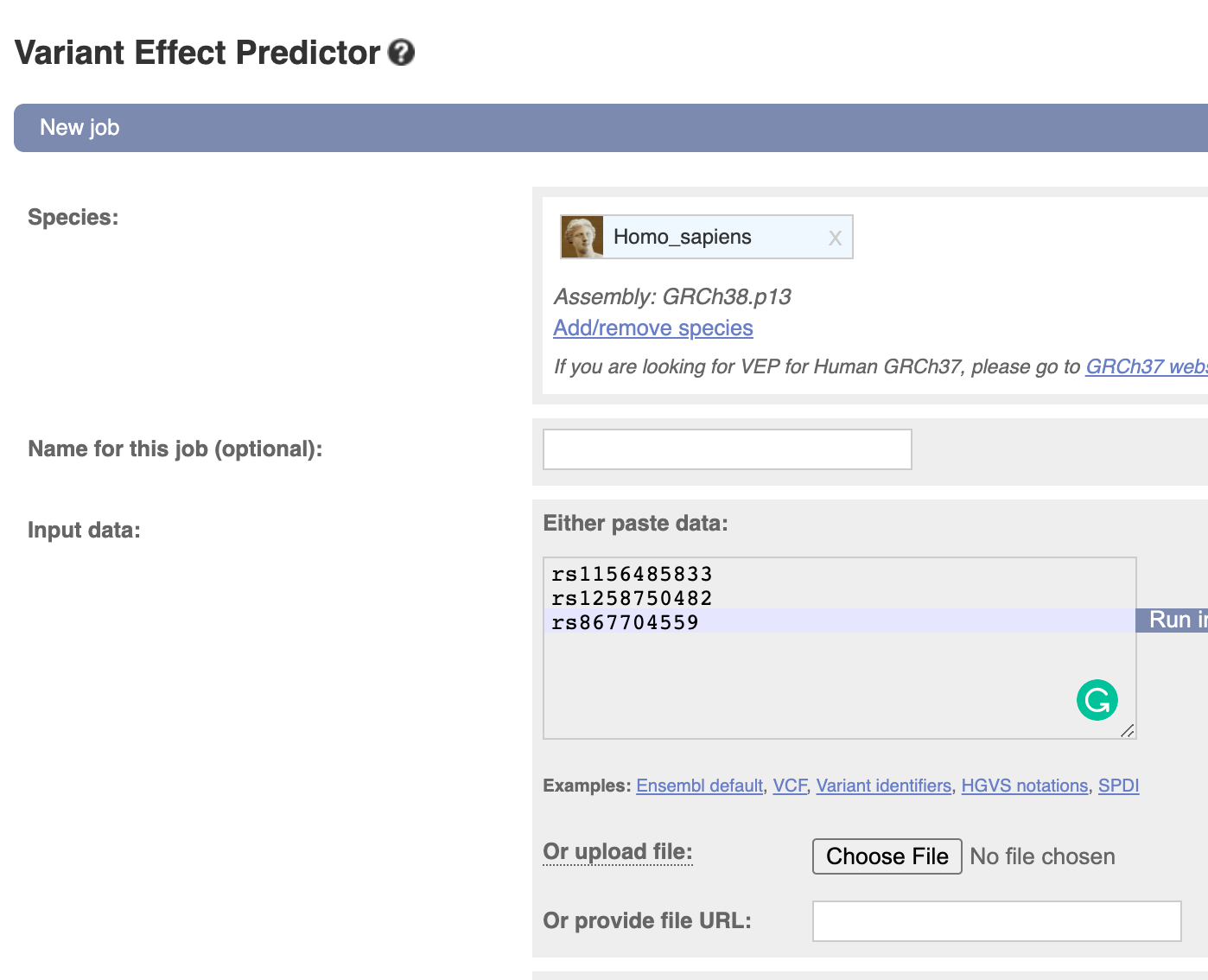
选择合适数据库,提交后可以看到查询状态

完成后点击View results

可查看基本信息,也可以导出
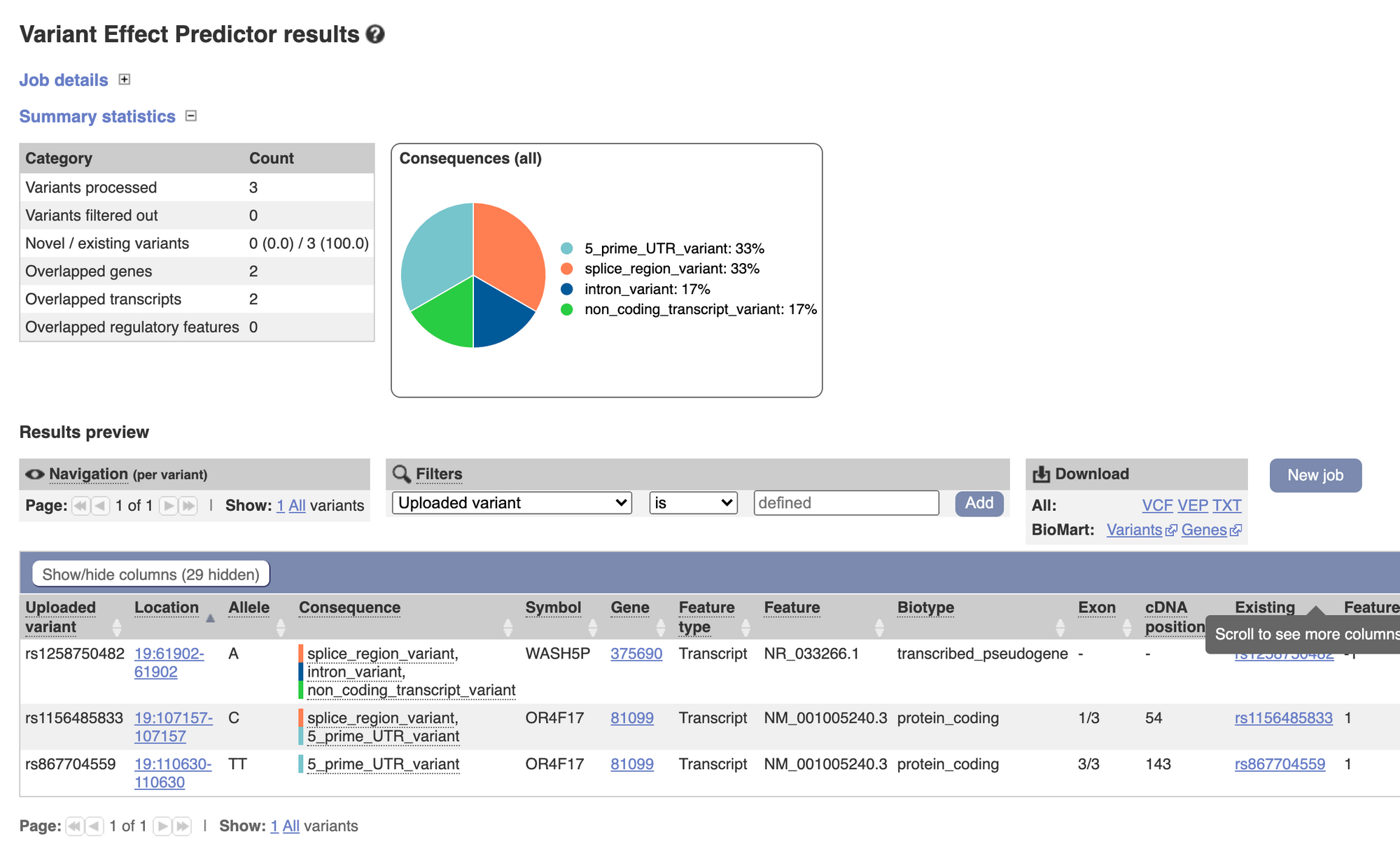
2.2.2 位点转换rsID时,使用如下Ensembl默认的输入格式即可:

3.大量转换时的命令行工具
3.1 可使用如上所述VEP工具的命令行版本
下载地址与文档见:Ensembl Variant Effect Predictor (VEP)
3.2 也可以使用ANNOVAR
安装与使用具体使用方法见:使用ANNOVAR 对Variants进行功能注释 Annotation POST-GWAS analysis
这里只简单介绍rsID与chr:pos互相转换的方法
3.2.1 rsID转chr:pos
使用ANNOVAR的convert2annovar.pl 程序与-format rsid选项即可注释位置信息,使用例如下所示,
(注意参考基因组的版本)
| 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 |
|
3.2.2 chr:pos转rsID
位点信息转化为rsID时需要使用基于筛选的注释:
这里使用avsnp150,具体描述可见:Download ANNOVAR - ANNOVAR Documentation

使用方法如下:
| 1 2 3 4 5 |
|
输入文件使用上述的(类bed文件),ex1.avinput
| 1 2 3 4 5 |
|
输出文件如下,ex1.avinput.hg19_avsnp150_dropped,
(注意某些SNP的rsID有合并等原因,版本不同rsID注释结果不一定相同)
| 1 2 3 4 5 |
|
注释rsID可能会遇到的问题:
Assigning dbSNP Identifiers - ANNOVAR Documentation
参考:
dbsnp:https://www.ncbi.nlm.nih.gov/snp/
annovar:ANNOVAR Documentation
vep:Ensembl Variant Effect Predictor (VEP)
snpnexus:SNP Annotation Tool


