- 1jmeter 压测 kafka_kafkajmeter.jar
- 2关于各种Stable diffusion模型,看这篇就可以了!_stable diffusion 模型
- 3城市级联选择优化:H5使用的vant,后管使用Element_使用vant省市区数据+element plus 实现省市区的选择
- 4MySQL 慢查询优化案例_mysql distinct 慢查询优化
- 5Qt/QML学习-动画元素
- 6OPenGL 学习笔记之 VAO VBO EBO 以及SHADER 并使用其绘制三角形_使用vao,vbo, ebo绘制一个三维图像
- 7第二十四记·Spark SQL配置及使用_如何配置spark sql
- 8java的JDK选择和在win11的安装与配置_win11 安装jdk
- 9Hadoop项目(一个类似于云盘的文件存储系统)_基于hadoop的项目
- 10python tokenize怎么用_tokenize --- 对 Python 代码使用的标记解析器 — Python 3.9.1 說明文件...
SciPy 1.12 中文文档(六十七)
赞
踩
scipy.stats.circstd
原文链接:
docs.scipy.org/doc/scipy-1.12.0/reference/generated/scipy.stats.circstd.html#scipy.stats.circstd
scipy.stats.circstd(samples, high=6.283185307179586, low=0, axis=None, nan_policy='propagate', *, normalize=False, keepdims=False)
- 1
计算假设样本在范围 [low, high] 中的圆形标准偏差。
参数:
samples:array_like
输入数组。
high:float 或 int,可选
样本范围的高边界。默认为 2*pi。
low:float 或 int,可选
样本范围的低边界。默认为 0。
normalize:boolean,可选
如果为 True,则返回的值等于 sqrt(-2*log(R)),并且不依赖于变量单位。如果为 False(默认),返回的值将按 ((high-low)/(2*pi)) 缩放。
axis:int 或 None,默认为 None
如果为整数,则计算统计量的输入轴(例如行)。输入的每个轴切片的统计量将出现在输出的相应元素中。如果为 None,则在计算统计量之前将进行展平。
nan_policy:{‘propagate’, ‘omit’, ‘raise’}
定义如何处理输入的 NaN。
-
propagate:如果在计算统计量的轴切片(例如行)中存在 NaN,则输出的对应条目将为 NaN。 -
omit:执行计算时将忽略 NaN。如果在计算统计量的轴切片中保留的数据不足,则输出的对应条目将为 NaN。 -
raise:如果存在 NaN,则会引发ValueError。
keepdims:bool,默认为 False
如果设置为 True,则减少的轴将作为大小为一的维度保留在结果中。使用此选项,结果将正确广播到输入数组。
返回:
circstd:float
圆形标准偏差。
另请参见
circmean
圆形均值。
circvar
圆形方差。
注意
这使用了来自[1]的圆形标准偏差的定义。本质上,计算如下。
import numpy as np
C = np.cos(samples).mean()
S = np.sin(samples).mean()
R = np.sqrt(C**2 + S**2)
l = 2*np.pi / (high-low)
circstd = np.sqrt(-2*np.log(R)) / l
- 1
- 2
- 3
- 4
- 5
- 6
在小角度极限下,它返回接近‘线性’标准偏差的数字。
从 SciPy 1.9 开始,np.matrix 输入(不推荐新代码使用)在执行计算之前将转换为 np.ndarray。在这种情况下,输出将是一个标量或适当形状的 np.ndarray,而不是 2D 的 np.matrix。类似地,虽然忽略掩码数组的掩码元素,但输出将是一个标量或 np.ndarray,而不是带有 mask=False 的掩码数组。
引用
[1]
Mardia, K. V. (1972). 2. 在 方向数据的统计 (pp. 18-24). Academic Press. DOI:10.1016/C2013-0-07425-7。
示例
>>> import numpy as np
>>> from scipy.stats import circstd
>>> import matplotlib.pyplot as plt
>>> samples_1 = np.array([0.072, -0.158, 0.077, 0.108, 0.286,
... 0.133, -0.473, -0.001, -0.348, 0.131])
>>> samples_2 = np.array([0.111, -0.879, 0.078, 0.733, 0.421,
... 0.104, -0.136, -0.867, 0.012, 0.105])
>>> circstd_1 = circstd(samples_1)
>>> circstd_2 = circstd(samples_2)
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
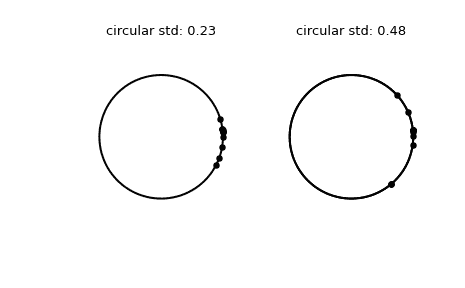
绘制样本。
>>> fig, (left, right) = plt.subplots(ncols=2)
>>> for image in (left, right):
... image.plot(np.cos(np.linspace(0, 2*np.pi, 500)),
... np.sin(np.linspace(0, 2*np.pi, 500)),
... c='k')
... image.axis('equal')
... image.axis('off')
>>> left.scatter(np.cos(samples_1), np.sin(samples_1), c='k', s=15)
>>> left.set_title(f"circular std: {np.round(circstd_1, 2)!r}")
>>> right.plot(np.cos(np.linspace(0, 2*np.pi, 500)),
... np.sin(np.linspace(0, 2*np.pi, 500)),
... c='k')
>>> right.scatter(np.cos(samples_2), np.sin(samples_2), c='k', s=15)
>>> right.set_title(f"circular std: {np.round(circstd_2, 2)!r}")
>>> plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
scipy.stats.sobol_indices
scipy.stats.sobol_indices(*, func, n, dists=None, method='saltelli_2010', random_state=None)
- 1
Sobol’的全局敏感性指数。
参数:
funccallable 或 dict(str, array_like)
如果func是可调用的,则用于计算 Sobol 指数的函数。其签名必须为:
func(x: ArrayLike) -> ArrayLike
- 1
具有形状(d, n)的x和形状(s, n)的输出,其中:
-
d是func的输入维度(输入变量数), -
s是func的输出维度(输出变量数), -
n是样本数量(见下文n)。
函数评估值必须是有限的。
如果func是字典,则包含来自三个不同数组的函数评估。键必须为:f_A、f_B和f_AB。f_A和f_B应该具有形状(s, n),而f_AB应该具有形状(d, s, n)。这是一个高级功能,滥用可能导致分析错误。
nint
用于生成矩阵A和B的样本数。必须是 2 的幂次方。对func进行评估的总点数将为n*(d+2)。
distslist(distributions),可选
每个参数的分布列表。参数的分布取决于应用程序,并应谨慎选择。假设参数是独立分布的,这意味着它们的值之间没有约束或关系。
分布必须是具有ppf方法的类的实例。
如果func是可调用的,则必须指定,否则将被忽略。
methodCallable 或 str,默认为‘saltelli_2010’
用于计算第一阶和总 Sobol 指数的方法。
如果是可调用的,则其签名必须是:
func(f_A: np.ndarray, f_B: np.ndarray, f_AB: np.ndarray)
-> Tuple[np.ndarray, np.ndarray]
- 1
- 2
具有形状(s, n)的f_A, f_B和形状(d, s, n)的f_AB。这些数组包含来自三组不同样本的函数评估。输出是形状为(s, d)的第一个和总索引的元组。这是一个高级功能,滥用可能导致分析错误。
random_state{None, int, numpy.random.Generator}, 可选
如果random_state是 int 或 None,则使用np.random.default_rng(random_state)创建一个新的numpy.random.Generator。如果random_state已经是Generator实例,则使用提供的实例。
返回:
resSobolResult
具有属性的对象:
形状为(s, d)的第一阶 Sobol 指数。
第一阶 Sobol 指数。
形状为(s, d)的总阶 Sobol 指数。
总阶 Sobol 指数。
以及方法:
bootstrap(confidence_level: float, n_resamples: int) -> BootstrapSobolResult
一种提供指数置信区间的方法。查看
scipy.stats.bootstrap获取更多详细信息。自助法是在一阶和总阶指数上进行的,并且它们作为属性
first_order和total_order存在于 BootstrapSobolResult 中。
注意
Sobol’ 方法 [1], [2] 是一种基于方差的敏感性分析,用于获取每个参数对感兴趣量(QoIs,即 func 的输出)方差的贡献。各自的贡献可以用来排列参数,并通过计算模型的有效(或平均)维度来评估模型的复杂性。
注意
假设参数是独立分布的。每个参数仍然可以遵循任何分布。事实上,分布非常重要,应该与参数的实际分布匹配。
它使用函数方差分解来探索
[\mathbb{V}(Y) = \sum_{i}^{d} \mathbb{V}i (Y) + \sum{i<j}^{d} \mathbb{V}{ij}(Y) + … + \mathbb{V}{1,2,…,d}(Y),]
引入条件方差:
[\mathbb{V}i(Y) = \mathbb{\mathbb{V}}[\mathbb{E}(Y|x_i)] \qquad \mathbb{V}{ij}(Y) = \mathbb{\mathbb{V}}[\mathbb{E}(Y|x_i x_j)] - \mathbb{V}_i(Y) - \mathbb{V}_j(Y),]
Sobol’ 指数表示为
[S_i = \frac{\mathbb{V}i(Y)}{\mathbb{V}[Y]} \qquad S{ij} =\frac{\mathbb{V}_{ij}(Y)}{\mathbb{V}[Y]}.]
(S_{i}) 对应于一阶项,评估第 i 个参数的贡献,而 (S_{ij}) 对应于二阶项,说明第 i 和第 j 个参数之间交互的贡献。这些方程可以推广到计算更高阶项;然而,它们的计算成本高昂,并且其解释较为复杂。这就是为什么只提供一阶指数的原因。
总阶指数代表了参数对 QoI 方差的全局贡献,定义如下:
[S_{T_i} = S_i + \sum_j S_{ij} + \sum_{j,k} S_{ijk} + … = 1 - \frac{\mathbb{V}[\mathbb{E}(Y|x_{\sim i})]}{\mathbb{V}[Y]}.]
一阶指数总和最多为 1,而总阶指数至少为 1。如果没有相互作用,则一阶和总阶指数相等,并且一阶和总阶指数总和为 1。
警告
负的 Sobol’ 值是由于数值误差造成的。增加点数 n 应该会有所帮助。
为了进行良好的分析,所需的样本数量随问题的维数增加而增加。例如,对于三维问题,考虑至少 n >= 2**12。模型越复杂,需要的样本就越多。
即使对于纯加法模型,由于数值噪声,指数的总和也可能不为 1。
参考文献
[1]
Sobol, I. M… “Sensitivity analysis for nonlinear mathematical models.” Mathematical Modeling and Computational Experiment, 1:407-414, 1993.
[2]
Sobol, I. M. (2001). “非线性数学模型的全局敏感性指数及其蒙特卡罗估计。”《数学与计算机仿真》,55(1-3):271-280,DOI:10.1016/S0378-4754(00)00270-6,2001.
[3]
Saltelli, A. “利用模型评估计算敏感性指数的最佳方法。”《计算物理通讯》,145(2):280-297,DOI:10.1016/S0010-4655(02)00280-1,2002.
[4]
Saltelli, A., M. Ratto, T. Andres, F. Campolongo, J. Cariboni, D. Gatelli, M. Saisana, 和 S. Tarantola. “全局敏感性分析入门。” 2007.
[5]
Saltelli, A., P. Annoni, I. Azzini, F. Campolongo, M. Ratto, 和 S. Tarantola. “基于方差的模型输出敏感性分析。总敏感性指数的设计和估计器。”《计算物理通讯》,181(2):259-270,DOI:10.1016/j.cpc.2009.09.018,2010.
[6]
Ishigami, T. 和 T. Homma. “计算机模型不确定性分析中的重要性量化技术。” IEEE,DOI:10.1109/ISUMA.1990.151285,1990。
Examples
以下是对 Ishigami 函数的一个例子 [6]
[Y(\mathbf{x}) = \sin x_1 + 7 \sin² x_2 + 0.1 x_3⁴ \sin x_1,]
其中 (\mathbf{x} \in [-\pi, \pi]³)。该函数表现出强非线性和非单调性。
请注意,Sobol’指数假设样本是独立分布的。在本例中,我们使用每个边缘上的均匀分布。
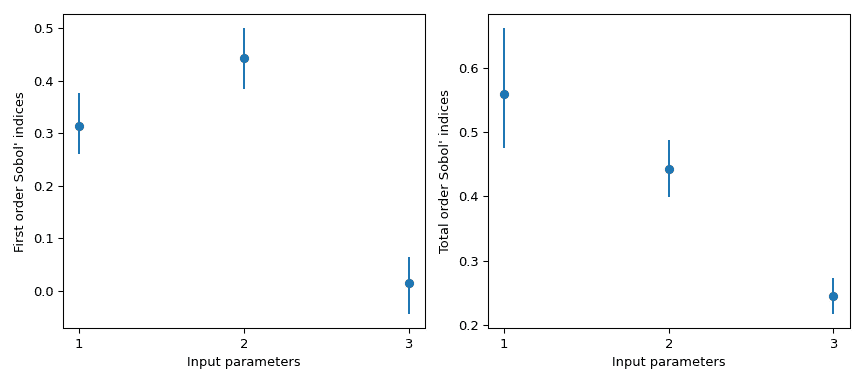
>>> import numpy as np >>> from scipy.stats import sobol_indices, uniform >>> rng = np.random.default_rng() >>> def f_ishigami(x): ... f_eval = ( ... np.sin(x[0]) ... + 7 * np.sin(x[1])**2 ... + 0.1 * (x[2]**4) * np.sin(x[0]) ... ) ... return f_eval >>> indices = sobol_indices( ... func=f_ishigami, n=1024, ... dists=[ ... uniform(loc=-np.pi, scale=2*np.pi), ... uniform(loc=-np.pi, scale=2*np.pi), ... uniform(loc=-np.pi, scale=2*np.pi) ... ], ... random_state=rng ... ) >>> indices.first_order array([0.31637954, 0.43781162, 0.00318825]) >>> indices.total_order array([0.56122127, 0.44287857, 0.24229595])
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
可以使用自举法获取置信区间。
>>> boot = indices.bootstrap()
- 1
然后,这些信息可以很容易地进行可视化。
>>> import matplotlib.pyplot as plt >>> fig, axs = plt.subplots(1, 2, figsize=(9, 4)) >>> _ = axs[0].errorbar( ... [1, 2, 3], indices.first_order, fmt='o', ... yerr=[ ... indices.first_order - boot.first_order.confidence_interval.low, ... boot.first_order.confidence_interval.high - indices.first_order ... ], ... ) >>> axs[0].set_ylabel("First order Sobol' indices") >>> axs[0].set_xlabel('Input parameters') >>> axs[0].set_xticks([1, 2, 3]) >>> _ = axs[1].errorbar( ... [1, 2, 3], indices.total_order, fmt='o', ... yerr=[ ... indices.total_order - boot.total_order.confidence_interval.low, ... boot.total_order.confidence_interval.high - indices.total_order ... ], ... ) >>> axs[1].set_ylabel("Total order Sobol' indices") >>> axs[1].set_xlabel('Input parameters') >>> axs[1].set_xticks([1, 2, 3]) >>> plt.tight_layout() >>> plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
Note
默认情况下,scipy.stats.uniform 的支持为 [0, 1]。通过参数 loc 和 scale,可以获得 [loc, loc + scale] 上的均匀分布。
这一结果尤为有趣,因为一阶指数 (S_{x_3} = 0),而其总体指数为 (S_{T_{x_3}} = 0.244)。这意味着与 (x_3) 的高阶交互作用导致了差异。几乎 25% 的观察方差是由 (x_3) 和 (x_1) 之间的相关性造成的,尽管 (x_3) 本身对 QoI 没有影响。
以下提供了关于该函数的 Sobol’指数的视觉解释。让我们在 ([-\pi, \pi]³) 中生成 1024 个样本,并计算输出的值。
>>> from scipy.stats import qmc
>>> n_dim = 3
>>> p_labels = ['$x_1$', '$x_2$', '$x_3$']
>>> sample = qmc.Sobol(d=n_dim, seed=rng).random(1024)
>>> sample = qmc.scale(
... sample=sample,
... l_bounds=[-np.pi, -np.pi, -np.pi],
... u_bounds=[np.pi, np.pi, np.pi]
... )
>>> output = f_ishigami(sample.T)
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
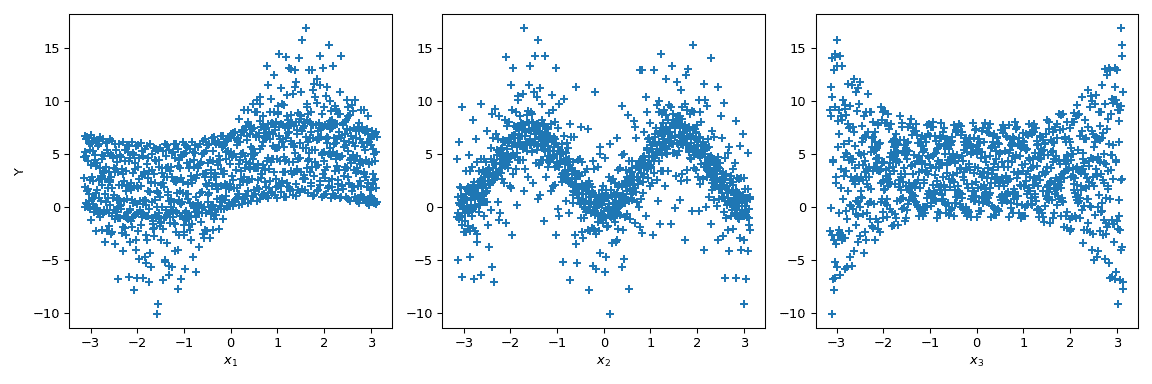
现在我们可以根据每个参数绘制输出的散点图。这提供了一种视觉方式来理解每个参数对函数输出的影响。
>>> fig, ax = plt.subplots(1, n_dim, figsize=(12, 4))
>>> for i in range(n_dim):
... xi = sample[:, i]
... ax[i].scatter(xi, output, marker='+')
... ax[i].set_xlabel(p_labels[i])
>>> ax[0].set_ylabel('Y')
>>> plt.tight_layout()
>>> plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
现在 Sobol’ 又进了一步:通过给定参数值(黑线),对输出值进行条件计算均值。这对应于术语 (\mathbb{E}(Y|x_i))。对这个术语的方差计算给出 Sobol’ 指数的分子。
>>> mini = np.min(output) >>> maxi = np.max(output) >>> n_bins = 10 >>> bins = np.linspace(-np.pi, np.pi, num=n_bins, endpoint=False) >>> dx = bins[1] - bins[0] >>> fig, ax = plt.subplots(1, n_dim, figsize=(12, 4)) >>> for i in range(n_dim): ... xi = sample[:, i] ... ax[i].scatter(xi, output, marker='+') ... ax[i].set_xlabel(p_labels[i]) ... for bin_ in bins: ... idx = np.where((bin_ <= xi) & (xi <= bin_ + dx)) ... xi_ = xi[idx] ... y_ = output[idx] ... ave_y_ = np.mean(y_) ... ax[i].plot([bin_ + dx/2] * 2, [mini, maxi], c='k') ... ax[i].scatter(bin_ + dx/2, ave_y_, c='r') >>> ax[0].set_ylabel('Y') >>> plt.tight_layout() >>> plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
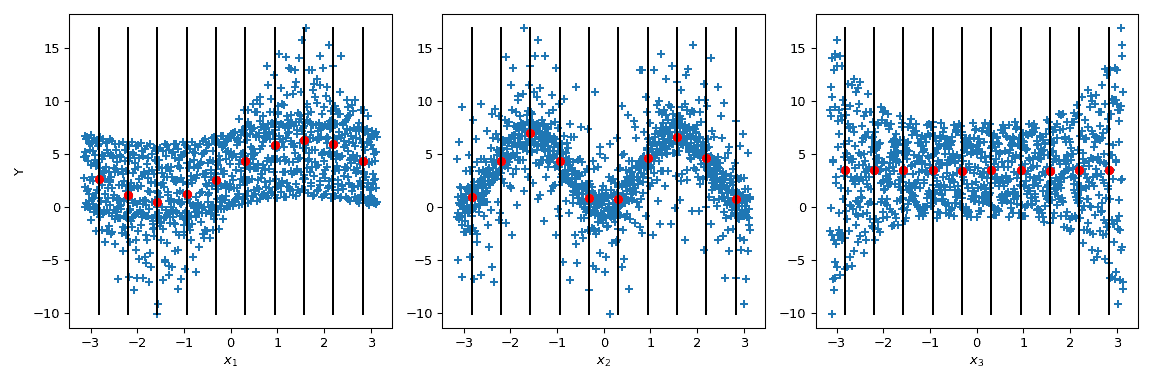
看看 (x_3),均值的方差为零,导致 (S_{x_3} = 0)。但我们可以进一步观察到输出的方差在 (x_3) 的参数值范围内并不是恒定的。这种异方差性可以通过更高阶的交互作用来解释。此外,在 (x_1) 上也能注意到异方差性,这表明 (x_3) 和 (x_1) 之间存在交互作用。在 (x_2) 上,方差似乎是恒定的,因此可以假设与这个参数的交互作用为零。
这种情况在视觉上分析起来相当简单——尽管这只是一种定性分析。然而,当输入参数的数量增加时,这种分析变得不现实,因为很难对高阶项进行结论。因此,使用 Sobol’ 指数的好处显而易见。
scipy.stats.ppcc_max
原文:
docs.scipy.org/doc/scipy-1.12.0/reference/generated/scipy.stats.ppcc_max.html#scipy.stats.ppcc_max
scipy.stats.ppcc_max(x, brack=(0.0, 1.0), dist='tukeylambda')
- 1
计算最大化 PPCC 的形状参数。
概率图相关系数(PPCC)图可用于确定单参数分布族的最佳形状参数。ppcc_max 返回使得给定数据到单参数分布族的概率图相关系数最大化的形状参数。
参数:
x 类似数组
输入数组。
brack 元组,可选
三元组(a,b,c),其中(a<b<c)。如果 bracket 包含两个数(a,c),则它们被假定为向下搜索的起始区间(参见 scipy.optimize.brent)。
dist 字符串或 stats.distributions 实例,可选
分布或分布函数名称。对象足够像一个 stats.distributions 实例(即它们有一个 ppf 方法)也被接受。默认为 'tukeylambda'。
返回:
shape_value 浮点数
使概率图相关系数达到其最大值的形状参数。
另请参阅
ppcc_plot, probplot, boxcox
注意
brack 关键字作为起始点,在极端情况下很有用。可以使用绘图来获取最大值位置的粗略视觉估计,以便在其附近开始搜索。
参考文献
[1]
J.J. Filliben,“用于正态性的概率图相关系数检验”,Technometrics,Vol. 17,pp. 111-117,1975 年。
[2]
工程统计手册,NIST/SEMATEC,www.itl.nist.gov/div898/handbook/eda/section3/ppccplot.htm
示例
首先,我们从形状参数为 2.5 的威布尔分布生成一些随机数据:
>>> import numpy as np
>>> from scipy import stats
>>> import matplotlib.pyplot as plt
>>> rng = np.random.default_rng()
>>> c = 2.5
>>> x = stats.weibull_min.rvs(c, scale=4, size=2000, random_state=rng)
- 1
- 2
- 3
- 4
- 5
- 6
为这些数据生成威布尔分布的 PPCC 图。
>>> fig, ax = plt.subplots(figsize=(8, 6))
>>> res = stats.ppcc_plot(x, c/2, 2*c, dist='weibull_min', plot=ax)
- 1
- 2
我们计算形状应达到其最大值的位置,并在那里画一条红线。该线应与 PPCC 图中的最高点重合。
>>> cmax = stats.ppcc_max(x, brack=(c/2, 2*c), dist='weibull_min')
>>> ax.axvline(cmax, color='r')
>>> plt.show()
- 1
- 2
- 3
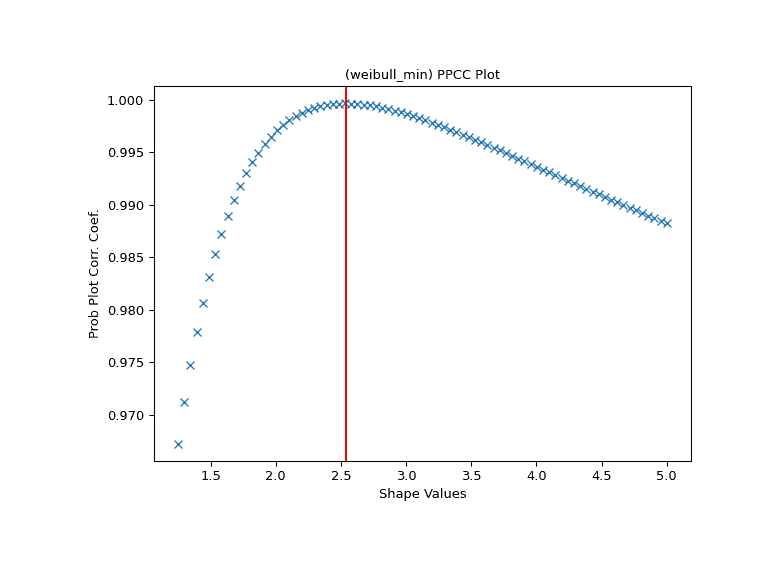
scipy.stats.ppcc_plot
原文链接:
docs.scipy.org/doc/scipy-1.12.0/reference/generated/scipy.stats.ppcc_plot.html#scipy.stats.ppcc_plot
scipy.stats.ppcc_plot(x, a, b, dist='tukeylambda', plot=None, N=80)
- 1
计算并可选地绘制概率图相关系数。
概率图相关系数(PPCC)图可用于确定单参数分布族的最佳形状参数。它不能用于没有形状参数(如正态分布)或具有多个形状参数的分布。
默认情况下使用 Tukey-Lambda 分布(stats.tukeylambda)。Tukey-Lambda PPCC 图通过近似正态分布从长尾到短尾分布进行插值,因此在实践中特别有用。
参数:
x array_like
输入数组。
a, b 标量
使用的形状参数的下限和上限。
dist str 或 stats.distributions 实例,可选
分布或分布函数名称。也接受足够像 stats.distributions 实例的对象(即它们有 ppf 方法)。默认值为 'tukeylambda'。
plot 对象,可选
如果给定,则绘制 PPCC 对形状参数的图。plot 是一个具有 “plot” 和 “text” 方法的对象。可以使用 matplotlib.pyplot 模块或 Matplotlib Axes 对象,或具有相同方法的自定义对象。默认值为 None,表示不创建图表。
N int,可选
水平轴上的点数(从 a 到 b 等距分布)。
返回:
svals ndarray
用于计算 ppcc 的形状值。
ppcc ndarray
计算的概率图相关系数值。
参见
ppcc_max,probplot,boxcox_normplot,tukeylambda
参考文献
J.J. Filliben,《正态性的概率图相关系数检验》,《Technometrics》,第 17 卷,第 111-117 页,1975 年。
示例
首先我们从形状参数为 2.5 的 Weibull 分布生成一些随机数据,并绘制数据的直方图:
>>> import numpy as np
>>> from scipy import stats
>>> import matplotlib.pyplot as plt
>>> rng = np.random.default_rng()
>>> c = 2.5
>>> x = stats.weibull_min.rvs(c, scale=4, size=2000, random_state=rng)
- 1
- 2
- 3
- 4
- 5
- 6
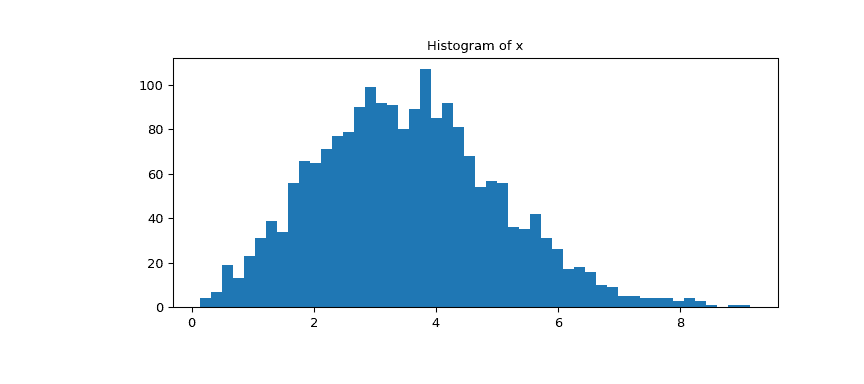
查看数据的直方图。
>>> fig1, ax = plt.subplots(figsize=(9, 4))
>>> ax.hist(x, bins=50)
>>> ax.set_title('Histogram of x')
>>> plt.show()
- 1
- 2
- 3
- 4
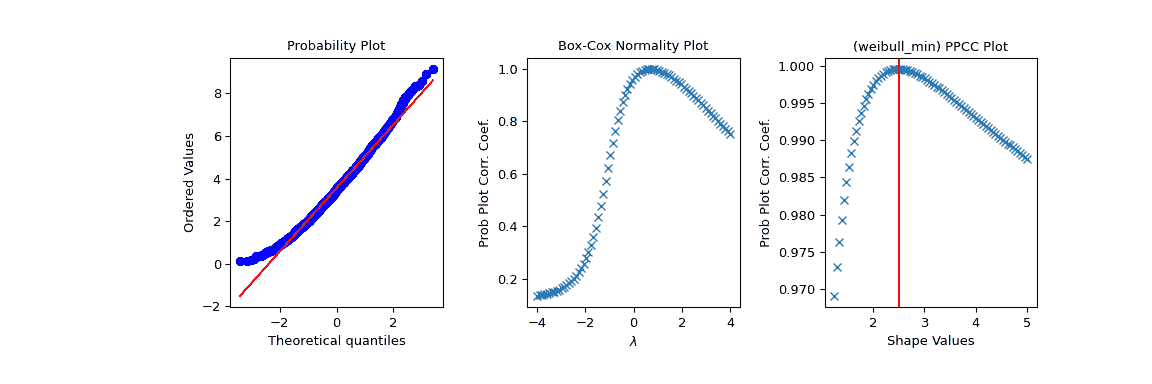
现在我们使用 PPCC 图及相关概率图和 Box-Cox normplot 探索这些数据。在我们预期 PPCC 值最大的形状参数 c 处绘制了一条红线:
>>> fig2 = plt.figure(figsize=(12, 4))
>>> ax1 = fig2.add_subplot(1, 3, 1)
>>> ax2 = fig2.add_subplot(1, 3, 2)
>>> ax3 = fig2.add_subplot(1, 3, 3)
>>> res = stats.probplot(x, plot=ax1)
>>> res = stats.boxcox_normplot(x, -4, 4, plot=ax2)
>>> res = stats.ppcc_plot(x, c/2, 2*c, dist='weibull_min', plot=ax3)
>>> ax3.axvline(c, color='r')
>>> plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
scipy.stats.probplot
原文链接:
docs.scipy.org/doc/scipy-1.12.0/reference/generated/scipy.stats.probplot.html#scipy.stats.probplot
scipy.stats.probplot(x, sparams=(), dist='norm', fit=True, plot=None, rvalue=False)
- 1
计算概率图的分位数,并可选择显示图。
生成样本数据的概率图,与指定理论分布的分位数(默认情况下为正态分布)进行比较。probplot 可选地计算数据的最佳拟合线,并使用 Matplotlib 或给定的绘图函数绘制结果。
参数:
x 类似数组
probplot 创建图的样本/响应数据。
sparams 元组,可选
特定于分布的形状参数(形状参数加上位置和尺度)。
dist 字符串或 stats.distributions 实例,可选
分布或分布函数名称。默认值为 ‘norm’,用于正态概率图。看起来足够像 stats.distributions 实例的对象(即它们具有 ppf 方法)也被接受。
fit 布尔值,可选
如果为 True(默认值),则对样本数据拟合最小二乘回归(最佳拟合)线。
plot 对象,可选
如果提供,则绘制分位数图。如果给定并且fit 为 True,则还绘制最小二乘拟合。plot 是一个必须具有“plot”和“text”方法的对象。可以使用 matplotlib.pyplot 模块或 Matplotlib Axes 对象,或具有相同方法的自定义对象。默认值为 None,表示不创建任何图。
rvalue 布尔值,可选
如果提供了 plot 并且fit 为 True,则将 rvalue 设置为 True 会在图中包含确定系数。默认值为 False。
返回:
(osm, osr) ndarrays 元组
具有理论分位数元组(osm,或顺序统计中位数)和有序响应(osr)的元组。osr 简单地是排序后的输入 x。有关如何计算 osm 的详细信息,请参阅注释部分。
(slope, intercept, r) 浮点数元组,可选
包含最小二乘拟合结果的元组,如果probplot 执行拟合。r 是确定系数的平方根。如果 fit=False 和 plot=None,则不返回此元组。
注释
即使提供了 plot,probplot 不会显示或保存图形;在调用 probplot 后应使用 plt.show() 或 plt.savefig('figname.png')。
probplot 生成一个概率图,不应与 Q-Q 图或 P-P 图混淆。Statsmodels 具有更广泛的类似功能,请参见 statsmodels.api.ProbPlot。
用于理论分位数(概率图的横轴)的公式是 Filliben 的估计:
quantiles = dist.ppf(val), for
0.5**(1/n), for i = n
val = (i - 0.3175) / (n + 0.365), for i = 2, ..., n-1
1 - 0.5**(1/n), for i = 1
- 1
- 2
- 3
- 4
- 5
其中 i 表示第 i 个排序值,n 是总值的数量。
例子
>>> import numpy as np
>>> from scipy import stats
>>> import matplotlib.pyplot as plt
>>> nsample = 100
>>> rng = np.random.default_rng()
- 1
- 2
- 3
- 4
- 5
自由度较小的 t 分布:
>>> ax1 = plt.subplot(221)
>>> x = stats.t.rvs(3, size=nsample, random_state=rng)
>>> res = stats.probplot(x, plot=plt)
- 1
- 2
- 3
自由度较大的 t 分布:
>>> ax2 = plt.subplot(222)
>>> x = stats.t.rvs(25, size=nsample, random_state=rng)
>>> res = stats.probplot(x, plot=plt)
- 1
- 2
- 3
两个正态分布的混合,使用广播:
>>> ax3 = plt.subplot(223)
>>> x = stats.norm.rvs(loc=[0,5], scale=[1,1.5],
... size=(nsample//2,2), random_state=rng).ravel()
>>> res = stats.probplot(x, plot=plt)
- 1
- 2
- 3
- 4
标准正态分布:
>>> ax4 = plt.subplot(224)
>>> x = stats.norm.rvs(loc=0, scale=1, size=nsample, random_state=rng)
>>> res = stats.probplot(x, plot=plt)
- 1
- 2
- 3
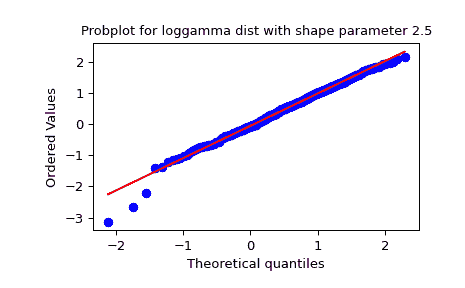
使用 dist 和 sparams 关键字生成一个 loggamma 分布的新图:
>>> fig = plt.figure()
>>> ax = fig.add_subplot(111)
>>> x = stats.loggamma.rvs(c=2.5, size=500, random_state=rng)
>>> res = stats.probplot(x, dist=stats.loggamma, sparams=(2.5,), plot=ax)
>>> ax.set_title("Probplot for loggamma dist with shape parameter 2.5")
- 1
- 2
- 3
- 4
- 5
用 Matplotlib 显示结果:
>>> plt.show()
- 1
scipy.stats.boxcox_normplot
scipy.stats.boxcox_normplot(x, la, lb, plot=None, N=80)
- 1
计算 Box-Cox 正态性图形的参数,可选择显示。
Box-Cox 正态性图形直观显示最佳转换参数,用于 boxcox 以获得接近正态分布的结果。
参数:
xarray_like
输入数组。
la, lbscalar
传递给 boxcox 的 lmbda 值的下限和上限。如果生成了绘图,这些值也是绘图的水平轴的限制。
plotobject, optional
如果给定,绘制分位数和最小二乘拟合。plot 是一个具有“plot”和“text”方法的对象。可以使用 matplotlib.pyplot 模块或 Matplotlib Axes 对象,或具有相同方法的自定义对象。默认为 None,即不创建绘图。
Nint, optional
水平轴上的点数(从 la 到 lb 等距分布)。
返回:
lmbdasndarray
进行 Box-Cox 变换的 lmbda 值。
ppccndarray
通过将 Box-Cox 变换的输入 x 拟合到正态分布时从 probplot 获取的概率图相关系数。
参见
probplot, boxcox, boxcox_normmax, boxcox_llf, ppcc_max
注意
即使给出 plot,调用 probplot 后,boxcox_normplot 不显示或保存图形;应使用 plt.show() 或 plt.savefig('figname.png')。
示例:
>>> from scipy import stats
>>> import matplotlib.pyplot as plt
- 1
- 2
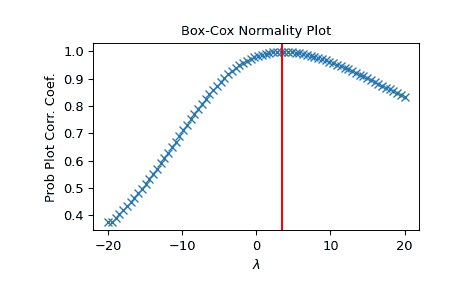
生成一些非正态分布的数据,并创建 Box-Cox 图形:
>>> x = stats.loggamma.rvs(5, size=500) + 5
>>> fig = plt.figure()
>>> ax = fig.add_subplot(111)
>>> prob = stats.boxcox_normplot(x, -20, 20, plot=ax)
- 1
- 2
- 3
- 4
确定并绘制最优 lmbda 以转换 x 并在同一图中绘制:
>>> _, maxlog = stats.boxcox(x)
>>> ax.axvline(maxlog, color='r')
- 1
- 2
>>> plt.show()
- 1
scipy.stats.yeojohnson_normplot
scipy.stats.yeojohnson_normplot(x, la, lb, plot=None, N=80)
- 1
计算 Yeo-Johnson 正态性图的参数,并可选择显示它。
Yeo-Johnson 正态性图以图形方式显示最佳变换参数,以便在 yeojohnson 中获得接近正态的分布。
参数:
xarray_like
输入数组。
la, lbscalar
传递给 yeojohnson 用于 Yeo-Johnson 变换的 lmbda 的下限和上限。如果生成了图形,则这也是图的水平轴的限制。
plotobject, optional
如果提供,则绘制分位数和最小二乘拟合。plot 是一个必须具有“plot”和“text”方法的对象。可以使用 matplotlib.pyplot 模块或 Matplotlib Axes 对象,或具有相同方法的自定义对象。默认为 None,表示不创建任何图。
Nint, optional
水平轴上的点数(从 la 到 lb 等距分布)。
返回:
lmbdasndarray
进行 Yeo-Johnson 变换的 lmbda 值。
ppccndarray
概率图相关系数,从 probplot 中获取,用于将 Box-Cox 变换后的输入 x 拟合到正态分布。
参见
probplot, yeojohnson, yeojohnson_normmax, yeojohnson_llf, ppcc_max
注意事项
即使给定 plot,调用 boxcox_normplot 后,图形也不会显示或保存;应在调用 probplot 后使用 plt.show() 或 plt.savefig('figname.png')。
版本 1.2.0 中的新功能。
示例
>>> from scipy import stats
>>> import matplotlib.pyplot as plt
- 1
- 2
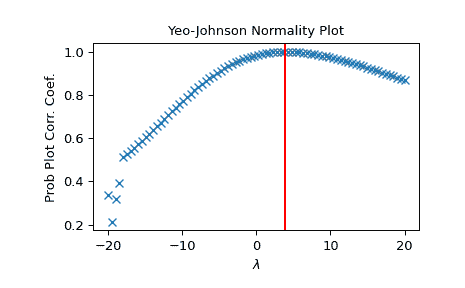
生成一些非正态分布的数据,并创建一个 Yeo-Johnson 图:
>>> x = stats.loggamma.rvs(5, size=500) + 5
>>> fig = plt.figure()
>>> ax = fig.add_subplot(111)
>>> prob = stats.yeojohnson_normplot(x, -20, 20, plot=ax)
- 1
- 2
- 3
- 4
确定并绘制最佳 lmbda,将 x 转换并在同一图中绘制:
>>> _, maxlog = stats.yeojohnson(x)
>>> ax.axvline(maxlog, color='r')
- 1
- 2
>>> plt.show()
- 1
scipy.stats.gaussian_kde
class scipy.stats.gaussian_kde(dataset, bw_method=None, weights=None)
- 1
使用高斯核进行核密度估计的表示。
核密度估计是以非参数化方式估计随机变量的概率密度函数(PDF)的一种方法。 gaussian_kde 适用于单变量和多变量数据。 它包括自动带宽确定。 该估计对于单峰分布效果最佳; 双峰或多峰分布往往会过度平滑。
参数:
数据集array_like
用于估计的数据点。 在单变量数据的情况下,这是一个 1-D 数组,否则是一个形状为(# dims,# data)的 2-D 数组。
bw_methodstr,标量或可调用对象,可选
用于计算估计器带宽的方法。 这可以是“scott”,“silverman”,标量常数或可调用对象。 如果是标量,则会直接用作 kde.factor。 如果是可调用对象,则应该只接受一个 gaussian_kde 实例作为参数并返回一个标量。 如果为 None(默认值),则使用“scott”。 有关详细信息,请参阅注释。
权重array_like,可选
数据点的权重。 这必须与数据集具有相同的形状。 如果为 None(默认),则假定样本的权重相等。
注意事项
带宽选择强烈影响从 KDE 得到的估计(远远超过核的实际形状)。 带宽选择可以通过“经验法”,交叉验证, “插件方法”或其他方式来完成; 参见 [3],[4] 进行评论。 gaussian_kde 使用经验法,默认为斯科特法则。
斯科特法则 [1],实现为scotts_factor,是:
n**(-1./(d+4)),
- 1
与 n 为数据点数,d 为维数。 在数据点权重不均匀的情况下,scotts_factor 变为:
neff**(-1./(d+4)),
- 1
与 neff 为有效数据点数。 Silverman 法则 [2],实现为silverman_factor,是:
(n * (d + 2) / 4.)**(-1. / (d + 4)).
- 1
或在数据点权重不均匀的情况下:
(neff * (d + 2) / 4.)**(-1. / (d + 4)).
- 1
可以在 [1] 和 [2] 找到关于这个多维实现的数学描述。
对一组加权样本,有效数据点数量neff的定义为:
neff = sum(weights)² / sum(weights²)
- 1
如[5]所详述。
gaussian_kde当前不支持数据位于其表达空间的低维子空间中。对于这样的数据,考虑执行主成分分析/降维,并使用gaussian_kde处理转换后的数据。
参考文献
[1] (1,2,3)
D.W. Scott, “多元密度估计:理论、实践与可视化”, John Wiley & Sons, New York, Chicester, 1992.
[2] (1,2)
B.W. Silverman, “统计学与数据分析中的密度估计”, Vol. 26, 统计学与应用概率论丛书, Chapman and Hall, London, 1986.
[3]
B.A. Turlach, “Kernel Density Estimation 中的带宽选择:一项回顾”, CORE 和 Institut de Statistique, Vol. 19, pp. 1-33, 1993.
[4]
D.M. Bashtannyk 和 R.J. Hyndman, “用于核条件密度估计的带宽选择”, Computational Statistics & Data Analysis, Vol. 36, pp. 279-298, 2001.
[5]
Gray P. G., 1969, Journal of the Royal Statistical Society. Series A (General), 132, 272
示例
生成一些随机的二维数据:
>>> import numpy as np
>>> from scipy import stats
>>> def measure(n):
... "Measurement model, return two coupled measurements."
... m1 = np.random.normal(size=n)
... m2 = np.random.normal(scale=0.5, size=n)
... return m1+m2, m1-m2
- 1
- 2
- 3
- 4
- 5
- 6
- 7
>>> m1, m2 = measure(2000)
>>> xmin = m1.min()
>>> xmax = m1.max()
>>> ymin = m2.min()
>>> ymax = m2.max()
- 1
- 2
- 3
- 4
- 5
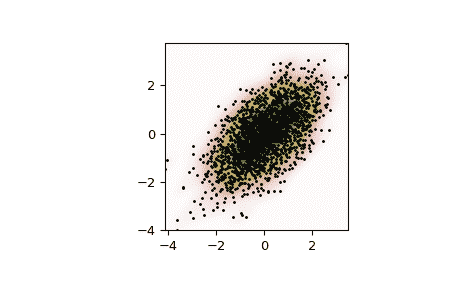
对数据执行核密度估计:
>>> X, Y = np.mgrid[xmin:xmax:100j, ymin:ymax:100j]
>>> positions = np.vstack([X.ravel(), Y.ravel()])
>>> values = np.vstack([m1, m2])
>>> kernel = stats.gaussian_kde(values)
>>> Z = np.reshape(kernel(positions).T, X.shape)
- 1
- 2
- 3
- 4
- 5
绘制结果:
>>> import matplotlib.pyplot as plt
>>> fig, ax = plt.subplots()
>>> ax.imshow(np.rot90(Z), cmap=plt.cm.gist_earth_r,
... extent=[xmin, xmax, ymin, ymax])
>>> ax.plot(m1, m2, 'k.', markersize=2)
>>> ax.set_xlim([xmin, xmax])
>>> ax.set_ylim([ymin, ymax])
>>> plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
属性:
datasetndarray
用于初始化gaussian_kde的数据集。
dint
维度的数量。
nint
数据点的数量。
neffint
有效数据点的数量。
1.2.0 版本中的新功能。
factorfloat
从kde.covariance_factor获取的带宽因子。kde.factor的平方乘以数据的协方差矩阵进行 kde 估计。
covariancendarray
dataset的协方差矩阵,按计算得到的带宽(kde.factor)进行缩放。
inv_covndarray
covariance的逆矩阵。
方法
evaluate(points) | 对一组点评估估计的概率密度函数。 |
|---|---|
__call__(points) | 对一组点评估估计的概率密度函数。 |
integrate_gaussian(mean, cov) | 通过多变量高斯函数乘以估计的密度并在整个空间上积分。 |
integrate_box_1d(low, high) | 计算 1D 概率密度函数在两个边界之间的积分。 |
integrate_box(low_bounds, high_bounds[, maxpts]) | 计算 pdf 在矩形区间上的积分。 |
integrate_kde(other) | 计算该核密度估计与另一个核密度估计的乘积的积分。 |
pdf(x) | 在提供的点集上评估估计的 pdf。 |
logpdf(x) | 在提供的点集上评估估计的 pdf 的对数。 |
resample([size, seed]) | 从估计的 pdf 中随机抽样数据集。 |
set_bandwidth([bw_method]) | 使用给定方法计算估计器带宽。 |
covariance_factor() | 计算乘以数据协方差矩阵以获得核协方差矩阵的系数 (kde.factor)。 |
scipy.stats.DegenerateDataWarning
exception scipy.stats.DegenerateDataWarning(msg=None)
- 1
在数据退化且结果可能不可靠时发出警告。
with_traceback()
- 1
Exception.with_traceback(tb) – 将 self.traceback 设置为 tb 并返回 self。
scipy.stats.ConstantInputWarning
exception scipy.stats.ConstantInputWarning(msg=None)
- 1
当数据中所有值完全相等时发出警告。
with_traceback()
- 1
Exception.with_traceback(tb) – set self.traceback to tb and return self.
scipy.stats.NearConstantInputWarning
exception scipy.stats.NearConstantInputWarning(msg=None)
- 1
在数据中所有值几乎相等时发出警告。
with_traceback()
- 1
Exception.with_traceback(tb) – 设置 self.traceback 为 tb 并返回 self。
scipy.stats.FitError
原文:
docs.scipy.org/doc/scipy-1.12.0/reference/generated/scipy.stats.FitError.html#scipy.stats.FitError
exception scipy.stats.FitError(msg=None)
- 1
表示拟合分布到数据时的错误条件。
with_traceback()
- 1
Exception.with_traceback(tb) – 将 self.traceback 设置为 tb 并返回 self。
从源码构建
注意
如果你只想安装 SciPy,我们建议使用二进制文件 - 详细信息请参见安装。
从源码构建 SciPy 需要先设置系统级依赖项(编译器、BLAS/LAPACK 库等),然后调用构建命令。构建可以用来在本地安装 SciPy、开发 SciPy 本身或构建可重新分发的二进制包。可能希望定制构建方式的各个方面。本指南将涵盖所有这些方面。此外,它还提供了有关 SciPy 构建工作方式的背景信息,并链接到通用 Python 构建和打包文档的最新指南,这些内容是相关的。
系统级别依赖
SciPy 使用编译代码以提升速度,这意味着你需要安装编译器和一些其他系统级别(即非 Python/非 PyPI)的依赖项来在你的系统上构建它。
注意
如果你使用的是 Conda,你可以跳过本节中的步骤 - 除了在 Windows 上安装编译器或在 macOS 上安装 Apple 开发者工具之外。mamba env create -f environment.yml命令将自动安装所有其他依赖项。
如果你想使用系统自带的 Python 和pip,你需要:
-
C、C++和 Fortran 编译器(通常是
gcc、g++和gfortran)。 -
Python 头文件(通常是名为
python3-dev或python3-devel的包) -
用于依赖检测的
pkg-config。
要安装 SciPy 的构建要求,可以执行:
sudo apt install -y gcc g++ gfortran libopenblas-dev liblapack-dev pkg-config python3-pip python3-dev
- 1
或者,你可以执行:
sudo apt build-dep scipy
- 1
此命令安装构建 SciPy 所需的一切,其优势在于包管理器处理新的依赖项或更新所需版本。
要安装 SciPy 的构建要求,可以执行:
sudo dnf install gcc-gfortran python3-devel openblas-devel lapack-devel pkgconfig
- 1
或者,你可以执行:
sudo dnf builddep scipy
- 1
此命令安装构建 SciPy 所需的一切,其优势在于包管理器处理新的依赖项或更新所需版本。
要安装 SciPy 的构建要求,可以执行:
sudo yum install gcc-gfortran python3-devel openblas-devel lapack-devel pkgconfig
- 1
或者,你可以执行:
sudo yum-builddep scipy
- 1
此命令安装构建 SciPy 所需的一切,其优势在于包管理器处理新的依赖项或更新所需版本。
要安装 SciPy 的构建要求,可以执行:
sudo pacman -S gcc-fortran openblas pkgconf
- 1
安装 Apple 开发者工具。一个简单的方法是打开终端窗口,输入以下命令:
xcode-select --install
- 1
然后按照提示进行操作。Apple 开发者工具包括 Git、Clang C/C++编译器和其他可能需要的开发工具。
不要使用 macOS 系统自带的 Python。建议使用python.org 安装程序或 Homebrew、MacPorts 或 Fink 等软件包管理器来安装 Python。
您还需要安装 Fortran 编译器、BLAS 和 LAPACK 库以及 pkg-config 这些其他系统依赖项。建议使用Homebrew来安装这些依赖项:
brew install gfortran openblas pkg-config
- 1
注意
从 SciPy >=1.2.0 开始,我们不支持使用系统自带的 Accelerate 库来编译 BLAS 和 LAPACK。它不支持足够新的 LAPACK 接口。计划在 2023 年进行更改,因为 macOS 13.3 引入了对 Accelerate 的重大升级,解决了所有已知问题。
在 Windows 上构建 SciPy 需要一套兼容的 C、C++和 Fortran 编译器。与其他平台相比,这在 Windows 上要复杂一些,因为 MSVC 不支持 Fortran,而 gfortran 和 MSVC 不能一起使用。您需要选择其中一组编译器:
-
MinGW-w64 编译器(
gcc、g++、gfortran) - 推荐,因为最容易安装,并且是 SciPy 自己 CI 和二进制文件使用的工具 -
MSVC + Intel Fortran(
ifort) -
Intel 编译器(
icc、ifort)
相较于 macOS 和 Linux,在 Windows 上构建 SciPy 稍显复杂,因为需要设置这些编译器。不像在其他平台上可以直接在命令提示符中运行单行命令。
首先安装 Microsoft Visual Studio - 建议使用 2019 社区版或更新版本(参见Visual Studio 下载站点)。即使使用 MinGW-w64 或 Intel 编译器,也需要安装以确保具有 Windows 通用 C 运行时(在使用 Mingw-w64 时不需要 Visual Studio 的其他组件,如果需要可以取消选择以节省磁盘空间)。
有几个 MinGW-w64 二进制发行版。我们推荐使用 RTools 版本,可以通过 Chocolatey(参见这里的 Chocolatey 安装说明)安装:
choco install rtools -y --no-progress --force --version=4.0.0.20220206
- 1
如遇问题,建议使用与SciPy GitHub Actions CI jobs for Windows中使用的确切版本相同。
MSVC 安装程序不会将编译器添加到系统路径中,并且安装位置可能会更改。要查询安装位置,MSVC 附带有一个vswhere.exe命令行实用工具。为了在您使用的 shell 中使 C/C++编译器可用,您需要运行一个适合位数和架构的.bat文件(例如,对于 64 位 Intel CPU,请使用vcvars64.bat)。
欲获得详细指导,请参阅从命令行使用 Microsoft C++工具集。
与 MSVC 类似,Intel 编译器设计为与您正在使用的 shell 中的激活脚本 (Intel\oneAPI\setvars.bat) 一起使用。这使得编译器可以在路径中使用。有关详细指导,请参阅 Intel® oneAPI HPC Toolkit for Windows 入门指南。
注意
编译器应该在系统路径上(即 PATH 环境变量应包含可以找到编译器可执行文件的目录)才能被找到,MSVC 是个例外,如果 PATH 上没有其他编译器,它会自动找到。您可以使用任何 shell(例如 Powershell,cmd 或 Git Bash)来调用构建。要检查是否符合条件,请在您使用的 shell 中尝试调用 Fortran 编译器(例如 gfortran --version 或 ifort --version)。
警告
在使用 conda 环境时,由于过时的 Fortran 编译器可能会导致环境创建失败。如果发生这种情况,请从 environment.yml 中删除 compilers 条目,然后重试。Fortran 编译器应按照本节描述的方式安装。
从源码构建 SciPy
如果您只想从源码安装 SciPy 一次而不进行任何开发工作,则建议使用 pip 来构建和安装。否则,推荐使用 conda。
注意
如果您尚未安装 conda,我们建议使用 Mambaforge;任何 conda 都可以使用。
从源码构建以使用 SciPy
如果您使用的是 conda 环境,pip 仍然是调用 SciPy 源码构建的工具。重要的是始终使用 --no-build-isolation 标志来执行 pip install 命令,以避免针对 PyPI 上的 numpy 轮构建。为了使其工作,您必须先将其余构建依赖项安装到 conda 环境中:
# Either install all SciPy dev dependencies into a fresh conda environment
mamba env create -f environment.yml
# Or, install only the required build dependencies
mamba install python numpy cython pythran pybind11 compilers openblas meson-python pkg-config
# To build the latest stable release:
pip install scipy --no-build-isolation --no-binary scipy
# To build a development version, you need a local clone of the SciPy git repository:
git clone https://github.com/scipy/scipy.git
cd scipy
git submodule update --init
pip install . --no-build-isolation
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
# To build the latest stable release:
pip install scipy --no-binary scipy
# To build a development version, you need a local clone of the SciPy git repository:
git clone https://github.com/scipy/scipy.git
cd scipy
git submodule update --init
pip install .
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
用于 SciPy 开发的源码构建
如果您希望从源码构建以便于在 SciPy 自身上工作,请首先克隆 SciPy 存储库:
git clone https://github.com/scipy/scipy.git
cd scipy
git submodule update --init
- 1
- 2
- 3
然后,您需要执行以下操作:
-
创建一个专用的开发环境(虚拟环境或 conda 环境),
-
安装所有需要的依赖项(构建,以及 测试, 文档 和 可选 依赖项),
-
使用我们的
dev.py开发者接口构建 SciPy。
步骤(3)始终相同,步骤(1)和(2)在 conda 和虚拟环境之间有所不同:
要创建一个 scipy-dev 开发环境并安装每个必需和可选的依赖项,请运行:
mamba env create -f environment.yml
mamba activate scipy-dev
- 1
- 2
注意
有许多工具可以管理虚拟环境,如 venv,virtualenv/virtualenvwrapper,pyenv/pyenv-virtualenv,Poetry,PDM,Hatch 等。这里我们使用 Python 标准库中的基本工具 venv。您可以使用任何其他工具;我们只需要一个已激活的 Python 环境即可。
在名为 venv 的新目录中创建并激活虚拟环境(请注意,激活命令可能因您的操作系统和 Shell 而异,请参阅“venv 工作原理”中的 venv 文档)。
python -m venv venv
source venv/bin/activate
- 1
- 2
python -m venv venv
source venv/bin/activate
- 1
- 2
python -m venv venv
.\venv\Scripts\activate
- 1
- 2
然后使用以下命令从 PyPI 安装 Python 级依赖项(参见 pyproject.toml):
# Build dependencies
python -m pip install numpy cython pythran pybind11 meson-python ninja pydevtool rich-click
# Test and optional runtime dependencies
python -m pip install pytest pytest-xdist pytest-timeout pooch threadpoolctl asv gmpy2 mpmath hypothesis
# Doc build dependencies
python -m pip install sphinx "pydata-sphinx-theme==0.9.0" sphinx-design matplotlib numpydoc jupytext myst-nb
# Dev dependencies (static typing and linting)
python -m pip install mypy typing_extensions types-psutil pycodestyle ruff cython-lint
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
要在已激活的开发环境中构建 SciPy,请运行:
python dev.py build
- 1
这将在存储库内安装 SciPy(默认情况下在 build-install 目录中)。然后您可以运行测试(python dev.py test),进入 IPython(python dev.py ipython),或者进行其他开发步骤,如构建 HTML 文档或运行基准测试。dev.py 接口具有自我文档功能,请参阅 python dev.py --help 和 python dev.py <subcommand> --help 以获取详细指导。
IDE 支持和可编辑安装
虽然 dev.py 接口是我们在 SciPy 上推荐的工作方式,但它有一个限制:由于自定义安装位置的原因,使用 dev.py 安装的 SciPy 将不会在 IDE 中被自动识别(例如,通过“运行”按钮运行脚本或者通过视觉设置断点)。这在 就地构建(或“可编辑安装”)中可以更好地工作。
支持可编辑安装。重要的是要理解 在给定的存储库克隆中,您可以使用可编辑安装或 dev.py,但不能两者都用。如果使用可编辑安装,您必须直接使用 pytest 和其他开发工具,而不是使用 dev.py。
若要使用可编辑安装,请确保从干净的存储库开始(如果之前使用过 dev.py,请运行 git clean -xdf),并且如本页面上方描述的那样正确设置所有依赖项。然后执行:
# Note: the --no-build-isolation is important! meson-python will
# auto-rebuild each time SciPy is imported by the Python interpreter.
pip install -e . --no-build-isolation
# To run the tests for, e.g., the `scipy.linalg` module:
pytest scipy/linalg
- 1
- 2
- 3
- 4
- 5
- 6
对 SciPy 代码进行更改时,包括编译代码,无需手动重新构建或重新安装。当您运行 git clean -xdf 时,它会删除构建的扩展模块,同时记住也要使用 pip uninstall scipy 卸载 SciPy。
请参阅 meson-python 文档中有关可编辑安装更多细节的介绍。
自定义构建
-
选择编译器和自定义构建
-
BLAS 和 LAPACK
-
交叉编译
-
构建可重分发的二进制文件
背景信息
-
理解 Meson
-
内省构建步骤
-
Meson 和
distutils的工作方式
编译器选择和定制构建
原文链接:
docs.scipy.org/doc/scipy-1.12.0/building/compilers_and_options.html
选择特定的编译器
Meson 支持标准的环境变量 CC, CXX 和 FC 来选择特定的 C、C++ 和/或 Fortran 编译器。这些环境变量在Meson 文档中的参考表中有详细说明。
请注意,环境变量仅在清理构建时才会被应用,因为它们会影响配置阶段(即 meson setup)。增量重建不会对环境变量的更改做出反应 - 您必须运行 git clean -xdf 并进行完整重建,或者运行 meson setup --reconfigure。
添加自定义编译器或链接器标志
Meson 的设计偏向通过传递给 meson setup 的命令行选项配置构建。它提供了许多内置选项:
-
要启用调试构建和优化级别,请参见下一节关于“构建类型”的内容,
-
以便以便携带的方式启用
-Werror,可以通过-Dwerror=true完成, -
启用警告级别的方法是通过
-Dwarning_level=<val>完成,其中<val>可选值为{0, 1, 2, 3, everything}, -
还有许多其他内置选项,例如激活 Visual Studio (
-Dvsenv=true) 和使用链接时优化 (-Db_lto),或者更改默认的 C++ 语言级别 (-Dcpp_std='c++17') 或链接器标志 (-Dcpp_link_args='-Wl,-z,defs')。
要了解更多选项,请参阅Meson 内置选项文档页面。
Meson 还支持标准的环境变量 CFLAGS, CXXFLAGS, FFLAGS 和 LDFLAGS 以注入额外的标志 - 与前一节中相同的警告一样,这些环境变量仅在清理构建时才会被接受,而不是增量构建。
使用 Meson 进行不同的构建类型
Meson 在配置项目时提供了不同的构建类型。您可以在Meson 文档的“核心选项”部分中查看可用的构建类型选项。
假设您是从头开始构建的(如果需要,请执行 git clean -xdf),您可以按以下步骤配置构建以使用 debug 构建类型:
meson setup build --buildtype debug --prefix=$PWD/build-install
- 1
现在,您可以使用 dev.py 接口进一步构建、安装和测试 SciPy:
python dev.py -s linalg
- 1
这将有效,因为在初始配置之后,Meson 将记住配置选项。
控制构建并行性
默认情况下,ninja 将启动 2*n_cpu + 2 个并行构建作业,其中 n_cpu 是物理 CPU 核心数。在绝大多数情况下,这是可以接受的,并且可以获得接近最优的构建时间。但在某些情况下,当机器的 RAM 相对于 CPU 核心数较少时,可能会导致作业内存不足。如果发生这种情况,请降低作业数 N,以确保每个作业至少有 2 GB RAM。例如,启动 6 个作业:
python -m pip install . -Ccompile-args="-j6"
- 1
或:
python dev.py build -j6
- 1
并行使用 GCC 和 Clang 进行构建
在同一个仓库中拥有几个不同的 SciPy 构建可能非常有用,例如用于比较两个编译器之间的差异以诊断问题。如前所述,Meson 完全是非就地构建,因此不同的构建不会互相干扰。在本节的其余部分中,我们假设 GCC 是默认的。例如,让我们使用 GCC 和 Clang 进行构建。
-
使用 GCC 进行构建:
python dev.py build- 1
使用上述命令,Meson 将使用(默认的)GCC 编译器在 build 目录中进行构建,并安装到 build-install 目录中。
-
使用 Clang 进行构建:
CC=clang CXX=clang++ FC=gfortran python dev.py --build-dir=build-clang build- 1
使用上述命令,Meson 将使用 Clang、Clang++ 和 Gfortran 编译器在 build-clang 目录中进行构建,并将 SciPy 安装到 build-clang-install 中。
Meson 将记住 build-clang 目录下的编译器选择,并且无法更改,因此每次未来调用 python dev.py --build-dir=build-clang <command> 时将自动使用 Clang。
提示:使用别名可以使这个过程更加简便,例如 alias dev-clang="python dev.py --build-dir=build-clang",然后执行 dev-clang build。
拥有两个构建的常见原因是进行比较。例如,要在两个编译器的构建中运行 scipy.linalg 测试,请执行:
python dev.py -s linalg # run tests for the GCC build
python dev.py --build-dir build-clang -s linalg # run tests for the Clang build
- 1
- 2
BLAS 和 LAPACK
原文链接:
docs.scipy.org/doc/scipy-1.12.0/building/blas_lapack.html
选择 BLAS 和 LAPACK 库
除了默认的 OpenBLAS,通过 Meson 构建选项 实现 BLAS 和 LAPACK 库的选择。例如,要选择普通的 libblas 和 liblapack(这通常是 Linux 发行版上的 Netlib BLAS/LAPACK,并且可以在 conda-forge 上动态切换实现),请使用:
$ # for a development build
$ python dev.py build -C-Dblas=blas -C-Dlapack=lapack
$ # to build and install a wheel
$ python -m build -Csetup-args=-Dblas=blas -Csetup-args=-Dlapack=lapack
$ pip install dist/scipy*.whl
$ # Or, with pip>=23.1, this works too:
$ python -m pip -Csetup-args=-Dblas=blas -Csetup-args=-Dlapack=lapack
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
其他应该可以工作的选项(只要安装了 pkg-config 或 CMake 支持),包括 mkl、atlas 和 blis。
使用 pkg-config 在非标准位置检测库
BLAS 和 LAPACK 检测的内部工作原理是 Meson 首先尝试使用 pkg-config 发现指定的库,然后使用 CMake。如果你只有一个独立的共享库文件(例如,在 /a/random/path/lib/ 中的 armpl_lp64.so 和对应的头文件在 /a/random/path/include/ 中),那么你需要自己编写一个 pkg-config 文件。它应该具有相同的名称(所以在这个例子中是 armpl_lp64.pc),可以放置在任何位置。PKG_CONFIG_PATH 环境变量应设置为指向 .pc 文件的位置。该文件的内容应为:
libdir=/path/to/library-dir # e.g., /a/random/path/lib
includedir=/path/to/include-dir # e.g., /a/random/path/include
version=1.2.3 # set to actual version
extralib=-lm -lpthread -lgfortran # if needed, the flags to link in dependencies
Name: armpl_lp64
Description: ArmPL - Arm Performance Libraries
Version: ${version}
Libs: -L${libdir} -larmpl_lp64 # linker flags
Libs.private: ${extralib}
Cflags: -I${includedir}
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
要检查这是否按预期工作,您应该能够运行:
$ pkg-config --libs armpl_lp64
-L/path/to/library-dir -larmpl_lp64
$ pkg-config --cflags armpl_lp64
-I/path/to/include-dir
- 1
- 2
- 3
- 4
指定要使用的 Fortran ABI
一些线性代数库使用 g77 ABI(也称为“f2c 调用约定”),其他使用 GFortran ABI,这两种 ABI 不兼容。因此,如果您使用 gfortran 构建 SciPy 并链接到像 MKL 这样使用 g77 ABI 构建的线性代数库,将会出现异常或段错误。SciPy 通过使用 ABI 包装器解决了这个问题,ABI 包装器依赖于 CBLAS API 来处理 BLAS API 中少数函数存在的问题。
请注意,SciPy 在构建时需要知道需要执行的操作,构建系统将自动检查线性代数库是否为 MKL 或 Accelerate(这两者始终使用 g77 ABI),如果是,则使用 CBLAS API 而不是 BLAS API。如果自动检测失败或用户希望覆盖此自动检测机制以构建针对普通的 libblas/liblapack(例如 conda-forge 所做的就是这样),请使用 -Duse-g77-abi=true 构建选项。例如:
$ python -m build -C-Duse-g77-abi=true -Csetup-args=-Dblas=blas -Csetup-args=-Dlapack=lapack
- 1
工作进行中
计划完全支持这些选项,但目前不能直接使用:
-
ILP64(64 位整数大小)构建:SciPy 的大部分支持使用 ILP64 BLAS/LAPACK。请注意,支持仍然不完整,因此 SciPy 还 需要 LP64(32 位整数大小)BLAS/LAPACK。
-
自动从多个可能的 BLAS 和 LAPACK 选项中选择,并按用户指定的优先顺序进行选择
跨编译
原文链接:
docs.scipy.org/doc/scipy-1.12.0/building/cross_compilation.html
跨编译是一个复杂的主题,我们目前只添加了一些希望有帮助的提示。截至 2023 年 5 月,基于crossenv的跨编译已知可以工作,例如在 conda-forge 中使用。没有crossenv的跨编译需要一些手动覆盖。您可以通过向meson-python传递选项来指示这些覆盖项到meson setup中。
所有已知成功进行 SciPy 跨编译的发行版都在使用python -m build(pypa/build),但使用pip也应该是可能的。以下是这些发行版上 SciPy“构建配方”的链接:
另请参阅Meson 关于跨编译的文档以了解您可能需要向 Meson 传递哪些选项才能成功进行跨编译。
一个常见问题是,numpy和pythran需要运行 Python 代码以获取它们的包含目录。这往往效果不佳,可能会意外地从构建(本地)Python 中获取软件包,而不是主机(跨)Python,或者需要crossenv或 QEMU 来运行主机 Python。为了避免这个问题,在您的跨文件中指定相关目录的路径:
[constants]
sitepkg = '/abspath/to/host-pythons/site-packages/'
[properties]
numpy-include-dir = sitepkg + 'numpy/core/include'
pythran-include-dir = sitepkg + 'pythran'
- 1
- 2
- 3
- 4
- 5
- 6
要获取有关跨编译的更多详细信息和当前状态,请参阅:
-
SciPy 跨编译需求和问题的跟踪问题:scipy#14812
-
Python 中的跨编译状态:pypackaging-native 关键问题页面
构建可再分发的二进制文件
原文:
docs.scipy.org/doc/scipy-1.12.0/building/redistributable_binaries.html
当使用python -m build或pip wheel构建 SciPy 轮子时,该轮子将依赖外部共享库(至少是 BLAS/LAPACK 和 Fortran 编译器运行时库,也许还有其他库)。因此,这样的轮子只能在构建它们的系统上运行。有关更多背景信息,请参阅 “构建和安装或上传工件”下的 pypackaging-native 内容。
因此,这样的轮子是生成可分发的二进制文件的中间阶段。最终的二进制文件可能是一个轮子 - 在这种情况下,请运行auditwheel(Linux)、delocate(macOS)或delvewheel(Windows)来将所需的共享库打包到轮子中。
最终的二进制文件也可能是另一种打包格式(例如 .rpm、.deb 或 .conda 包)。在这种情况下,有特定于打包生态系统的工具,首先将轮子安装到临时区域,然后使该安装位置中的扩展模块可重定位(例如通过重写 RPATHs),最后将其重新打包为最终的包格式。
了解 Meson
原文链接:
docs.scipy.org/doc/scipy-1.12.0/building/understanding_meson.html
构建 SciPy 依赖于以下工具,这些工具可视为构建系统的一部分:
-
meson:Meson 构建系统,可作为纯 Python 包从 PyPI 或 conda-forge 安装 -
ninja:Meson 调用的实际构建工具(例如,调用编译器)。也可以从 PyPI(所有常见平台)或 conda-forge 安装。 -
pkg-config:用于发现依赖项(特别是 BLAS/LAPACK)的工具。在 conda-forge(以及 Homebrew、Chocolatey 和 Linux 包管理器)可用,但未在 PyPI 上打包。 -
meson-python:Python 构建后端(即通过pyproject.toml中的钩子由pip或pypa/build调用的东西)。这是 Meson 之上的一个薄层,主要作用是(a)与构建前端接口,以及(b)生成具有有效文件名和元数据的 sdists 和 wheels。
使用 Meson 进行构建分为几个阶段:
-
配置阶段(
meson setup)用于检测编译器、依赖项和构建选项,并创建构建目录和build.ninja文件, -
编译阶段(
meson compile或ninja),在这里编译作为构建 SciPy 包的一部分的扩展模块, -
安装阶段(
meson install)用于从源和构建目录安装可安装文件到目标安装目录,
Meson 拥有良好的构建依赖跟踪系统,因此第二次调用构建将仅重新构建任何源或依赖项发生更改的目标。
了解更多关于 Meson 的信息
Meson 拥有非常好的文档;阅读它是值得的,通常也是“如何做 X”的最佳答案来源。此外,可以免费获取 Meson 的详尽 pdf 书籍,网址为nibblestew.blogspot.com/2021/12/this-year-receive-gift-of-free-meson.html。
若要了解 Meson 使用的设计原则的更多信息,最近链接的讲座从mesonbuild.com/Videos也是一个很好的资源。
构建阶段的解释
这仅供教学目的;不应单独执行这些阶段。根目录中的 dev.py 脚本也包含这些步骤,并且可以用于深入研究。
假设我们从一个干净的 repo 和完全设置好的 conda 环境开始:
git clone git@github.com:scipy/scipy.git
git submodule update --init
mamba env create -f environment.yml
mamba activate scipy-dev
- 1
- 2
- 3
- 4
现在运行构建的配置阶段,并指示 Meson 将构建工件放在相对于 repo 根目录的build/和本地安装在build-install/下,请执行:
meson setup build --prefix=$PWD/build-install
- 1
然后运行构建的编译阶段,请执行:
ninja -C build
- 1
在上述命令中,-C后面跟着构建目录的名称。您可以同时拥有多个构建目录。Meson 是完全非原位的,因此这些构建不会相互干扰。例如,您可以在不同的目录中拥有 GCC 构建、Clang 构建和调试构建。
然后将 SciPy 安装到前缀(build-install/这里,但请注意这只是我们随意选择的名称):
meson install -C build
- 1
然后它将安装到build-install/lib/python3.11/site-packages/scipy,这不在您的 Python 路径中,所以要添加它(再次强调,这是为了学习目的,显式使用PYTHONPATH通常不是最佳选择):
export PYTHONPATH=$PWD/build-install/lib/python3.11/site-packages/
- 1
现在我们应该能够导入scipy并运行测试。请记住,我们需要移出仓库的根目录,以确保我们使用的是包而不是本地的scipy/源目录。
cd doc
python -c "from scipy import constants as s; s.test()"
- 1
- 2
上述命令运行单个模块constants的测试。还有其他运行测试的方法,例如:
pytest --pyargs scipy
- 1
完整的测试套件应该通过,在 Linux 上没有任何构建警告(至少在 CI 中强制使用-Werror的 GCC 版本),在其他平台上最多只能有少量警告。
检查构建步骤
原文链接:
docs.scipy.org/doc/scipy-1.12.0/building/introspecting_a_build.html
当您遇到特定 Python 扩展模块或其他构建目标的问题时,有多种方法可以确切了解构建系统正在执行的操作。除了查看感兴趣目标的meson.build内容外,这些还包括:
-
在构建目录中阅读生成的
build.ninja文件, -
使用
meson introspect了解有关目标的构建选项、依赖项和使用的标志, -
阅读
<build-dir>/meson-info/*.json以获取有关发现的依赖项、Meson 文件安装位置等详细信息。
所有这些内容都在构建的配置阶段之后(即meson setup运行后)可用。通常,查看此信息比运行构建并阅读完整的构建日志更有效。
ninja.build文件
例如,假设我们对scipy.linalg._decomp_update感兴趣。从scipy/linalg/meson.build我们了解到此扩展是使用模板化的 Cython 代码编写的,并且除了numpy目录外没有使用任何特殊的编译标志或包含目录。因此,下一步是查看build.ninja。在编辑器中打开该文件并搜索_decomp_update。您将找到适用的一组通用和特定目标的规则(注意,此代码块中的注释不在build.ninja中,仅在此文档部分中添加以解释正在发生的事情)。
# These rules are usually not needed to understand the problem, but can be looked up at the top of the file: rule c_COMPILER command = /home/username/anaconda3/envs/scipy-dev/bin/x86_64-conda-linux-gnu-cc $ARGS -MD -MQ $out -MF $DEPFILE -o $out -c $in deps = gcc depfile = $DEPFILE_UNQUOTED description = Compiling C object $out rule c_LINKER command = /home/username/anaconda3/envs/scipy-dev/bin/x86_64-conda-linux-gnu-cc $ARGS -o $out $in $LINK_ARGS description = Linking target $out # step 1: `.pyx.in` to `.pyx` code generation with Tempita build scipy/linalg/_decomp_update.pyx: CUSTOM_COMMAND ../scipy/linalg/_decomp_update.pyx.in | ../scipy/_build_utils/tempita.py /home/username/anaconda3/envs/scipy-dev/bin/python3.10 COMMAND = /home/username/anaconda3/envs/scipy-dev/bin/python3.10 ../scipy/_build_utils/tempita.py ../scipy/linalg/_decomp_update.pyx.in -o scipy/linalg description = Generating$ scipy/linalg/_decomp_update$ with$ a$ custom$ command # step 2: `.pyx` to `.c` compilation with Cython build scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so.p/_decomp_update.c: CUSTOM_COMMAND scipy/linalg/_decomp_update.pyx | /home/username/code/scipy/scipy/_build_utils/cythoner.py scipy/__init__.py scipy/linalg/__init__.py scipy/linalg/cython_blas.pyx DESC = Generating$ 'scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so.p/_decomp_update.c'. COMMAND = /home/username/anaconda3/envs/scipy-dev/bin/python3.10 /home/username/code/scipy/scipy/_build_utils/cythoner.py scipy/linalg/_decomp_update.pyx scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so.p/_decomp_update.c # step 3: use C compiler to go from `.c` to object file (`.o`) build scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so.p/meson-generated__decomp_update.c.o: c_COMPILER scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so.p/_decomp_update.c DEPFILE = scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so.p/meson-generated__decomp_update.c.o.d DEPFILE_UNQUOTED = scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so.p/meson-generated__decomp_update.c.o.d ARGS = -Iscipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so.p -Iscipy/linalg -I../scipy/linalg -I/home/username/anaconda3/envs/scipy-dev/lib/python3.10/site-packages/numpy/core/include -I/home/username/anaconda3/envs/scipy-dev/include/python3.10 -fvisibility=hidden -fdiagnostics-color=always -D_FILE_OFFSET_BITS=64 -Wall -Winvalid-pch -std=c99 -O2 -g -Wno-unused-but-set-variable -Wno-unused-function -Wno-conversion -Wno-misleading-indentation -fPIC -Wno-cpp # step 4: generate a symbol file (uses `meson --internal symbolextractor`); you can safely ignore this step build scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so.p/_decomp_update.cpython-310-x86_64-linux-gnu.so.symbols: SHSYM scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so IMPLIB = scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so # step 5: link the `.o` file to obtain the file extension module (`.so`) build scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so: c_LINKER scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so.p/meson-generated__decomp_update.c.o | /home/username/anaconda3/envs/scipy-dev/x86_64-conda-linux-gnu/sysroot/lib64/libm-2.12.so /home/username/anaconda3/envs/scipy-dev/x86_64-conda-linux-gnu/sysroot/usr/lib64/libm.a LINK_ARGS = -L/home/username/anaconda3/envs/scipy-dev/lib -Wl,--as-needed -Wl,--allow-shlib-undefined -shared -fPIC -Wl,--start-group -lm -Wl,--end-group -Wl,-O2 -Wl,--sort-common -Wl,--as-needed -Wl,-z,relro -Wl,-z,now -Wl,--disable-new-dtags -Wl,--gc-sections -Wl,--allow-shlib-undefined -Wl,-rpath,/home/username/anaconda3/envs/scipy-dev/lib -Wl,-rpath-link,/home/username/anaconda3/envs/scipy-dev/lib
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
使用meson introspect
如果我们想从另一个角度查看_decomp_update,可以使用(例如)meson introspect --targets -i <build-dir> > targets.json生成可读的 JSON。搜索生成的文件以查找我们感兴趣的目标显示:
{ "name": "_decomp_update", "id": "b4ac6f0@@_decomp_update@cus", "type": "custom", "defined_in": "/home/username/code/scipy/scipy/linalg/meson.build", "filename": [ "/home/username/code/scipy/build/scipy/linalg/_decomp_update.pyx" ], "build_by_default": false, "target_sources": [ { "language": "unknown", "compiler": [ "/home/username/anaconda3/envs/scipy-dev/bin/python3.10", "/home/username/code/scipy/scipy/_build_utils/tempita.py", "@INPUT@", "-o", "@OUTDIR@" ], "parameters": [], "sources": [ "/home/username/code/scipy/scipy/linalg/_decomp_update.pyx.in" ], "generated_sources": [] } ], "extra_files": [], "subproject": null, "installed": false }, { "name": "_decomp_update.cpython-310-x86_64-linux-gnu", "id": "b4ac6f0@@_decomp_update.cpython-310-x86_64-linux-gnu@sha", "type": "shared module", "defined_in": "/home/username/code/scipy/scipy/linalg/meson.build", "filename": [ "/home/username/code/scipy/build/scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so" ], "build_by_default": true, "target_sources": [ { "language": "c", "compiler": [ "/home/username/anaconda3/envs/scipy-dev/bin/x86_64-conda-linux-gnu-cc" ], "parameters": [ "-I/home/username/code/scipy/build/scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so.p", "-I/home/username/code/scipy/build/scipy/linalg", "-I/home/username/code/scipy/scipy/linalg", "-I/home/username/anaconda3/envs/scipy-dev/lib/python3.10/site-packages/numpy/core/include", "-I/home/username/anaconda3/envs/scipy-dev/include/python3.10", "-fvisibility=hidden", "-fdiagnostics-color=always", "-D_FILE_OFFSET_BITS=64", "-Wall", "-Winvalid-pch", "-std=c99", "-O2", "-g", "-Wno-unused-but-set-variable", "-Wno-unused-function", "-Wno-conversion", "-Wno-misleading-indentation", "-fPIC", "-Wno-cpp" ], "sources": [], "generated_sources": [ "/home/username/code/scipy/build/scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so.p/_decomp_update.c" ] } ], "extra_files": [], "subproject": null, "installed": true, "install_filename": [ "/home/username/code/scipy/build-install/lib/python3.10/site-packages/scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so" ] },
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
- 54
- 55
- 56
- 57
- 58
- 59
- 60
- 61
- 62
- 63
- 64
- 65
- 66
- 67
- 68
- 69
- 70
- 71
- 72
- 73
- 74
- 75
- 76
- 77
- 78
- 79
这告诉我们很多信息,例如将使用哪些包含目录、Cython 生成的 C 代码在何处找到以及使用了哪些编译标志。meson introspect --help对其全部功能范围和使用方法有很好的文档。
meson-info JSON 文件
<build-dir>/meson-info/中有许多不同的 JSON 文件。这些文件具有描述性名称,暗示其内容。例如,最终的_decomp_update扩展安装到何处在intro-install_plan.json中描述(注意,这些文件未经美化打印,通过 JSON 格式化器运行它们有助于):
"/home/username/code/scipy/build/scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so":{
"destination":"{py_platlib}/scipy/linalg/_decomp_update.cpython-310-x86_64-linux-gnu.so",
"tag":"runtime"
},
- 1
- 2
- 3
- 4
我们还可能对检测到的依赖项在构建的配置阶段有何安装计划等情况感兴趣。因此,我们查看intro-dependencies.json:
[ { "name":"python", "version":"3.10", "compile_args":[ "-I/home/username/anaconda3/envs/scipy-dev/include/python3.10" ], "link_args":[ ] }, { "name":"openblas", "version":"0.3.20", "compile_args":[ "-I/home/username/anaconda3/envs/scipy-dev/include" ], "link_args":[ "/home/username/anaconda3/envs/scipy-dev/lib/libopenblas.so" ] }, { "name":"threads", "version":"unknown", "compile_args":[ "-pthread" ], "link_args":[ "-pthread" ] } ]
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
这告诉我们有三个发现的依赖项。注意:numpy和其他几个构建时依赖项因尚未使用内置的dependency() Meson 命令进行搜索而未列出。
Meson 和 distutils 的工作方式
原文链接:
docs.scipy.org/doc/scipy-1.12.0/building/distutils_equivalents.html
旧的工作流(基于 numpy.distutils):
runtests.py 文件在提交 0f73f92255253ec5dff2de5ca45d8d3bdda03f92 中已被移除 [¹^_]。
-
python runtests.py -
python setup.py build_ext -i+export PYTHONPATH=/home/username/path/to/scipy/reporoot(然后在 SciPy 中编辑纯 Python 代码并使用python some_script.py运行)。 -
python setup.py develop- 这与(2)类似,除了在地方构建永久可见于环境中。 -
python setup.py bdist_wheel+pip install dist/scipy*.whl- 在当前环境中构建 wheel(即使用已安装的 numpy 等)并安装它。 -
pip install .- 在与pyproject.toml中的依赖项进行隔离构建并安装 wheel。注意:请小心,这通常不是用于开发安装的正确命令 - 通常你应该使用 (4) 或pip install . -v --no-build-isolation。
新的工作流(基于 Meson 和 meson-python):
-
python dev.py -
pip install -e . --no-build-isolation(参见meson-python文档) -
与(2)相同
-
python -m build --no-isolation+pip install dist/scipy*.whl- 参见 pypa/build。 -
pip install .
[¹^_]: GitHub 上的提交 0f73f92255253ec5dff2de5ca45d8d3bdda03f92。
开发者文档
下面您将找到有关贡献的一般信息。如需了解期望或计划在哪里需要帮助或新功能,请参阅路线图。如需更详细地了解 SciPy 项目的工作方式,请参阅组织部分。
贡献信息
-
SciPy 行为准则
-
贡献方式
-
贡献者快速入门指南
-
开发工作流程
-
SciPy 贡献者指南
路线图
-
SciPy 路线图
-
详细的 SciPy 路线图
-
工具链路线图
SciPy 组织
-
SciPy 核心开发者指南
-
SciPy API 开发指南
-
SciPy 项目治理
贡献信息
SciPy 行为准则
原文链接:
docs.scipy.org/doc/scipy-1.12.0/dev/conduct/code_of_conduct.html
简介
此行为准则适用于 SciPy 项目管理的所有空间,包括所有公共和私人邮件列表、问题跟踪器、维基、博客、Twitter 以及我们社区使用的任何其他沟通渠道。虽然 SciPy 项目不组织面对面的活动,但与我们社区相关的活动应具有与此相似精神的行为准则。
所有参与 SciPy 社区的人士,无论是正式参与还是非正式参与,或者声称与项目有任何关联的人,在任何与项目相关的活动中,特别是在代表项目时,都应遵守此行为准则。
此代码既不详尽也不完整。它用来概括我们对协作共享环境和目标的共同理解。请尽量遵循这一准则的精神,创造一个友好且富有成效的环境,以丰富周围的社区。
具体指南
我们努力做到:
-
请保持开放。我们欢迎任何人参与我们的社区。我们更倾向于使用公共沟通方法进行项目相关消息的传递,除非讨论的内容比较敏感。这也适用于帮助或项目相关支持的请求;公共支持请求不仅更有可能得到问题的答复,还能确保更容易发现和纠正任何不小心的错误。
-
具有共情、欢迎、友好和耐心。我们共同努力解决冲突,并假设他人有良好的意图。我们每个人偶尔可能会感到一些挫折,但我们不允许挫折演变成个人攻击。一个让人感到不舒服或受到威胁的社区是没有生产力的。
-
请务必协作。我们的工作将被他人使用,反过来我们也会依赖于他人的工作。当我们为项目利益而创造某事物时,我们愿意向他人解释其工作原理,以便他们能够在此基础上进行改进。我们做出的每一个决定都会影响用户和同事,因此我们在决策时认真对待这些后果。
-
请多提问题。没有人什么都懂!早些时候提问可以避免后面的很多问题,因此我们鼓励提问,尽管我们可能会把它们指向适当的论坛。我们将尽力响应并提供帮助。
-
我们在选择用词时要小心谨慎。在沟通中,我们要谨慎和尊重,对自己的言论负责任。善待他人。不要侮辱或贬低其他参与者。我们不会接受骚扰或其他排斥行为,例如:
- 针对他人的暴力威胁或语言。
- 性别歧视、种族歧视或其他歧视性笑话和用语。
- 发布性别暴露或暴力材料。
- 发布(或威胁发布)他人的个人身份信息(“doxing”)。
- 分享私人内容,如私下发送的电子邮件或非公开的内容,或未经发件人同意的未记录论坛,如 IRC 频道历史。
- 个人侮辱,尤其是使用种族主义或性别歧视的词语。
- 不受欢迎的性别关注。
- 过度的粗俗语言。请避免使用脏话;人们对脏话的敏感程度大不相同。
- 反复骚扰他人。一般来说,如果有人要求您停止,那么请停止。
- 提倡或鼓励上述任何行为。
多样性声明
SciPy 项目欢迎并鼓励所有人参与。我们致力于打造一个每个人都乐于参与的社区。尽管我们可能无法始终满足每个人的偏好,但我们会尽力友善对待每一位成员。
无论您如何定义自己或他人如何看待您:我们都欢迎您。尽管没有任何一份清单可以涵盖所有情况,我们明确尊重:年龄、文化、种族、基因型、性别认同或表达、语言、国籍、神经类型、表型、政治信仰、职业、种族、宗教、性取向、社会经济地位、亚文化和技术能力,只要这些不与本行为准则冲突。
尽管我们欢迎使用所有语言流利的人,但 SciPy 的开发工作是用英语进行的。
SciPy 社区的行为标准详见上述《行为准则》。我们社区的参与者应在所有互动中遵守这些标准,并帮助他人也做到如此(请参见下一节)。
报告指南
我们知道,在互联网上,通信往往以或演变为明显且公然的滥用。我们也意识到,有时人们可能会有不好的一天,或者对这些行为准则中的某些指导不太了解。在决定如何回应违反这一准则时,请记住这一点。
对于明显故意违反的情况,请向行为准则委员会报告(详见下文)。对于可能无意的违规行为,您可以在公开或私下向当事人指出此行为准则。如果您不愿意这样做,请随时直接向行为准则委员会报告,或者寻求委员会的建议,保证您的信息不会泄露。
您可以向 SciPy 行为准则委员会报告问题,联系邮箱为 scipy-conduct@googlegroups.com。目前,委员会成员包括:
-
Stefan van der Walt
-
Nathaniel J. Smith
-
Ralf Gommers
如果您的报告涉及任何委员会成员,或者他们觉得自己在处理中存在利益冲突,那么他们将自行退出考虑您的报告。或者,如果出于任何原因,您感到不舒服向委员会报告,您也可以联系:
-
SciPy Steering Committee 主席:Ralf Gommers 或
-
高级NumFOCUS 员工:conduct@numfocus.org
事件报告解决与行为准则执行
本节总结了最重要的点,更多细节请参阅 SciPy 行为准则 - 如何跟进报告。
我们将调查并回应所有投诉。SciPy 行为准则委员会和 SciPy 指导委员会(如果涉及)将保护报告人的身份,并将投诉内容视为保密(除非报告人另有同意)。
在严重且明显违规情况下,例如人身威胁或使用暴力、性别歧视或种族主义言论,我们将立即将发起者从 SciPy 通讯渠道中断开;详细信息请参阅手册。
对于不涉及明显严重违规的情况,处理接收到的行为准则违规报告的过程将是:
-
确认已收到报告
-
合理的讨论/反馈
-
调解(如果反馈没有帮助,并且只有报告人和被报告人同意)
-
透明决策执行(见决议),由行为准则委员会负责。
委员会将尽快回复任何报告,并且最迟在 72 小时内做出回应。
尾注
我们感谢以下文档背后的团体,我们从中获取内容和灵感:
-
针对他人的暴力威胁或语言。
- 性别歧视、种族歧视或其他歧视性笑话和用语。
- 发布性别暴露或暴力材料。
- 发布(或威胁发布)他人的个人身份信息(“doxing”)。
- 分享私人内容,如私下发送的电子邮件或非公开的内容,或未经发件人同意的未记录论坛,如 IRC 频道历史。
- 个人侮辱,尤其是使用种族主义或性别歧视的词语。
- 不受欢迎的性别关注。
- 过度的粗俗语言。请避免使用脏话;人们对脏话的敏感程度大不相同。
- 反复骚扰他人。一般来说,如果有人要求您停止,那么请停止。
- 提倡或鼓励上述任何行为。
多样性声明
SciPy 项目欢迎并鼓励所有人参与。我们致力于打造一个每个人都乐于参与的社区。尽管我们可能无法始终满足每个人的偏好,但我们会尽力友善对待每一位成员。
无论您如何定义自己或他人如何看待您:我们都欢迎您。尽管没有任何一份清单可以涵盖所有情况,我们明确尊重:年龄、文化、种族、基因型、性别认同或表达、语言、国籍、神经类型、表型、政治信仰、职业、种族、宗教、性取向、社会经济地位、亚文化和技术能力,只要这些不与本行为准则冲突。
尽管我们欢迎使用所有语言流利的人,但 SciPy 的开发工作是用英语进行的。
SciPy 社区的行为标准详见上述《行为准则》。我们社区的参与者应在所有互动中遵守这些标准,并帮助他人也做到如此(请参见下一节)。
报告指南
我们知道,在互联网上,通信往往以或演变为明显且公然的滥用。我们也意识到,有时人们可能会有不好的一天,或者对这些行为准则中的某些指导不太了解。在决定如何回应违反这一准则时,请记住这一点。
对于明显故意违反的情况,请向行为准则委员会报告(详见下文)。对于可能无意的违规行为,您可以在公开或私下向当事人指出此行为准则。如果您不愿意这样做,请随时直接向行为准则委员会报告,或者寻求委员会的建议,保证您的信息不会泄露。
您可以向 SciPy 行为准则委员会报告问题,联系邮箱为 scipy-conduct@googlegroups.com。目前,委员会成员包括:
-
Stefan van der Walt
-
Nathaniel J. Smith
-
Ralf Gommers
如果您的报告涉及任何委员会成员,或者他们觉得自己在处理中存在利益冲突,那么他们将自行退出考虑您的报告。或者,如果出于任何原因,您感到不舒服向委员会报告,您也可以联系:
-
SciPy Steering Committee 主席:Ralf Gommers 或
-
高级NumFOCUS 员工:conduct@numfocus.org
事件报告解决与行为准则执行
本节总结了最重要的点,更多细节请参阅 SciPy 行为准则 - 如何跟进报告。
我们将调查并回应所有投诉。SciPy 行为准则委员会和 SciPy 指导委员会(如果涉及)将保护报告人的身份,并将投诉内容视为保密(除非报告人另有同意)。
在严重且明显违规情况下,例如人身威胁或使用暴力、性别歧视或种族主义言论,我们将立即将发起者从 SciPy 通讯渠道中断开;详细信息请参阅手册。
对于不涉及明显严重违规的情况,处理接收到的行为准则违规报告的过程将是:
-
确认已收到报告
-
合理的讨论/反馈
-
调解(如果反馈没有帮助,并且只有报告人和被报告人同意)
-
透明决策执行(见决议),由行为准则委员会负责。
委员会将尽快回复任何报告,并且最迟在 72 小时内做出回应。
尾注
我们感谢以下文档背后的团体,我们从中获取内容和灵感:



