- 1UE4 HTC VIVE 多人联机_ue vr联机
- 2SQL 笛卡尔积现象_sql笛卡尔积怎么形成的
- 3Apollo配置中心搭建_apollo 配置中心搭建
- 4自古套路得人心:安全岗面经(人生苦短,请勿入坑二进制)
- 5timerfd和timer
- 62024年华为OD机试真题-测试用例执行计划-Python-OD统一考试(C卷)
- 7Spring Boot 3 + JWT + Security 联手打造安全帝国:一篇文章让你掌握未来!_springboot3 security
- 8项目经理的能力要求_项目经理能力要求
- 9四面字节跳动,抗住算法后艰难拿到后端offer,薪资18K_字节跳动hr面试薪水怎么谈
- 10Day 9: TextBlob——对文本进行情感分析_用textblob如何给大段文章做情感分析
Python绘制神经网络模型图_怎么在python图中画出网络模型架构
赞
踩
本文介绍基于Python语言,对神经网络模型的结构进行可视化绘图的方法。
最近需要进行神经网络结构模型的可视化绘图工作。查阅多种方法后,看到很多方法都比较麻烦,例如单纯利用graphviz模块,就需要手动用DOT语言进行图片描述,比较花时间;最终,发现利用第三方的ann_visualizer模块,可以实现对已有神经网络的直接可视化,过程较为方便,本文对此加以详细介绍。
此外,如果需要在MATLAB中实现神经网络构建与简单的可视化,大家可以查看MATLAB人工神经网络ANN代码;如果要借助软件或在线工具进行不需要代码的神经网络可视化,可以查看我们后期的博客。
相关环境的版本信息:Anaconda Navigator:1.10.0;Python:3.8.5。
首先,下载与安装必要的模块ann_visualizer。打开Anaconda Prompt (Soft)。
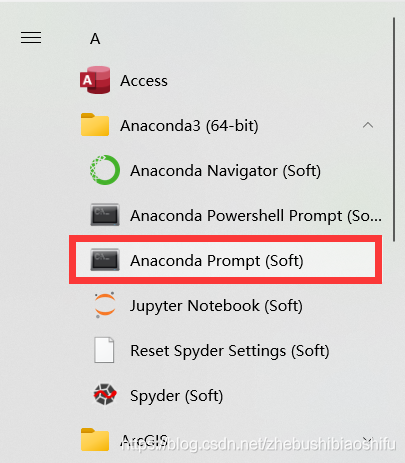
在弹出的界面中输入:
pip install ann_visualizer
即可完成ann_visualizer模块的安装。
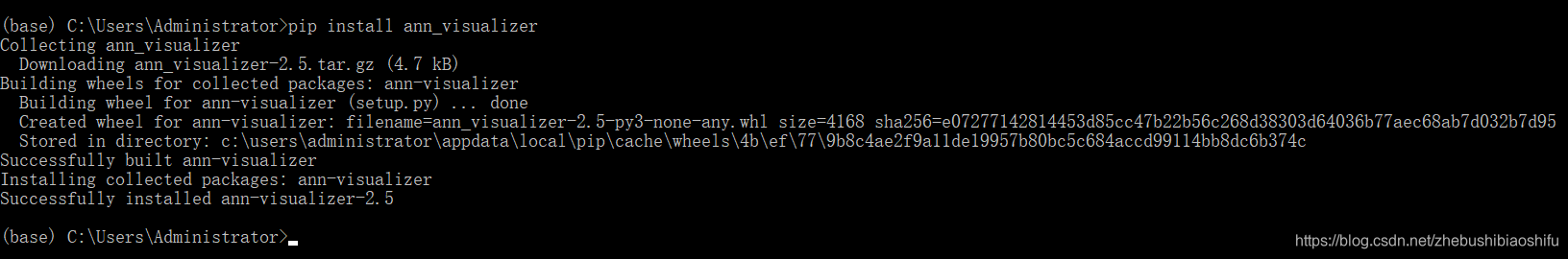
接下来,我们就可以借助以下仅仅一句代码对神经网络模型进行可视化了。
ann_viz(DNNModel,view=True,filename='G:/CropYield/02_CodeAndMap/01_SavedPicture/MyANN.gv',title='ANN')
其中,DNNModel就是我们已经建立好的神经网络模型,任意神经网络模型均可——可以是一个简单的浅层人工神经网络,也可以是一个相对复杂的全连接深度神经网络;view表示是否在代码执行后直接显示绘图结果;filename是绘图结果的保存位置,需要以.gv结尾;title就是神经网络图片的名称。
在这里,我就直接以Python TensorFlow深度神经网络回归:keras.Sequential中介绍并建立的深度神经网络加以可视化。
第一次运行代码时发现,出现以下报错:
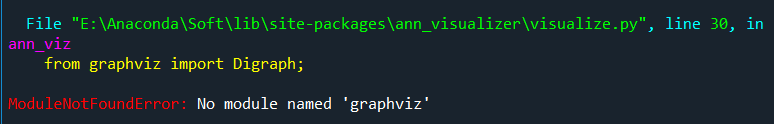
报错提示我没有安装graphviz模块,但其实之前在进行随机森林决策树的可视化(也就是Python实现随机森林RF并对比自变量的重要性)时,早已经将这一模块安装过了,并且当时用到graphviz这一模块的代码也没有报错。通过查阅,发现这里需要重新安装一下python-graphviz这个新的模块。因此我们打开Anaconda Prompt (Soft),输入代码:
conda install python-graphviz
如下图所示:
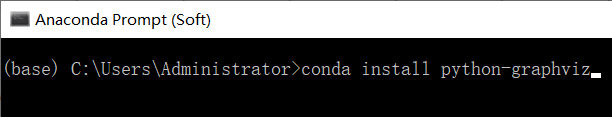
安装之后这里就不报错啦~
结果紧接着又报出了新的错误,说我的keras模块没有安装:

这就不对了,明明在进行深度神经网络构建时都没有出现问题,甚至在这一句报错的下方连深度神经网络的误差绘制曲线都能显示(误差曲线的精度的确很差,大家不用在意~因为这里我们仅仅是做一个示范,所以Epoch次数就调得很小),说明keras模块应该是没问题的。
随后考虑到,这里报错的keras是在ann_visualizer的文件环境下,可能是环境不同导致的。打开Anaconda Navigator,在base (root)环境下确实找不到keras:
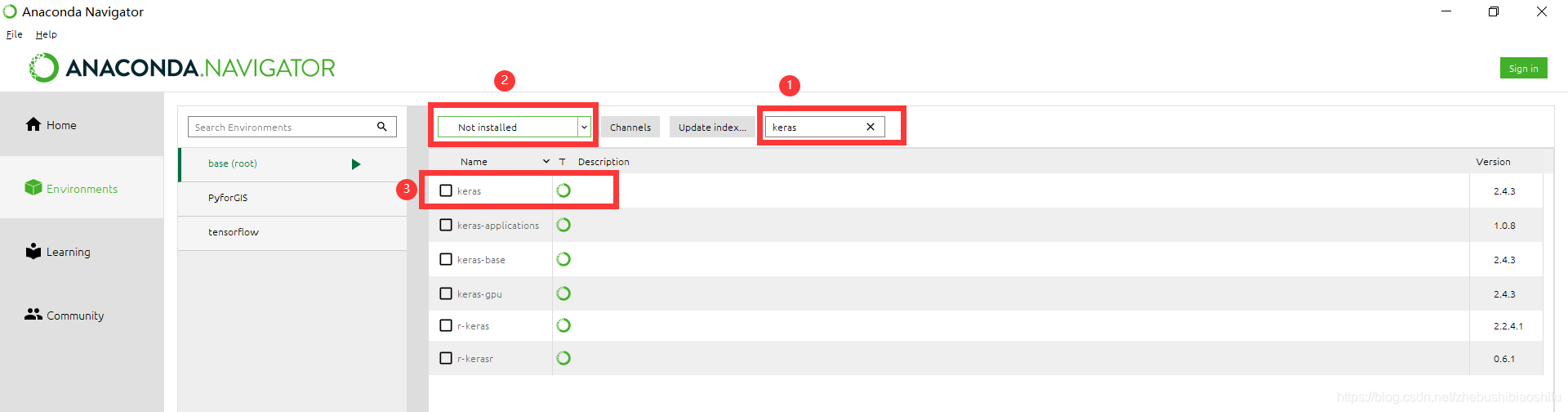
那么我这里就图方便,直接在base (root)环境下再安装一个keras。安装方法同上,输入代码即可:
pip install keras
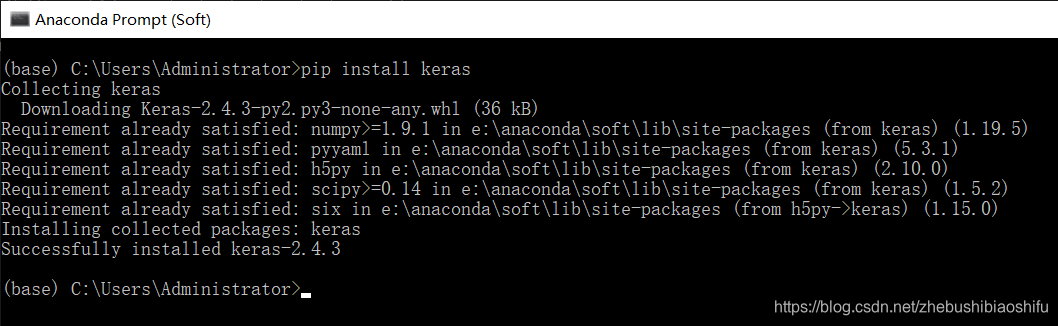
然后这里就不报错啦~
接下来,经过多次尝试发现,这一方法进行神经网络可视化时,一是不能存在正则化层与BatchNormalization层;二是LeakyReLU层与Dropout层的总数量不能过多,否则绘图结果会出现问题——这就显得这一可视化方法稍微有点鸡肋了,但是其对于基本的神经网络绘图而言其实也已经很不错了。因此,我就将Python TensorFlow深度神经网络回归:keras.Sequential中的神经网络上述对应的层删除或注释掉。
如下图,首先,将当初我的代码对应的LeakyReLU层与Dropout层注释掉:
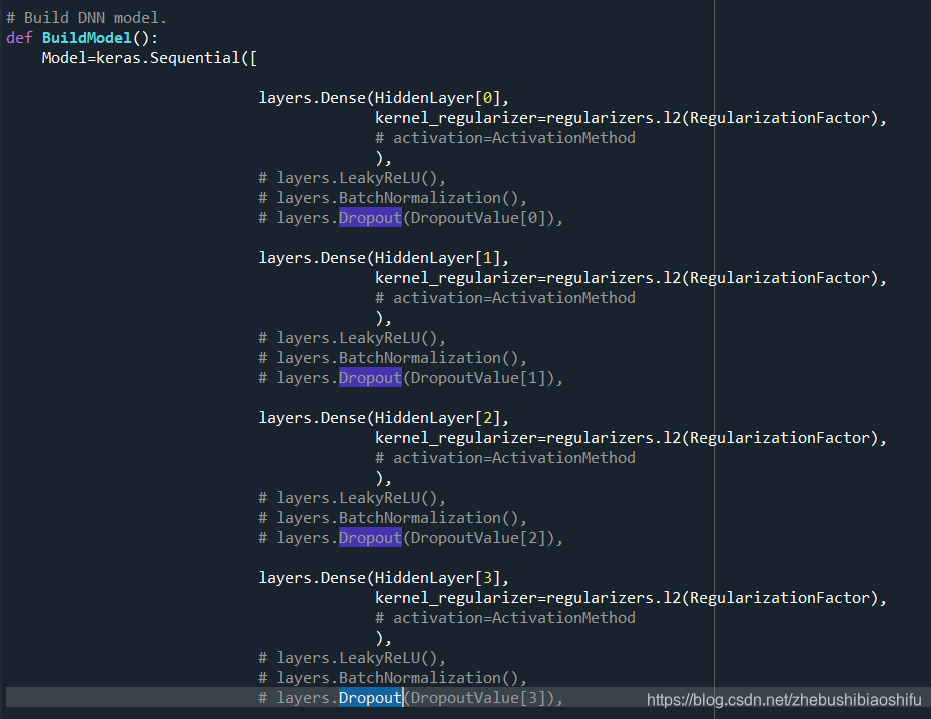
然后执行代码,即可进行神经网络的可视化。且绘制出的图将会自动打开在PDF阅读软件中,如下图(版面有限,这里就只是绘图结果的一部分)。
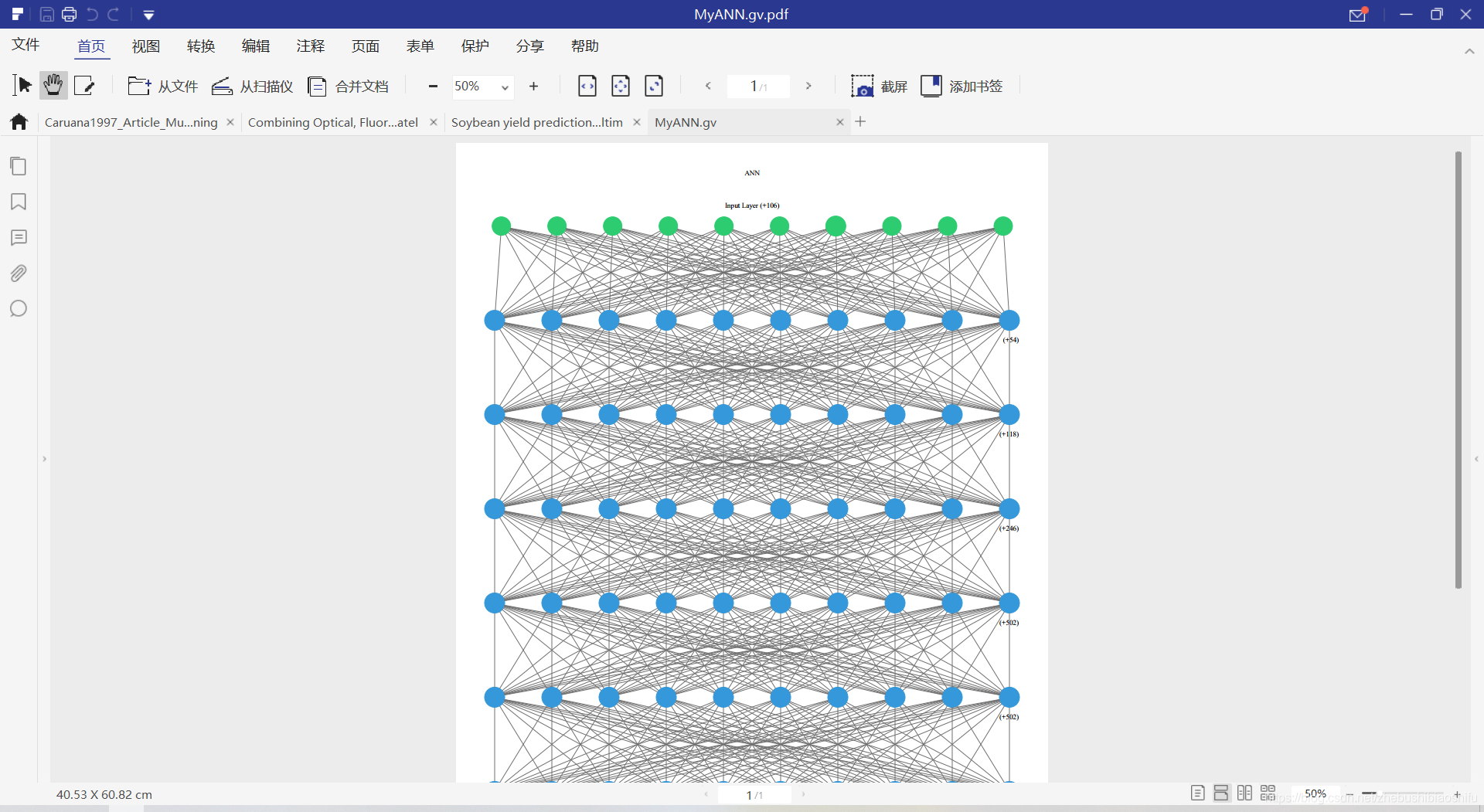
还是很不错的~我们还可以直接将其转换为图片格式,看起来就更直观了:

如果再取消Dropout层的注释,即绘图时加上Dropout层,也还是很不错的:
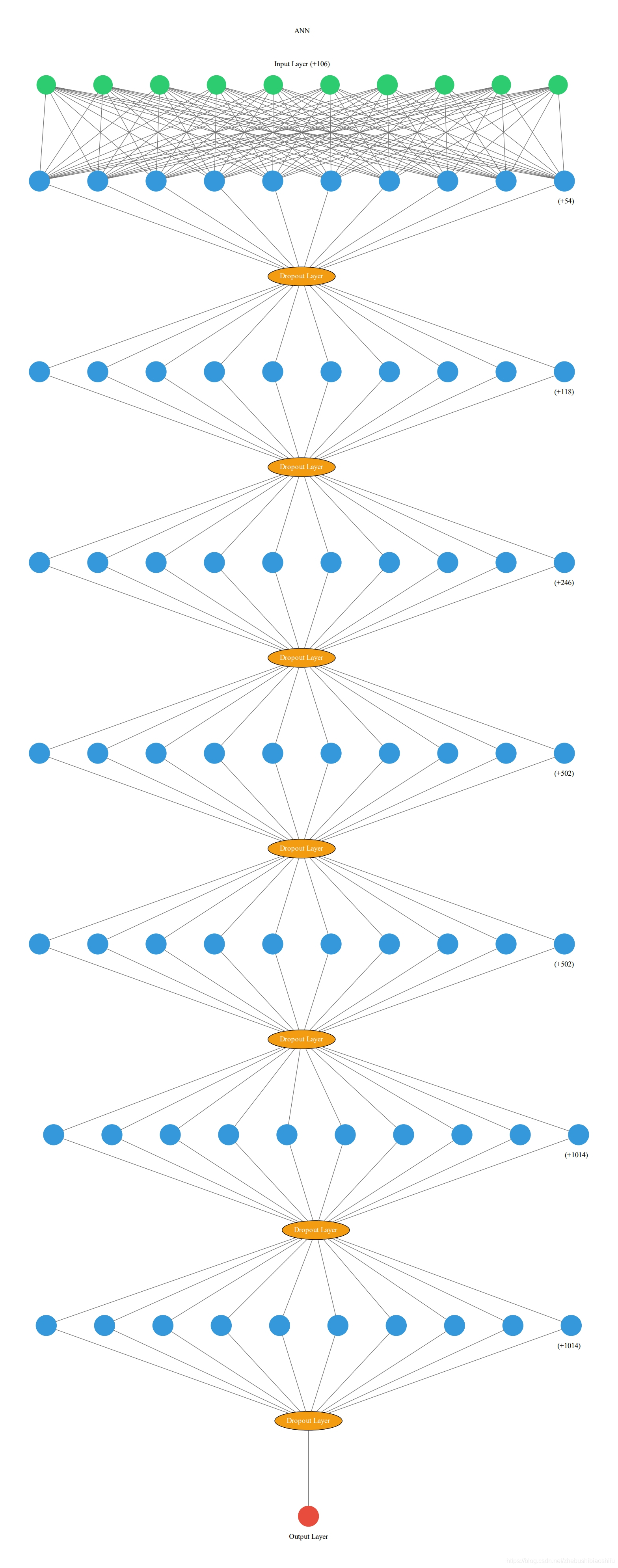
如果我们再加上LeakyReLU层,就成了这个乱七八糟、不太正确的样子(原图实在太大了,就只给大家截取图片的一部分):

可以看到,这样的话就有些问题了。
最后,我们看一下这个ann_visualizer第三方库的源代码,可以看到该库支持绘图的不同种类神经网络层;如果大家的神经网络包含这些层,就可以用ann_visualizer这一第三方库进行绘图。
至此,大功告成。



