- 1淘宝天猫1688通过图片搜索到相关产品API(拍立淘以图搜图API接口调用)_淘宝图片搜索apl
- 2必须干掉这10道,面试100%遇到!
- 36种方法实现css布局水平居中_gbk居中
- 4向量数据库之Lancedb学习记录_lancedberror: no vector column found to create ind
- 5【前端布局篇】响应式布局 Bootstrap 移动端布局_bootstrap 手机端ui
- 6卷积神经网络之父Yann LeCun 最新发声:自监督+世界模型,让 AI 像人类与一样学习与推理_神经网络进化出推理能力
- 7《系统集成项目管理工程师》知识点整理(持续更新)_系统项目集成管理师 知识点整理下载
- 8官宣!2024 MongoDB Developer Day来了!北上深三场等你集结!
- 9CSS实现垂直居中的6种办法_css垂直居中
- 10【重磅开源】MapleBoot权限控制使用介绍(菜单权限、按钮权限、数据权限)
使用TASSEL学习GWAS笔记(5/6):混合线性模型进行GWAS分析(MLM模型)_mlm模型运行时间很长吗
赞
踩
笔记计划分为六篇:
第一篇:读取plink基因型数据和表型数据
第二篇:对基因型数据质控:缺失质控,maf质控,hwe质控,样本质控
第三篇:基因型数据可视化:kingship,LD,MDS,PCA
第四篇:一般线性模型进行GWAS分析(GLM模型)
第五篇:混合线性模型进行GWAS分析(MLM模型)
第六篇:TASSEL结果可视化:QQ plot,曼哈顿图
已完成前四篇,本篇是第五篇。
1. 将质控的plink数据和表型数据读入到TASSEL软件
质控后的plink数据和表型数据:

读取表型数据到TASSEL中:
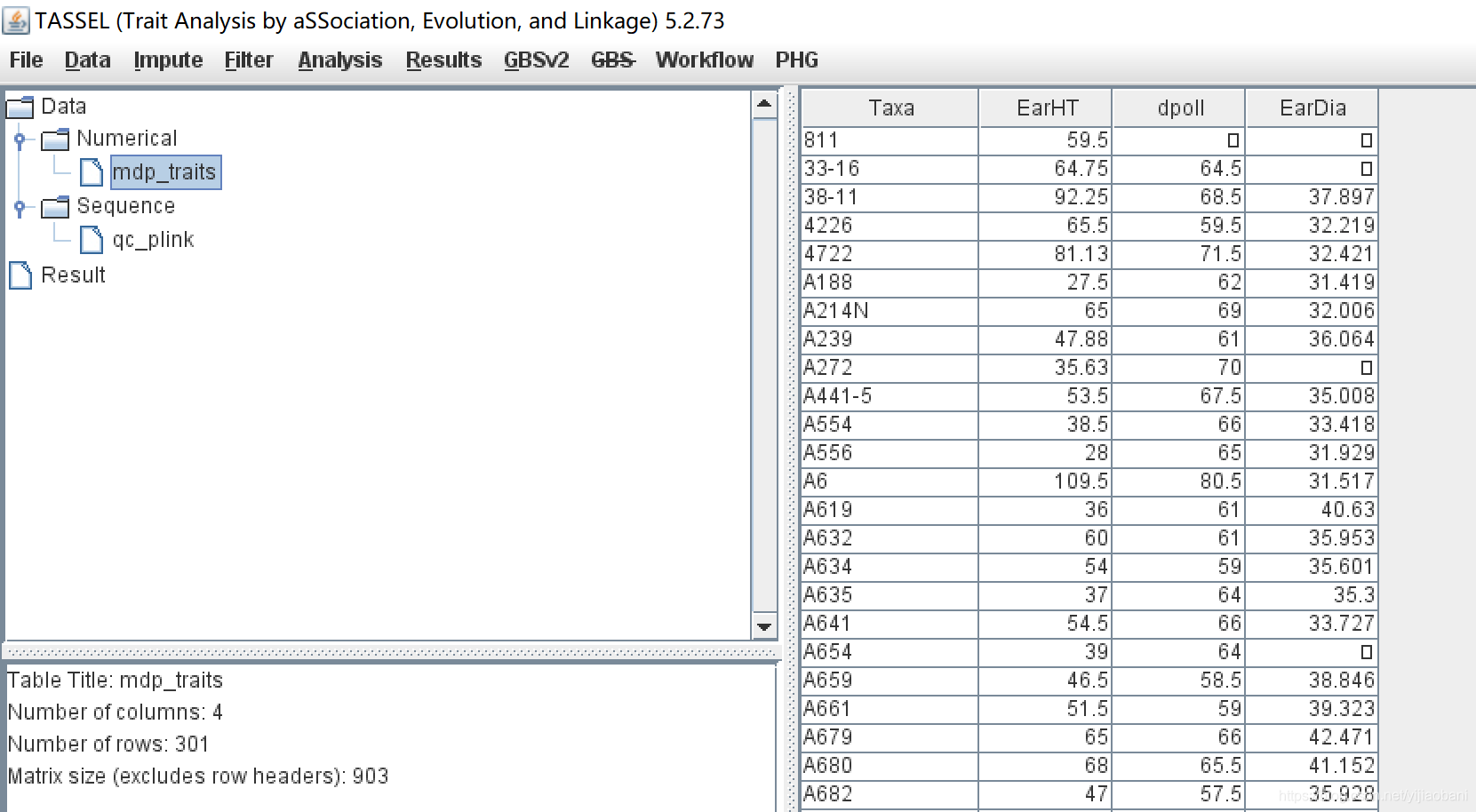
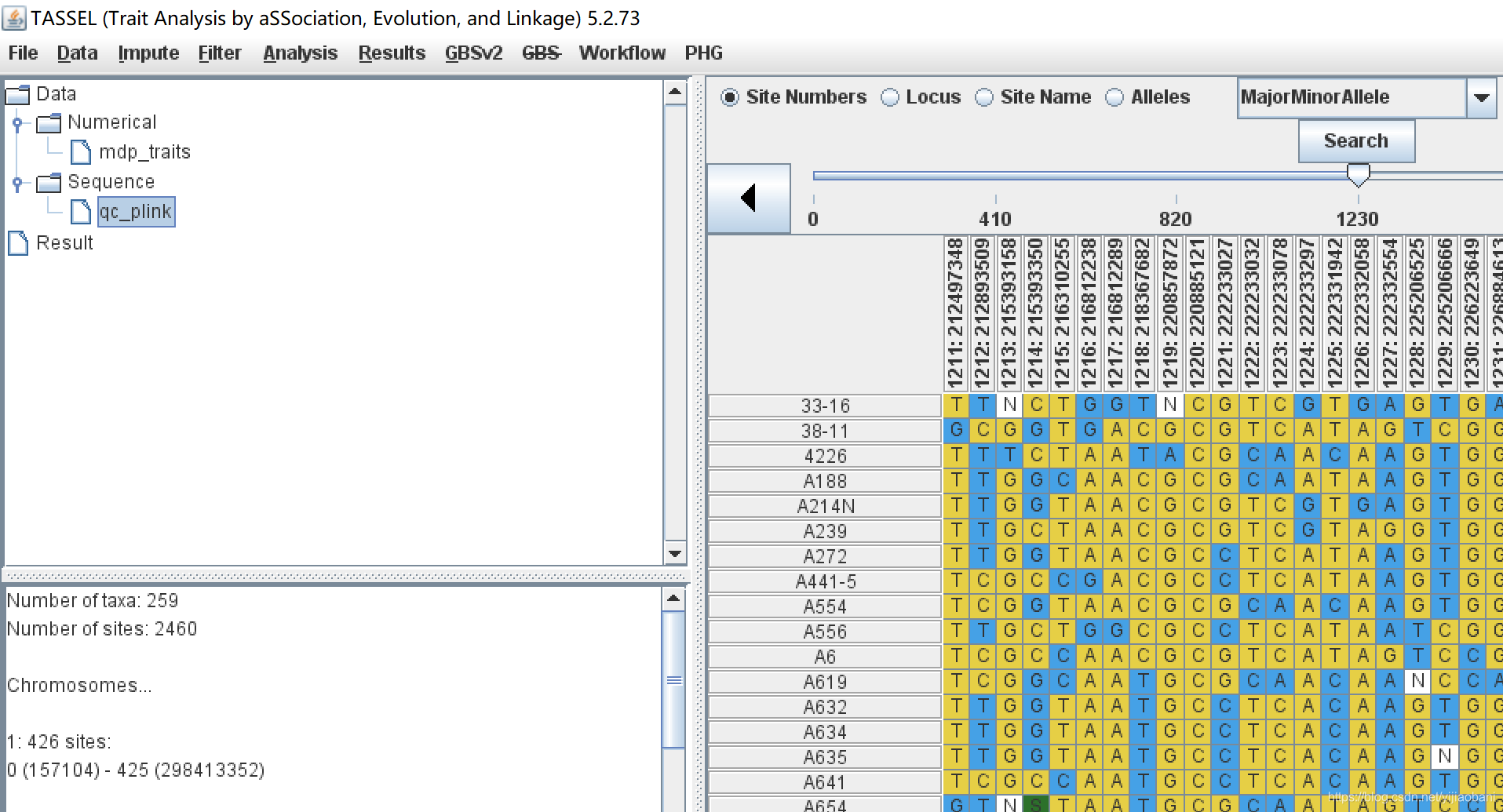
读取基因型数据到TASSEL中:
2. 一般线性模型(GLM)介绍

MLM模型中,将每个SNP作为固定因子进行回归分析,将亲缘关系矩阵(kinship或者A矩阵)作为随机因子,进行SNP的显著性检验,P值就是GWAS分析的p-value,effect就是SNP的effect值。如果有其它因素需要考虑,就放到协变量里面,比如性别,PCA,Q矩阵等。MLM和GLM不同的就是它考虑kinship的影响。
重点是对每个SNP做回归分析,提取effect和p-value。
3. 合并数据
TASSEL分析中,需要将分析的表型和基因型数据进行合并,合并为一个数据框,然后对该数据框进行分析。
3.1 对基因型数据进行PCA分析
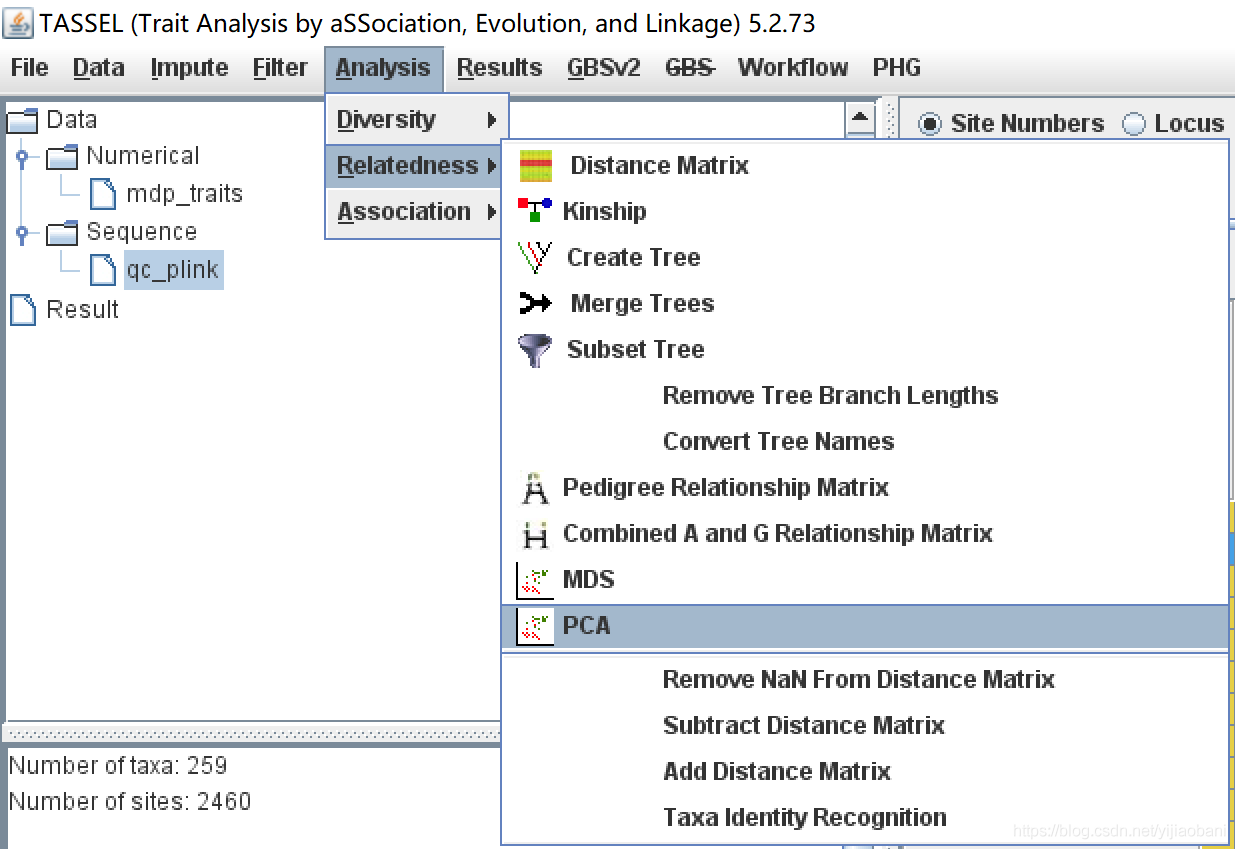
选中qc_plink基因型数据,点击菜单 Analysis --> Relatedness --> PCA,然后点击确定即可。
PCA分析结果:
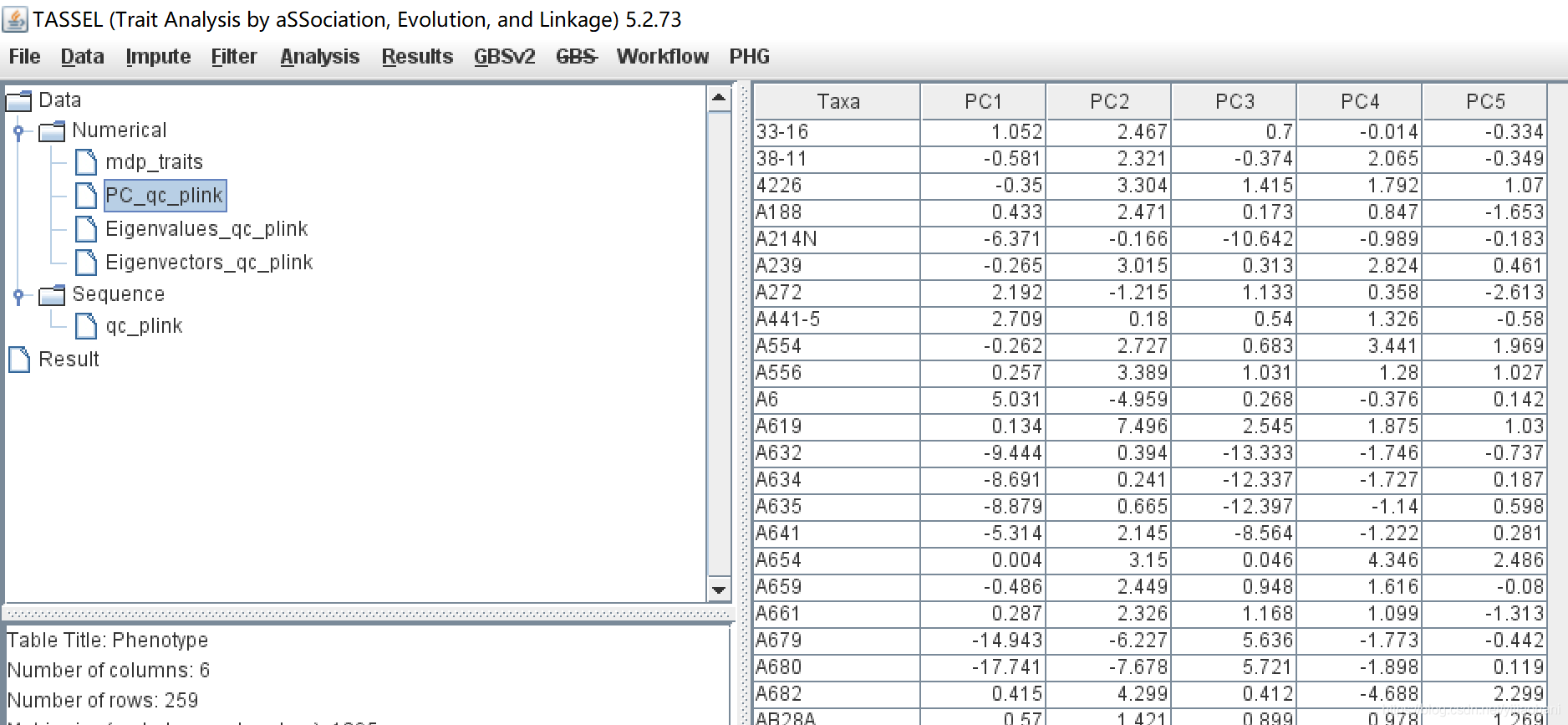
3.2 根据基因型数据计算kinship
选中基因型数据,点击菜单 Analysis --> Relatedness --> Kinship

默认选项即可:

查看kinship:

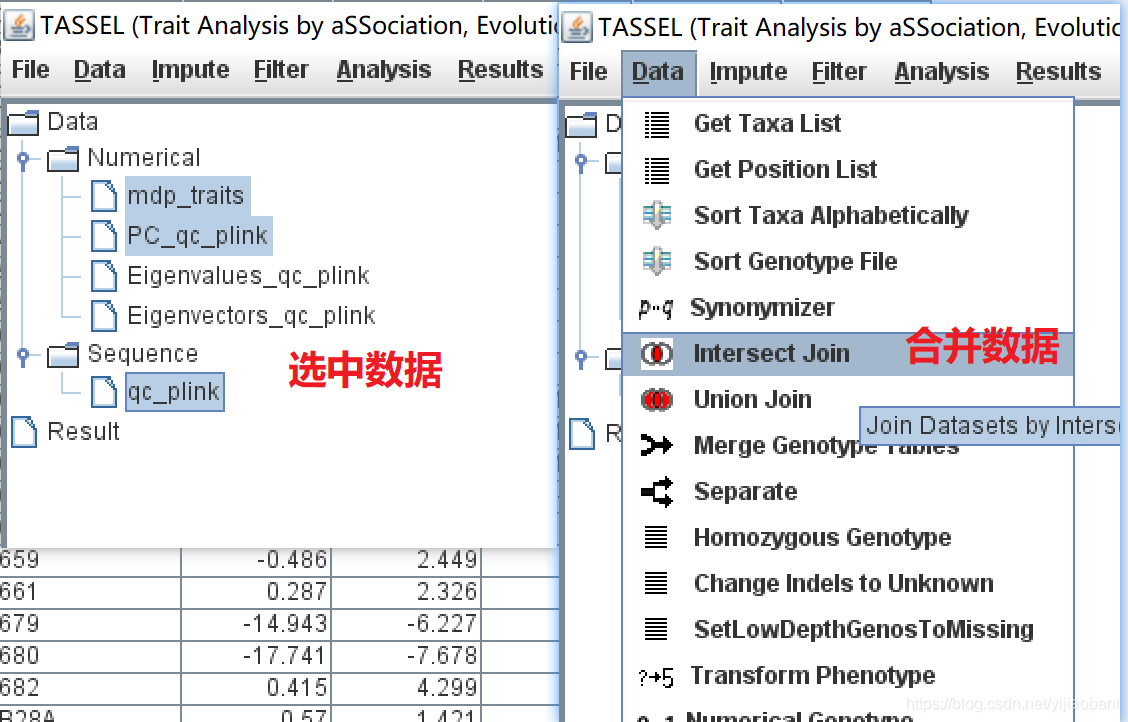
3.3 将PCA+表型+基因型合并
选中三个数据,然后点击Data中的Intersect Join,进行数据合并。注意,不要讲kinship放进去。
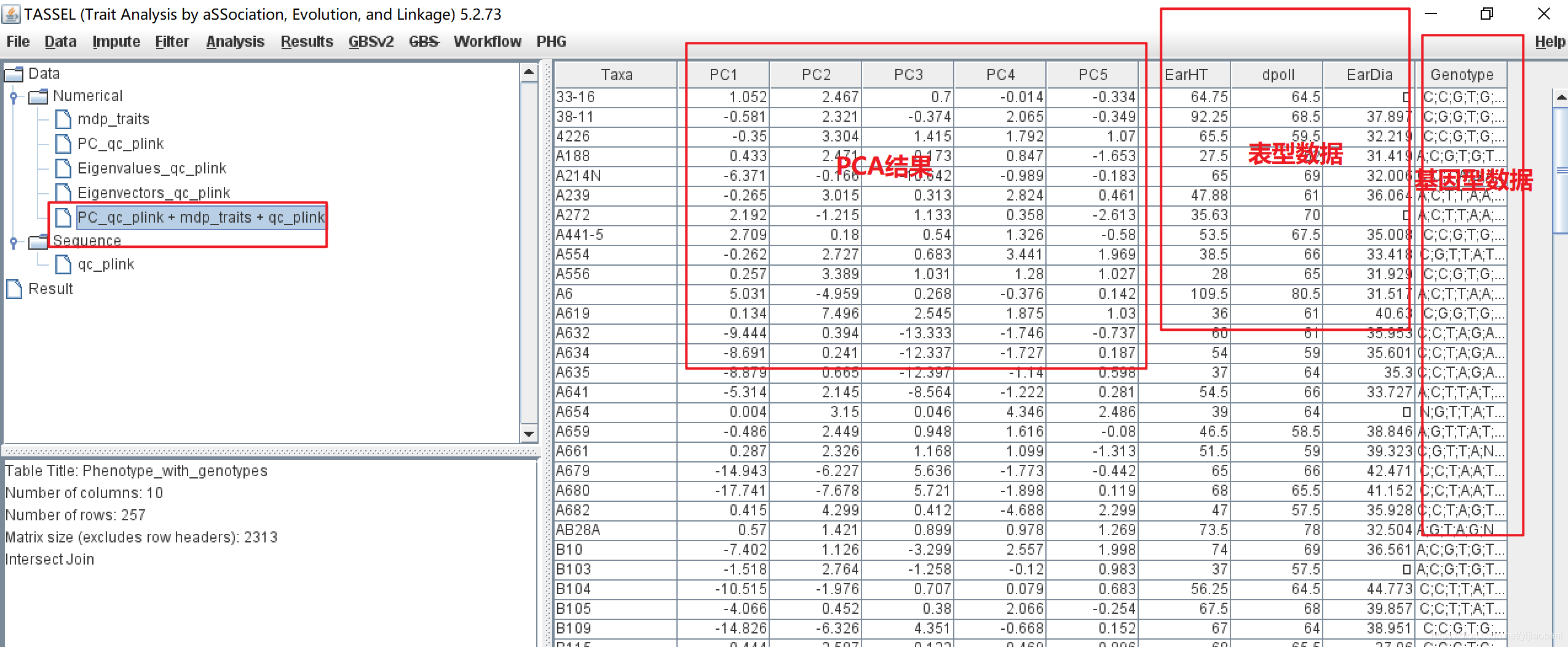
3.3 查看合并后的数据
可以看到,数据中包括ID,PCA及结果,表型性状数据,基因型数据。
4. MLM模型
选中合并后的数据 + kinship,点击Analysis --> Association --> MLM


点击默认Run运行即可。
5. MLM结果查看
可以看到,Result中有两个MLM结果,第一个为GWAS结果,第二个为每个SNP的效应值情况。看第一个就行。

因为这是多个性状的分析,所以所有结果放在了一起。
- 第一列为性状,这里包括三个性状,在进行作图时需要将数据分开
- 第二列为SNP名称
- 第三列为染色体名称
- 第四列为SNP的物理位置
- 第五列为df
- 第六列为F检验结果
- 第七列为p值
- ……
6. 导出结果


查看结果:

7. TASSEL中的结果可视化
QQ图:



曼哈顿图:


这里,曼哈顿图需要指定性状,这里我们选择EarDia这个性状进行可视化:

图片可以保存到本地。
ok,第五篇搞定了。下一篇是GWAS结果放到R语言中可视化,欢迎继续关注。
欢迎关注我的公众号:
育种数据分析之放飞自我。主要分享R语言,Python,育种数据分析,生物统计,数量遗传学,混合线性模型,GWAS和GS相关的知识。


