热门标签
热门文章
- 1重定向session失效怎么解决_接口幂等性解决方案
- 2Xcode添加库文件framework (转)_ios动态库framework在沙盒加载失败
- 34.fastapi的路径参数和查询参数的数据解析、验证_fastapi apirouter参数
- 4什么是边缘计算平台?
- 5LPA算法原理_lpa线性检测原理
- 6低端PC配置Android虚拟机_虚拟机安卓低版本
- 7【matplotlib】1-使用函数绘制图表_用matplotlib库绘制函数的图形
- 8python 绘制传感器数据的时频图_python如何绘制时频图
- 9AttributeError: ‘EnvSpec‘ object has no attribute ‘_kwargs‘ 解决方案_attributeerror: 'envspec' object has no attribute
- 10docker启动卡死问题排查
当前位置: article > 正文
msigdbr函数的一些见解
作者:AllinToyou | 2024-03-29 03:33:18
赞
踩
msigdbr
msigdbr函数的一些见解
对这个函数有问题主要是因为msigdb数据库出现鼠系列的数据,那我就想能不能用这些数据来做个GSEA或GSVA。但关于通路的数据是WikiPathways的。

那么用msigdb数据库来做GSVA/GSEA分析是就只能用C2 C6的数据。紧接着我看到一段话
我就开始怀疑之前做的会不会是错的,因为我之前没有转换的这一步,直接将鼠基因导入msigdbr函数,利用C2做的kegg富集分析。
所以我开始去观察这个函数的参数
KEGG_df_all <- msigdbr(species = "Mus musculus", # Homo sapiens or Mus musculus
category = "C2",
subcategory = "CP:KEGG")
- 1
- 2
- 3
可以看到这个函数是可以选择物种
那是不是这个函数会自动将基因转化为人基因呢,这样最终得到的结果才算有意义。
这样一来就得观察函数的源代码了。源代码没找到,最终想到一种方法来验证。
只改变species这个参数,观察结果。
library(msigdbr)
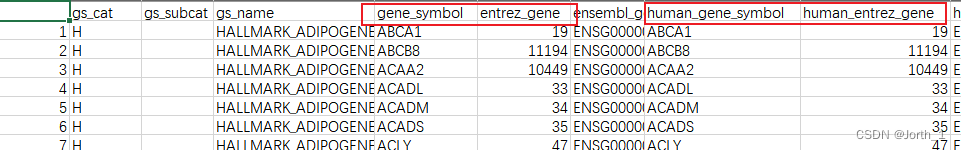
h_gene_sets <- msigdbr(species = "Homo sapiens", category = "H")
head(h_gene_sets)
- 1
- 2
- 3
library(msigdbr)
h_gene_sets <- msigdbr(species = "mouse", category = "H")
head(h_gene_sets)
- 1
- 2
- 3
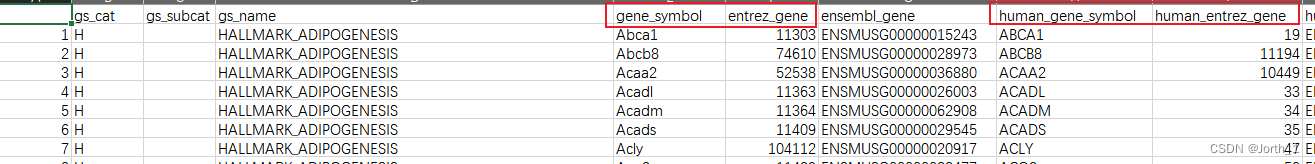
观察一下就能发现,gene_symbol为自动转化为人的基因,所以最终利用C2来做富集分析时可以的。
不过毕竟是人基因相关的通路,最终得到的结果肯定没有在鼠相关的通路说服力更强。
我的问题又来了,之前利用kegg数据库做的富集分析是不是也是利用人基因呢?
GSEA第3弹!MSigDB也开始支持小鼠功能基因集啦 - 知乎 (zhihu.com)
GSEA | MSigDB (gsea-msigdb.org)
(36条消息) wikipathway : 代谢通路专用数据库_生信修炼手册的博客-CSDN博客
WikiPathways - WikiPathways
声明:本文内容由网友自发贡献,不代表【wpsshop博客】立场,版权归原作者所有,本站不承担相应法律责任。如您发现有侵权的内容,请联系我们。转载请注明出处:https://www.wpsshop.cn/w/AllinToyou/article/detail/333490
推荐阅读
相关标签


