- 1FPGA实现 TCP/IP 协议栈 服务器 纯VHDL代码编写 提供4套vivado工程源码和技术支持_9527华安 csdn
- 2Git操作 --忽略文件_git忽略文件
- 3基于STM32F103ZET6单片机智能服药药盒开关语音无线设计23-070_stm32f103zet6闹钟
- 4java企业员工管理系统_员工管理系统idea
- 5MATLAB算法实战应用案例精讲-【自动驾驶】路径规划_matlab 路径规划
- 6Opencv | 基于ndarray的基本操作
- 7Docker overview_docker start --checkpoint looper2 looper2
- 8解决maven构建时警告:The artifact xxx has been relocated to xxx_the artifact mysql:mysql-connector-java:jar:8.0.33
- 9头歌educoder-Python程序设计-第四阶段 函数与模块-模块_要求判断是否存在两个整数,它们的和为a,积为b
- 10Android Camera开发入门(4):USB/UVC Camera的使用_android usb camera
DBSCAN聚类︱scikit-learn中一种基于密度的聚类方式
赞
踩
一、DBSCAN聚类概述
基于密度的方法的特点是不依赖于距离,而是依赖于密度,从而克服基于距离的算法只能发现“球形”聚簇的缺点。
DBSCAN的核心思想是从某个核心点出发,不断向密度可达的区域扩张,从而得到一个包含核心点和边界点的最大化区域,区域中任意两点密度相连。
1、伪代码
算法: DBSCAN
输入: E — 半径
MinPts — 给定点在 E 领域内成为核心对象的最小领域点数
D — 集合
输出:目标类簇集合
方法: repeat
-
判断输入点是否为核心对象- 1
-
找出核心对象的 E 领域中的所有直接密度可达点- 1
util 所有输入点都判断完毕
repeat
针对所有核心对象的 E 领域所有直接密度可达点找到最大密度相连对象集合,
中间涉及到一些密度可达对象的合并。
Util 所有核心对象的 E 领域都遍历完毕密度:空间中任意一点的密度是以该点为圆心,以EPS为半径的圆区域内包含的点数目
边界点:空间中某一点的密度,如果小于某一点给定的阈值minpts,则称为边界点
噪声点:不属于核心点,也不属于边界点的点,也就是密度为1的点
2、优点:
- 这类算法能克服基于距离的算法只能发现“类圆形”(凸)的聚类的缺点
- 可发现任意形状的聚类,且对噪声数据不敏感。
- 不需要指定类的数目cluster
- 算法中只有两个参数,扫描半径 (eps)和最小包含点数(min_samples)
3、缺点:
- 1、计算复杂度,不进行任何优化时,算法的时间复杂度是O(N^{2}),通常可利用R-tree,k-d tree, ball
tree索引来加速计算,将算法的时间复杂度降为O(Nlog(N))。 - 2、受eps影响较大。在类中的数据分布密度不均匀时,eps较小时,密度小的cluster会被划分成多个性质相似的cluster;eps较大时,会使得距离较近且密度较大的cluster被合并成一个cluster。在高维数据时,因为维数灾难问题,eps的选取比较困难。
- 3、依赖距离公式的选取,由于维度灾害,距离的度量标准不重要
- 4、不适合数据集集中密度差异很大的,因为eps和metric选取很困难
4、与其他聚类算法比较
来看两张图:
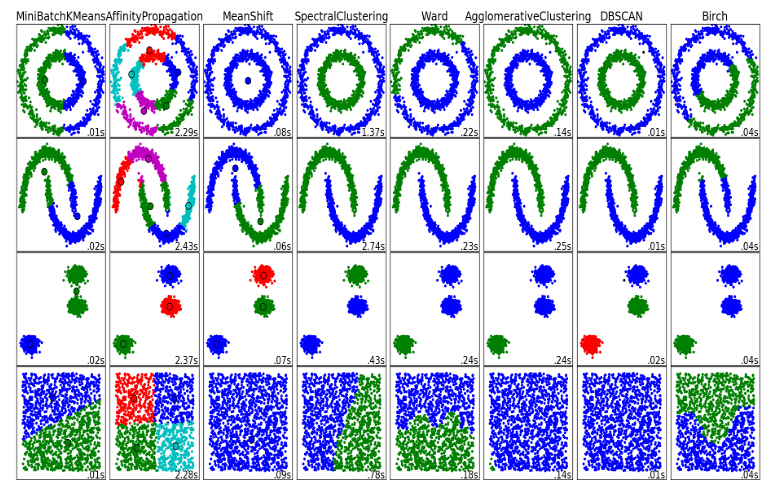
DBSCAN可以较快、较有效的聚类出来
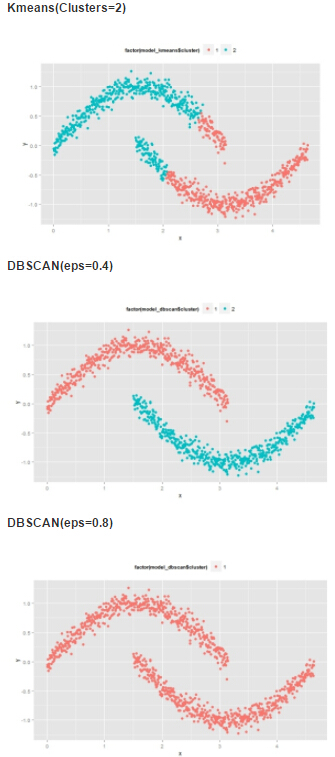
eps的取值对聚类效果的影响很大。
.
二、sklearn中的DBSCAN聚类算法
1、主要函数介绍:
DBSCAN(eps=0.5, min_samples=5, metric='euclidean', algorithm='auto', leaf_size=30, p=None, n_jobs=1)
- 1
最重要的两个参数:
eps:两个样本之间的最大距离,即扫描半径
min_samples :作为核心点的话邻域(即以其为圆心,eps为半径的圆,含圆上的点)中的最小样本数(包括点本身)。
其他参数:
metric :度量方式,默认为欧式距离,还有metric=‘precomputed’(稀疏半径邻域图)
algorithm:近邻算法求解方式,有四种:‘auto’, ‘ball_tree’, ‘kd_tree’, ‘brute’
leaf_size:叶的大小,在使用BallTree or cKDTree近邻算法时候会需要这个参数
n_jobs :使用CPU格式,-1代表全开
其他主要属性:
core_sample_indices_:核心样本指数。(此参数在代码中有详细的解释)
labels_:数据集中每个点的集合标签给,噪声点标签为-1。
components_ :核心样本的副本
运行式子:
model = sklearn.cluster.DBSCAN(eps_领域大小圆半径,min_samples_领域内,点的个数的阈值)
model.fit(data) 训练模型
model.fit_predict(data) 模型的预测方法
.
2、DBSCAN自编代码
import numpy import pandas import matplotlib.pyplot as plt #导入数据 data = pandas.read_csv("F:\\python 数据挖掘分析实战\\Data\\data (7).csv") plt.scatter( data['x'], data['y'] ) eps = 0.2; MinPts = 5; from sklearn.metrics.pairwise import euclidean_distances ptses = [] dist = euclidean_distances(data) for row in dist: #密度,空间中任意一点的密度是以该点为圆心、以 Eps 为半径的圆区域内包含的点数 density = numpy.sum(row<eps) pts = 0; if density>MinPts: #核心点(Core Points) #空间中某一点的密度,如果大于某一给定阈值MinPts,则称该为核心点 pts = 1 elif density>1 : #边界点(Border Points) #空间中某一点的密度,如果小于某一给定阈值MinPts,则称该为边界点 pts = 2 else: #噪声点(Noise Points) #数据集中不属于核心点,也不属于边界点的点,也就是密度值为1的点 pts = 0 ptses.append(pts) #把噪声点过滤掉,因为噪声点无法聚类,它们独自一类 corePoints = data[pandas.Series(ptses)!=0] coreDist = euclidean_distances(corePoints) #首先,把每个点的领域都作为一类 #邻域(Neighborhood) #空间中任意一点的邻域是以该点为圆心、以 Eps 为半径的圆区域内包含的点集合 cluster = dict(); i = 0; for row in coreDist: cluster[i] = numpy.where(row<eps)[0] i = i + 1 #然后,将有交集的领域,都合并为新的领域 for i in range(len(cluster)): for j in range(len(cluster)): if len(set(cluster[j]) & set(cluster[i]))>0 and i!=j: cluster[i] = list(set(cluster[i]) | set(cluster[j])) cluster[j] = list(); #最后,找出独立(也就是没有交集)的领域,就是我们最后的聚类的结果了 result = dict(); j = 0 for i in range(len(cluster)): if len(cluster[i])>0: result[j] = cluster[i] j = j + 1 #找出每个点所在领域的序号,作为他们最后聚类的结果标记 for i in range(len(result)): for j in result[i]: data.at[j, 'type'] = i plt.scatter( data['x'], data['y'], c=data['type'] )
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
- 54
- 55
- 56
- 57
- 58
- 59
- 60
- 61
- 62
- 63
- 64
- 65
- 66
- 67
- 68
- 69
- 70
- 71
- 72
- 73
- 74
- 75
- 76
3、实战案例:
# DBSCAN clustering algorithm print(__doc__) import numpy as np from sklearn.cluster import DBSCAN from sklearn import metrics from sklearn.datasets.samples_generator import make_blobs from sklearn.preprocessing import StandardScaler # Generate sample data centers = [[1, 1], [-1, -1], [1, -1]] X, labels_true = make_blobs(n_samples=750, centers=centers, cluster_std=0.4, random_state=0) X = StandardScaler().fit_transform(X) # Compute DBSCAN db = DBSCAN(eps=0.1, min_samples=10).fit(X) core_samples_mask = np.zeros_like(db.labels_, dtype=bool) core_samples_mask[db.core_sample_indices_] = True labels = db.labels_ # Number of clusters in labels, ignoring noise if present. n_clusters_ = len(set(labels)) - (1 if -1 in labels else 0) print('Estimated number of clusters: %d' % n_clusters_) print("Homogeneity: %0.3f" % metrics.homogeneity_score(labels_true, labels)) print("Completeness: %0.3f" % metrics.completeness_score(labels_true, labels)) print("V-measure: %0.3f" % metrics.v_measure_score(labels_true, labels)) print("Adjusted Rand Index: %0.3f" % metrics.adjusted_rand_score(labels_true, labels)) print("Adjusted Mutual Information: %0.3f" % metrics.adjusted_mutual_info_score(labels_true, labels)) print("Silhouette Coefficient: %0.3f" % metrics.silhouette_score(X, labels)) # import matplotlib.pyplot as plt # Black removed and is used for noise instead. unique_labels = set(labels) colors = [plt.cm.Spectral(each) for each in np.linspace(0, 1, len(unique_labels))] for k, col in zip(unique_labels, colors): if k == -1: # Black used for noise. col = [0, 0, 0, 1] class_member_mask = (labels == k) xy = X[class_member_mask & core_samples_mask] plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=tuple(col), markeredgecolor='k', markersize=14) xy = X[class_member_mask & ~core_samples_mask] plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=tuple(col), markeredgecolor='k', markersize=6) plt.title('Estimated number of clusters: %d' % n_clusters_) plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
- 54
- 55
- 56
- 57
- 58
- 59
- 60
- 61
- 62
- 63
- 64
最后的结果:
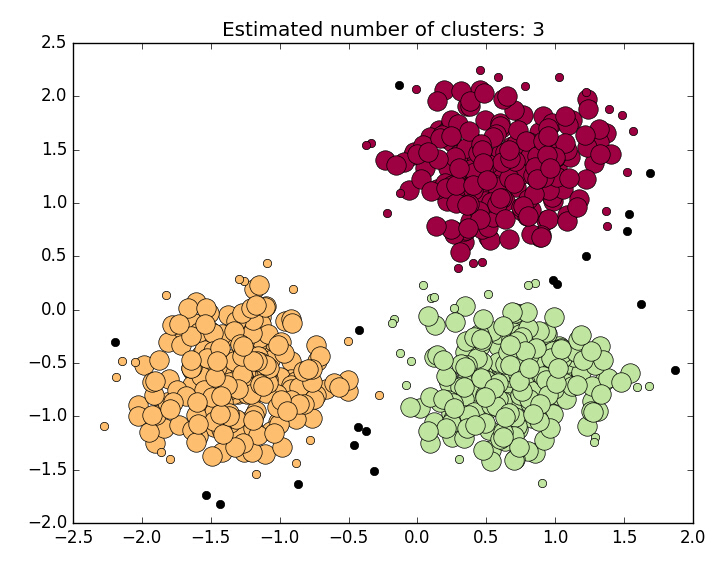
.
延伸一:DPEAK算法——密度最大值算法
本节来源:机器学习笔记(九)聚类算法及实践(K-Means,DBSCAN,DPEAK,Spectral_Clustering)、聚类 - 4 - 层次聚类、密度聚类(DBSCAN算法、密度最大值聚类)
密度最大值聚类是一种简洁优美的聚类算法, 可以识别各种形状的类簇, 并且参数很容易确定。用于找聚类中心和异常值的。
用DPEAK算法找到聚类中心之后,在用DBSCAN会更好
(1)我们首先给定一个半径范围r,然后对我们所有的样本,计算它的r邻域内的样本数目记作它的局部密度记作rho
(2)第二步,计算每个样本到密度比它高的点的距离的最小值记作sigma,有了这两个参数就可以进行我们下一步的筛选工作了
具体分成以下四种情况:
1 rho很小,sigma很大。这个样本周围的样本量很小,但是到比它密度大的点的距离还挺远的,这说明啥,它是个远离正常样本的异常值啊,在偏僻的小角落里搞自己的小动作啊,果断踢了它呀。
2 rho很大,sigma也很大。这个样本周围样本量很大,并且要找到比它密度还大的点要好远好远,这说明这个点是被众星环绕的啊,它就是这个簇的王,我们往往把它确定为簇中心。
3 rho很小,sigma也很小。样本周围的样本量很小,但要找到样本密度比它大的点没多远就有,说明这个点是一个处在边缘上的点,往往是一个簇的边界。
4 rho很大,sigma很小。该样本周围的样本量很大,但是密度比它还大的居然也不远,这种情况只会发生在你处在了簇中心的旁边时,很可惜,也许你是这个簇的核心成员,但你做不了这个簇的王。
好的,基于每个样本的rho和sigma,我们大概就能确定它们各自的所扮演的角色了,我们把大反派异常值从样本中剔除,然后把我们找到的rho和sigma都很大的点作为簇中心,再利用K-Means或者DBSCAN算法进行聚类就能得到相对比较好的结果。
延伸二:利用GPS轨迹和DBSCAN推断工作地居住地
来自文章《利用GPS轨迹和DBSCAN推断工作地居住地》
利用移动设备用户的GPS轨迹可以推断用户的工作地和居住地。本文中的数据集来自微软亚洲研究院的GeoLife GPS轨迹数据集
利用DBSCAN(Density-Based Spatial Clustering and Application with Noise)算法对数据集进行聚类。DBSCAN是一种聚类算法,这种算法通常用于对带有噪声的空间数据进行聚类。利用DBSCAN算法可以对GPS轨迹聚成4类。直观上来看,每一个聚类都表示该用户经常到访该区域。因此,可以假设用户的工作地和居住地就在这4个聚类中。
延伸三:HDBSCAN与Kmeans的聚类的一些纪要
HDBSCAN的安装与使用
安装的两种办法:
conda install -c conda-forge hdbscan # first
pip install hdbscan # second
- 1
- 2
应用:
import hdbscan
from sklearn.datasets import make_blobs
data, _ = make_blobs(1000)
clusterer = hdbscan.RobustSingleLinkage(cut=0.125, k=7)
cluster_labels = clusterer.fit_predict(data)
hierarchy = clusterer.cluster_hierarchy_
alt_labels = hierarchy.get_clusters(0.100, 5)
hierarchy.plot()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
DBSCAN vs HDBSCAN
HDBSCAN - Hierarchical Density-Based Spatial Clustering of Applications with Noise. Performs DBSCAN over varying epsilon values and integrates the result to find a clustering that gives the best stability over epsilon. This allows HDBSCAN to find clusters of varying densities (unlike DBSCAN), and be more robust to parameter selection.
如果输入数据的变量类型不同,部分是数值型(numerical),部分是分类变量(categorical),需要做特别处理。
方法1是将分类变量转化为数值型,但缺点在于如果使用独热编码(one hot encoding)可能会导致数据维度大幅度上升,如果使用标签编码(label encoding)无法很好的处理数据中的顺序(order)。方法2是对于数值型变量和分类变量分开处理,并将结果结合起来,具体可以参考Python的实现[1],如K-mode和K-prototype。
输出结果非固定,多次运行结果可能不同。
首先要意识到K-means中是有随机性的,从初始化到收敛结果往往不同。一种看法是强行固定随机性,比如设定sklearn中的random state为固定值。另一种看法是,如果你的K均值结果总在大幅度变化,比如不同簇中的数据量在多次运行中变化很大,那么K均值不适合你的数据,不要试图稳定结果 [2]
运行效率与性能之间的取舍。
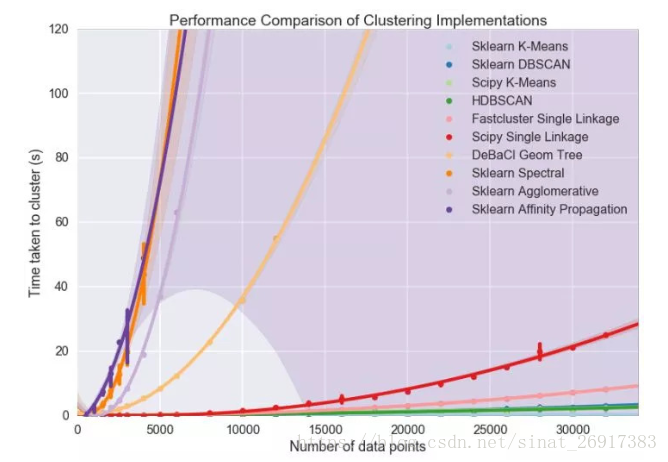
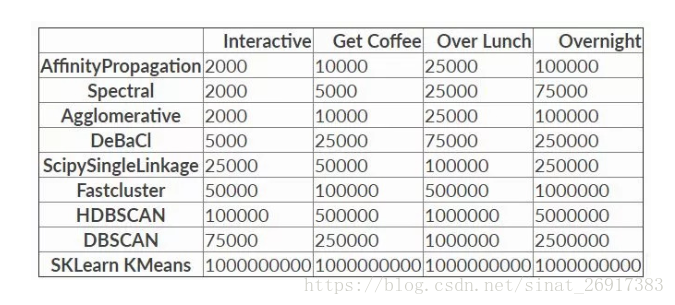
但数据量上升到一定程度时,如>10万条数据,那么很多算法都不能使用。最近读到的一篇对比不同算法性能随数据量的变化很有意思 [Benchmarking Performance and Scaling of Python Clustering Algorithms]。在作者的数据集上,当数据量超过一定程度时仅K均值和HDBSCAN可用。
因此不难看出,K均值算法最大的优点就是运行速度快,能够处理的数据量大,且易于理解。但缺点也很明显,就是算法性能有限,在高维上可能不是最佳选项。
一个比较粗浅的结论是,在数据量不大时,可以优先尝试其他算法。当数据量过大时,可以试试HDBSCAN。仅当数据量巨大,且无法降维或者降低数量时,再尝试使用K均值。
一个显著的问题信号是,如果多次运行K均值的结果都有很大差异,那么有很高的概率K均值不适合当前数据,要对结果谨慎的分析。
此外无监督聚类的评估往往不易,基本都是基于使用者的主观设计,如sklearn中提供的Silhouette Coefficient和 Calinski-Harabaz Index [5]。更多关于无监督学习如何评估可以参考 [微调:一个无监督学习算法,如何判断其好坏呢?]。
HDBSCAN案例
文档地址:https://hdbscan.readthedocs.io/en/latest/basic_hdbscan.html#
from sklearn.datasets import make_blobs import pandas as pd blobs, labels = make_blobs(n_samples=2000, n_features=10) import hdbscan clusterer = hdbscan.HDBSCAN() clusterer.fit(blobs) # 分组 clusterer.labels_ >>> array([2, 2, 2, ..., 2, 2, 0]) # 最大分组是什么 clusterer.labels_.max() >>>2 # 每组概率得分 clusterer.probabilities_ >>> array([ 0.83890858, 1. , 0.72629904, ..., 0.79456452, 0.65311137, 0.76382928])
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
分组为-1则代表为噪声点,不在某个群落之中,
这个概率得分说一下,代表集群中每个点的是该集群成员资格的强度:
- 如果是为0,代表,肯定不在这组里面,异常值、噪声值一般打分都为0
- 代表1,代表是所在群的中心位置
分类散点图:
import numpy as np import matplotlib.pyplot as plt import seaborn as sns import sklearn.datasets as data %matplotlib inline sns.set_context('poster') sns.set_style('white') sns.set_color_codes() plot_kwds = {'alpha' : 0.5, 's' : 80, 'linewidths':0} moons, _ = data.make_moons(n_samples=50, noise=0.05) blobs, _ = data.make_blobs(n_samples=50, centers=[(-0.75,2.25), (1.0, 2.0)], cluster_std=0.25) test_data = np.vstack([moons, blobs]) plt.scatter(test_data.T[0], test_data.T[1], color='b', **plot_kwds) import hdbscan clusterer = hdbscan.HDBSCAN(min_cluster_size=5, gen_min_span_tree=True) clusterer.fit(test_data) palette = sns.color_palette() cluster_colors = [sns.desaturate(palette[col], sat) if col >= 0 else (0.5, 0.5, 0.5) for col, sat in zip(clusterer.labels_, clusterer.probabilities_)] plt.scatter(test_data.T[0], test_data.T[1], c=cluster_colors, **plot_kwds)
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
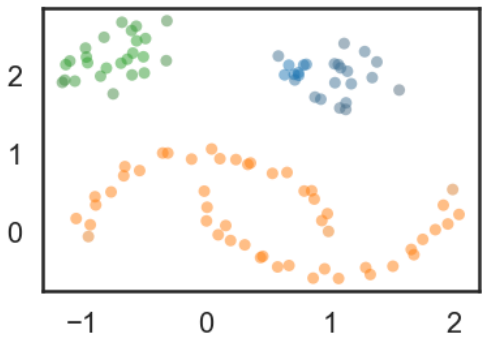
参考来源
聚类分析(五)基于密度的聚类算法 — DBSCAN
聚类算法第三篇-密度聚类算法DBSCAN
聚类算法初探(五)DBSCAN,作者: peghoty
聚类算法第一篇-概览
sklearn.cluster.DBSCAN
【挖掘模型】:Python-DBSCAN算法