EEG&CNN深度学习神经网络前置:处理EEG脑电信号生成时频图dataset_eeg一维信号转换成时频图
赞
踩
2023/3/9 -3/11 脑机接口学习内容一览:
前段时间做了比较多的提取频谱特征来进行机器学习,进而完成模式识别的任务。在这这一篇博客中,将尝试使用EEG信号,主要使用plt.specgram()函数来生成脑电信号的时频图dataset,为接下来使用神经网络进行深度学习做准备。
一、需求分析:
在本次工作开始之前,我们先对本次的尝试进行一次简单的需求分析。
(在参加服务外包期间因为没有做好这些基础工作吃了很大的亏,不得不频繁修改代码)
1.数据集
本次尝试采用的数据集仍然与往期博文对脑电信号进行特征提取并分类(二分类)相同,采用的是matlab脑电处理工具EEGlab的自带数据集。数据集中包括两种刺激rt与square,对于我们的工作来说是一个较为简单的二分类问题。
2.输出数据
根据参考资料【1】中对脑电数据源的要求,可知卷积核面向的脑电数据尽量具有图像结构,并且为一个三维张量,包括三个维度即时间,频率,频道。根据参考资料【4】中dataset讲解部分分析可得,可能需要输出的图片结构如图所示:

除此之外,我们根据参考资料【3】的建议,将生成图片以224像素大小的png格式输出。
二、处理流程
在这个环节,我们事先敲定一下处理的流程,避免出现想到哪里做到哪里这种没有规划的情况。
1.导入数据集;
2.使用之前预处理所用函数过一遍预处理,参考对脑电信号进行特征提取并分类(二分类)中的transform( )函数;
3.遍历训练集和测试集中的epochs片段,根据标签和频道来生成对应的时频图并保存到指定路径中。
三、完整代码
这次的代码时间较为简单。花那么长时间的主要原因在于需要调查适合于投入CNN网络的数据集格式与回想复习之前所学习的神经网络知识。一开始对mne不熟悉,没有立刻想起epochs的采样点具体数据是由get_data()得到的,也花费了不少查询时间。
需要注意的是程序生成图片的数量较多(约4000~5000张),所以跑的时间比较长,我运行了大概四个小时左右。
- import os
- import matplotlib.pyplot as plt
- import mne
-
-
- def transform(raw, event_id):
- # 有别于之前繁琐的预处理,这里尽量少采用预处理,保持EEG数据的完整性
- events, _ = mne.events_from_annotations(raw, event_id=event_id, chunk_duration=None)
- '''
- 根据事件创建epochs
- 在这个部分,提取epochs的各项参数需要自己仔细确定
- 比如tmin和tmax这两个值,可以通过raw的plot图像来大致确定
- picks这里选取的是eeg通道
- 同时使用了基线校正
- '''
- tmin, tmax = -0.2, 0.3
- epochs = mne.Epochs(raw=raw, events=events, event_id=event_id, tmin=tmin,
- tmax=tmax, baseline=(None, 0), preload=True, reject_tmax=0, reject_by_annotation=False)
- epochs_data = epochs.get_data()
- """
- print(epochs.get_data().shape)
- print(events.shape)
- (65, 32, 65)
- (65, 3)
- get_data()方法将带epoch的数据作为NumPy数组返回,形状为(n_epochs, n_channels, n_times):可选的picks参数根据索引、名称或类型选择频道的子集:
- 打印第一次的train处理部分可得epochs_train中包括65个片段
- event_train的[:, 2]为每个片段的event标识
- """
- events_data = events[:, 2]
- return epochs_data, events_data
-
-
- def draw_save(data, label_list, ch_names, save_path):
- # 这里对提取的数据进行绘制时频图并保存
- label_list = [str(i) for i in label_list]
- ch_len = len(ch_names)
- label_len = len(label_list)
- for count in range(label_len):
- # epochs标签(data.shape[0])
- for i in range(ch_len):
- # 通道数
- plt.rcParams['figure.figsize'] = (4.48, 4.48) # 2.24, 2.24 设置figure_size尺寸
- plt.rcParams['savefig.dpi'] = 50 # 图片像素 这样输出的就是4.48*50=224
- plt.specgram(data[count][i], NFFT=16, Fs=128, noverlap=10)
- k = str(i)
- q = str(count)
- path = save_path + label_list[count] + "/" + ch_names[i] + "_" + k + "_" + q + ".png"
- plt.savefig(path, bbox_inches='tight', pad_inches=0) # 后两项为去除白边
- # print("1")
-
- print(save_path + "has been finished\n")
-
-
- def main(file):
- raw = mne.io.read_raw_eeglab(file, preload=True, uint16_codec=None)
- print(raw.info)
-
- # 切出训练和测试集
- event_id = {'rt': 1, 'square': 2}
- raw_train = raw.copy()
- raw_train = raw_train.crop(0, 100)
- raw_test = raw.crop(100, 200)
-
- # 根据raw生成epochs,参考之前的transform()函数,具体在处理流程部分提供链接
- epochs_train, events_train = transform(raw_train, event_id)
- epochs_test, events_test = transform(raw_test, event_id)
- ch_names = raw.ch_names
- print(ch_names)
-
- train_save_path = os.path.join(os.getcwd(), "data/root/train/")
- draw_save(epochs_train, events_train, ch_names, train_save_path)
- test_save_path = os.path.join(os.getcwd(), "data/root/test/")
- draw_save(epochs_test, events_test, ch_names, test_save_path)
-
-
- if __name__ == "__main__" :
- # 获取父路径
- father_path = os.getcwd()
- # 设定子路径
- son_path = "eeglab-read-set/secdata.set"
- file = os.path.join(father_path, son_path)
- main(file)

四、结果展示
输出结果与之前的预期基本相同。
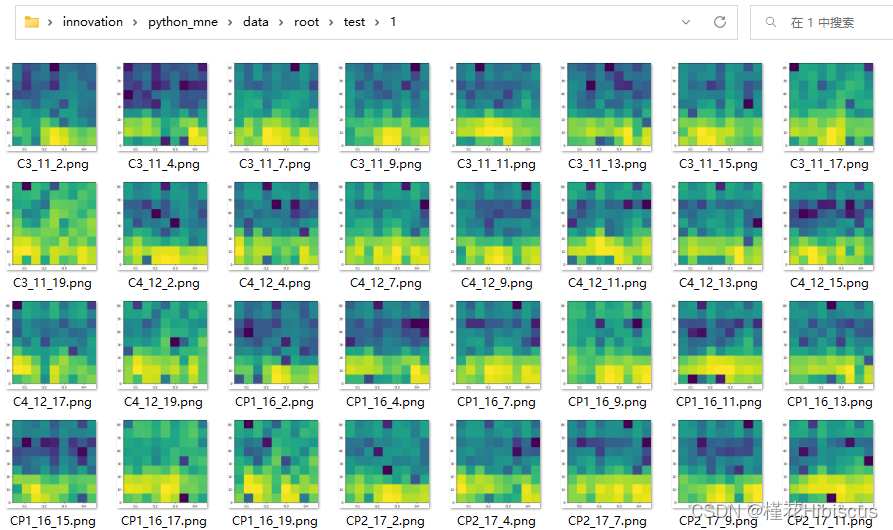
五、总结归纳
相比与之前,对于解决一个问题需要采用什么方法比较好有了较深的认识,对python的熟悉度也有了进一步的提高,但是解决问题的速度依然较慢。
六、参考文献:
【1】脑电EEG代码开源分享 【6. 分类模型-深度学习篇】
【3】python将批量音频信号(wav)转化为语谱图并保存(jpg/png)
【4】PyTorch深度学习快速入门教程(绝对通俗易懂!)【小土堆】
UPDATE
2023/3/13 增加后续cnn网络部分流程EEG深度学习实战:脑电信号通过CNN神经网络进行二分类


