- 1今年,中国AI大模型产业发展看这些_大模型数据
- 2Leetcode 73. 矩阵置零 C++_矩阵 所有值赋值为0 c++
- 3昇思MindSpore 25天学习打卡营|day1
- 4信号特征之希尔伯特变换(Python、C++、MATLAB实现)_python 希尔伯特变换
- 5idea用Git如何拉取、更新、上传提交项目(最新版idea2018.2.5版)超详细超简单版_idea git拖一个新增文件
- 6python查找图片_python 在屏幕中找 图片
- 7JAVAWEB项目页面设计之网上商城JSP页面设计一------JavaWeb_网站商城页面的web
- 8Oracle和My Sql返回结果集的存储过程
- 9超详细!一文搞定PID!嵌入式STM32-PID位置环和速度环_位置环pid分别代表什么
- 10【Android】如何使用ADB进行调试_adb调试
AlphaFold2: Highly accurate protein structure prediction with AlphaFold笔记
赞
踩
笔记:https://www.bilibili.com/video/BV1oR4y1K7Xr/?spm_id_from=333.788
DeepMind 2021年7月的发表在nature上的工作。同一天,University of Washington 发表了RoseTTAFold 并登在了8月的science的封面。
摘要
AlphaFold1不能在原子精度给出蛋白质结构。AlphaFold2在CASP14库结果较好。
整体架构
图1(1):CASP14各队的成绩比较,AlphFold2的误差为第一条竖线,为1埃原子精度
图1(2):绿色是实验室结果,蓝色为预测结果,黑色为碳原子
图2为PDB数据集结果
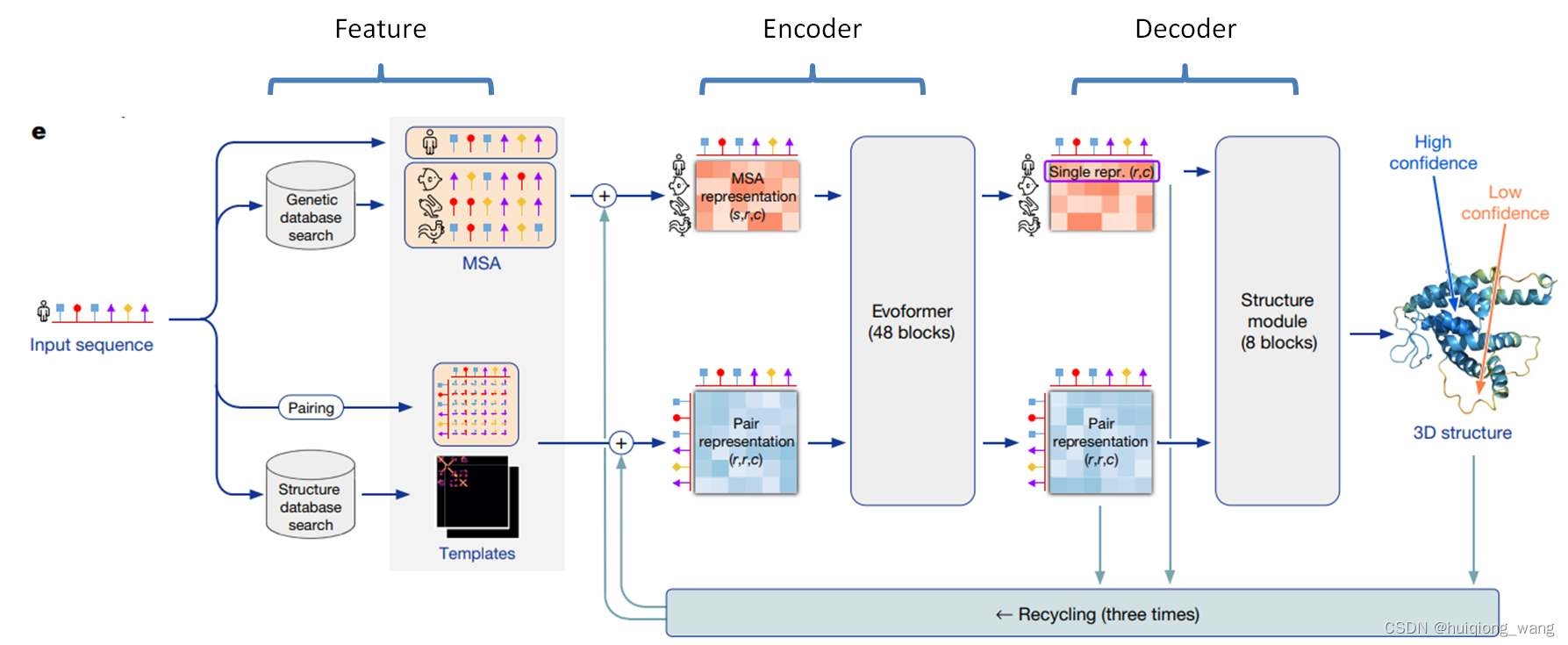
模型整体架构图
编码器
 编码器架构
编码器架构
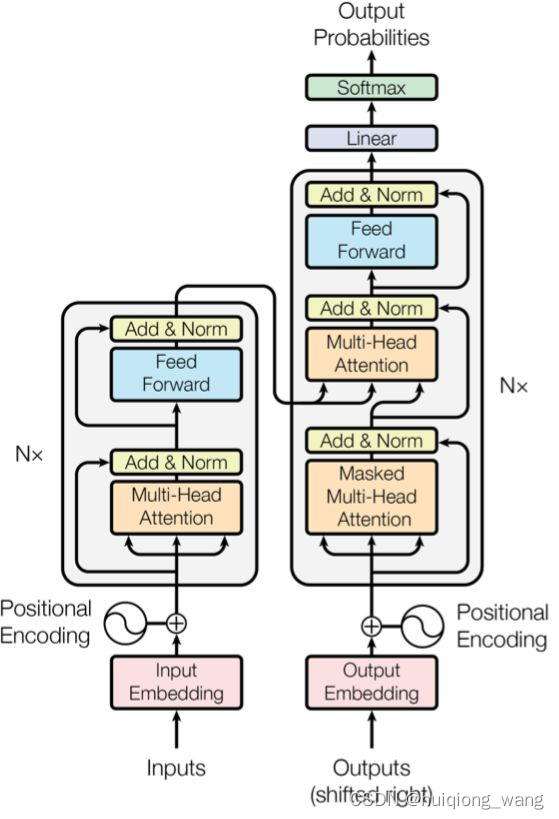
transoformer的编码器和解码器
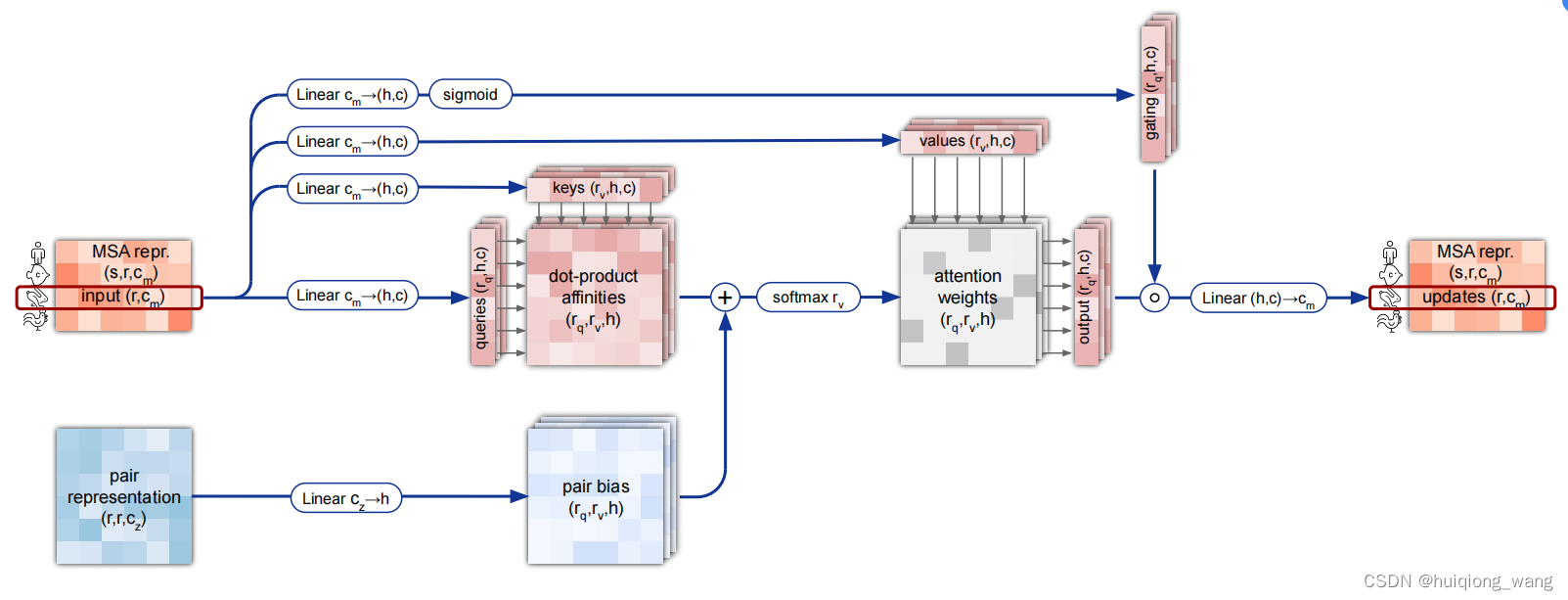
第一个自注意力实现细节:MSA row-wise gated self-attention with pair bias. Dimensions: s: sequences, r:
residues, c: channels, h: heads.

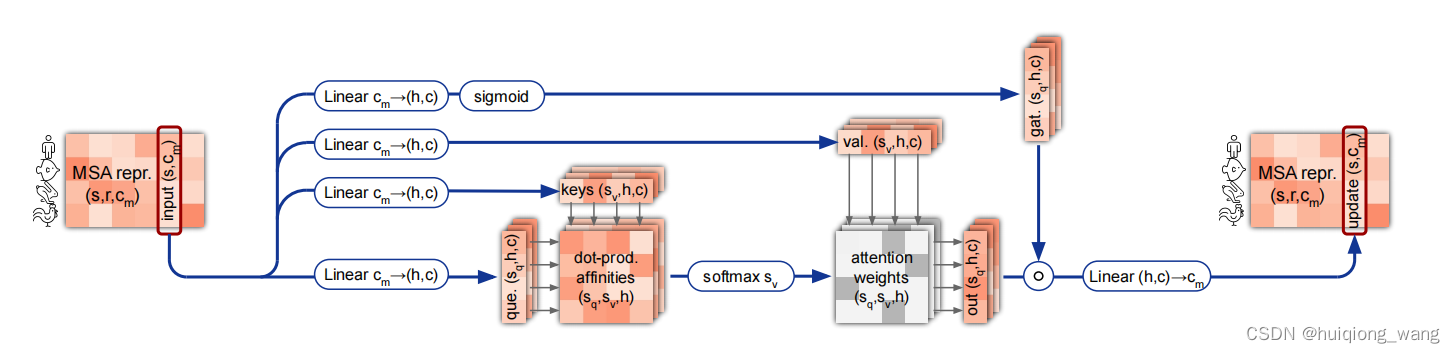
第二个自注意力实现细节,MSA column-wise gated self-attention. Dimensions: s: sequences, r: residues,c: channels, h: heads.
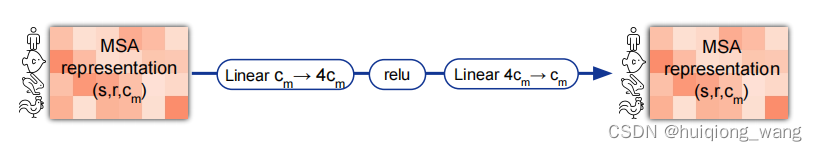
MLP层结构
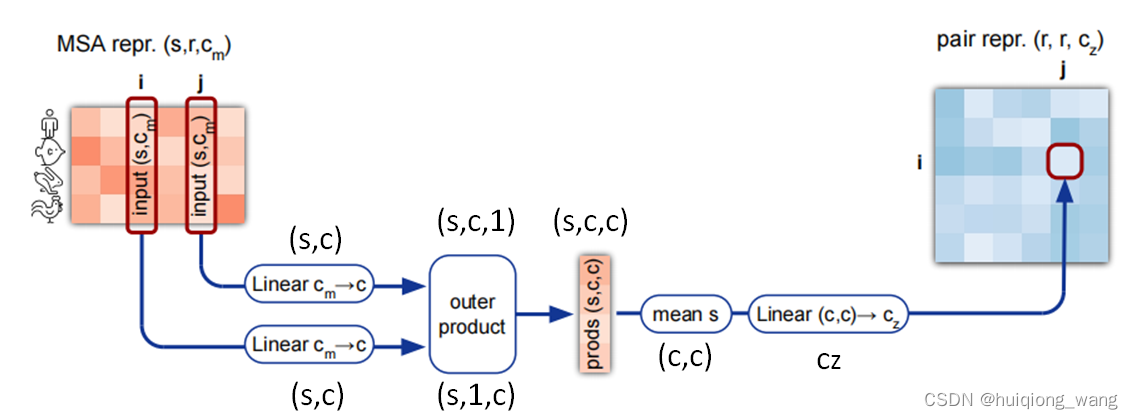
信息融合模块:Outer product mean. Dimensions: s: sequences, r: residues, c: channels.
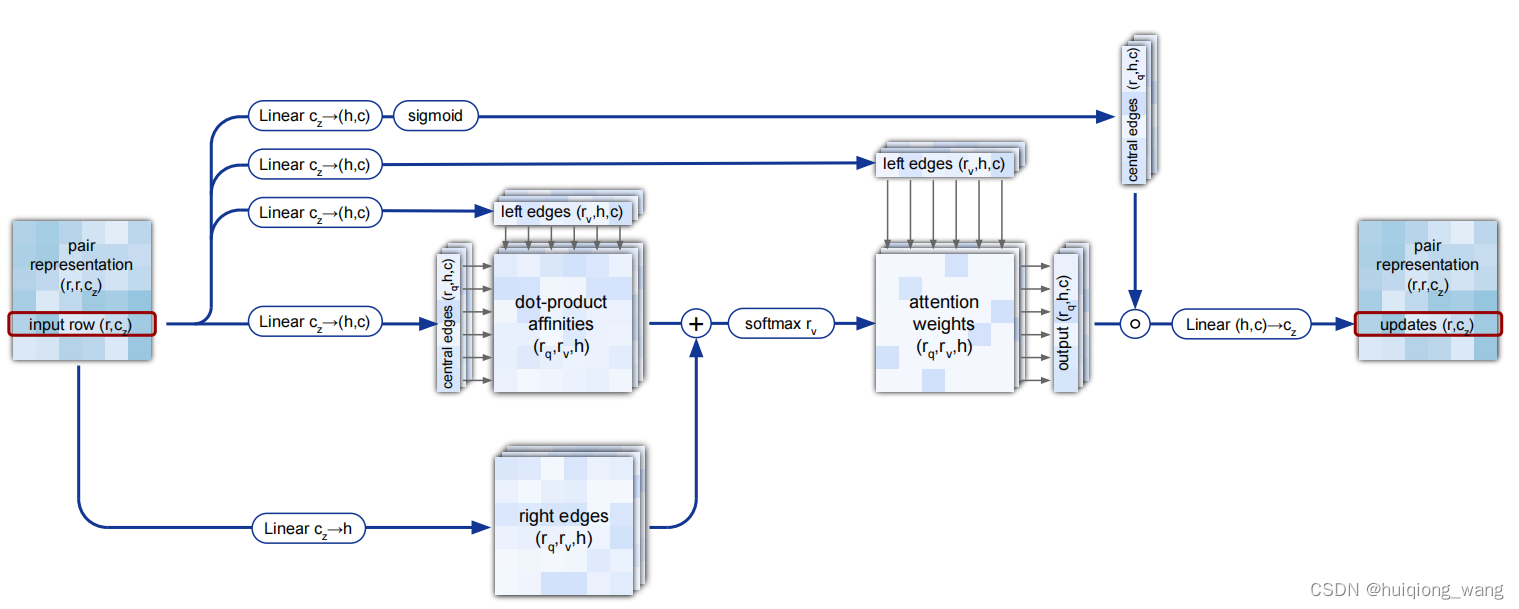
第三个自注意力机制与第一个注意力机制非常相似,Triangular self-attention around starting node. Dimensions: r: residues, c: channels, h: heads

第三个自注意力机制的伪代码

第四个自注意力机制的伪代码
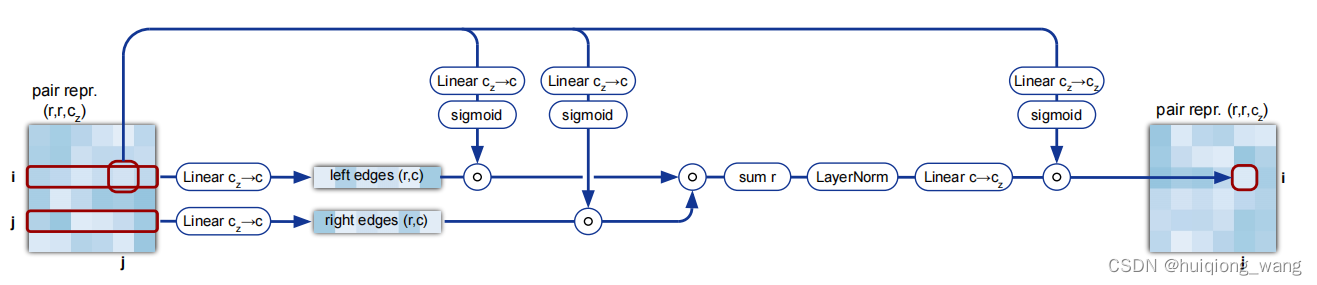
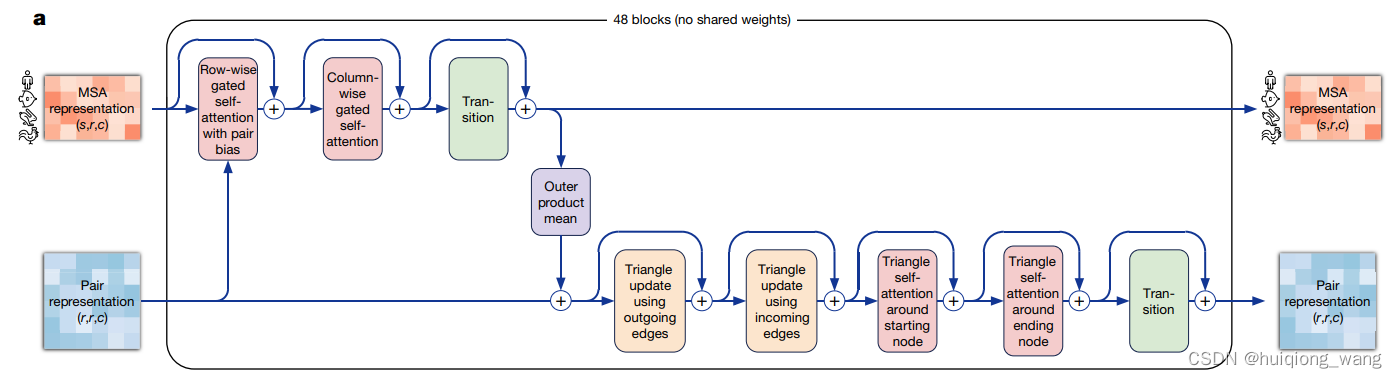
中间模块,用于减少计算开销和信息传递,Triangular multiplicative update using “outgoing” edges

信息传递
解码器
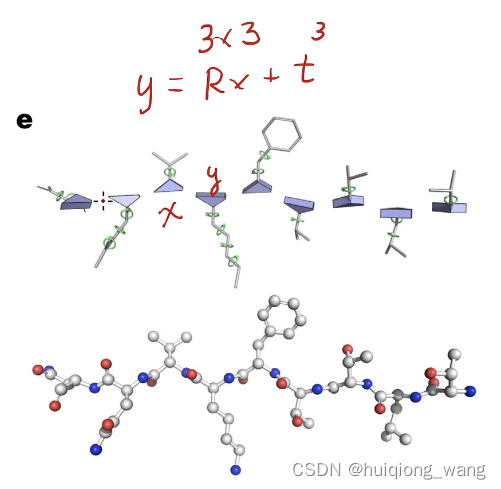
相对位子比绝对位子,能抵抗旋转和位移带来的表征不一致问题。
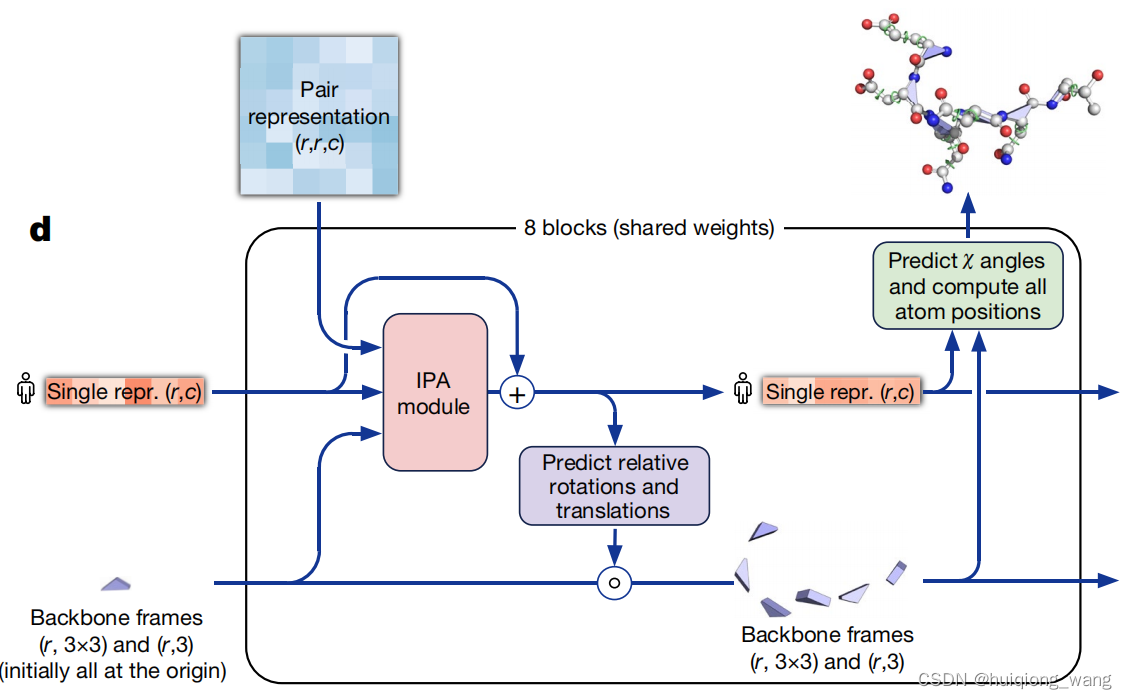
解码器整体结构
中间紫色模块是对主干的形态预测,右上角绿色模块是对分支结构的形态预测
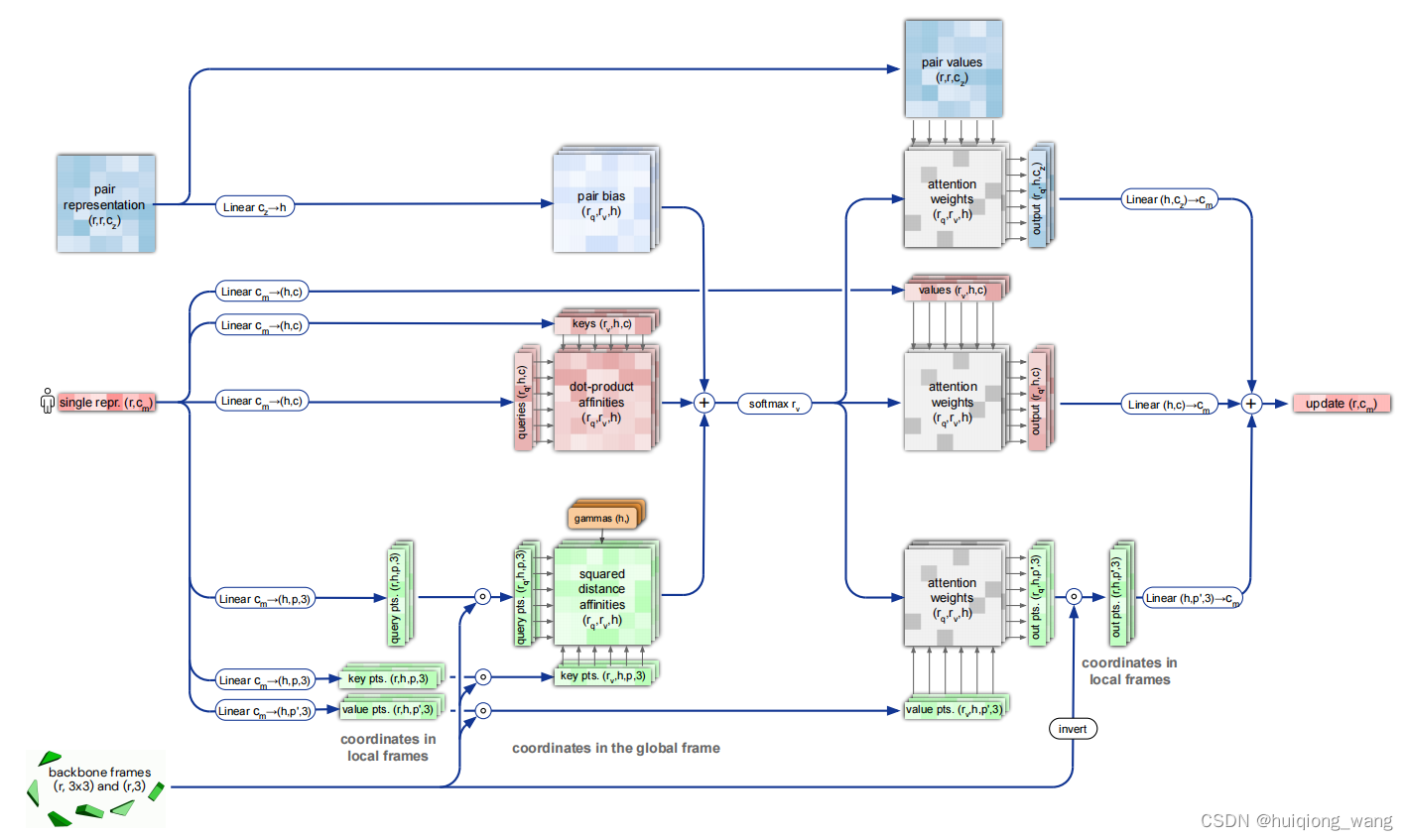
IPA模块结构,更加类似RNN和LSTM

IPA模块伪代码

角度偏移预测
训练
noisy student self-distillation:
pdb + 无标签的置信度高的数据组成新的训练集,循环往复
BERT:mask部分氨基酸或者对氨基酸做一些变换,通过预测被遮住的氨基酸,可以更好的建模
训练在128个TPU V3核上进行1周。


