- 1ConstraintLayout基本使用之toLeftOf 、toTopOf、toRightOf、toBottomOf_layout_constraintleft_toleftof
- 2小程序内嵌web-view实现路由切换到同级页面无效的解决方案
- 3一个人工智能的诞生_AI科学的诞生,世界人工智能之父发起的第一次研讨
- 4论题:基于深度学习的图像识别系统_基于深度学习的图像智能识别系统的研发立项目的
- 5【Java学习】java语言的执行模式--半编译和半解释型_java执行模式半解释型
- 6Java项目:42 ssm的高校专业信息管理系统设计与实现001
- 7基于VUE的ElementUi可视化表单设计器布局器_web表单设计器
- 8Xcode定义Preprocessor Macros,编译宏。
- 9pycharm ssh远程连接服务器 按这一步步走绝对连得上_pycharm ssh mapping
- 10安卓手机安卓Mysql,Termux安装MariaDB/MySQL数据库_最新termux安装 mariadb
【GEE】7、利用GEE进行遥感影像分类【随机森林分类】_gee分类交叉验证
赞
踩
1简介
在本模块中,我们将讨论以下概念:
- 监督和非监督图像分类之间的区别。
- Google Earth Engine 提供的各种分类算法的定义和应用。
- 如何使用 randomForest 设置和运行分类,以 aspen 存在和不存在作为示例数据集。
2背景
图像分类
人类自然倾向于将空间信息组织成组。从上面,我们识别出常见的地貌,如湖泊和河流、建筑物和道路、森林和沙漠。我们将这种具有相似特征的对象分组称为“图像分类”。但在全球范围内手动对对象进行分类和赋值将是一项无休止的任务。值得庆幸的是,使用遥感数据将不同的景观特征划分为分类类别已成为过去 40 年生态研究的主要内容。从农业发展和土地覆盖变化,到造林实践和污染监测,所有领域都进行了分类。
非监督与监督分类
的图像分类方法可以分为两类。首先,非监督分类涉及将潜在的预测变量应用于地理区域,并要求预测算法或先验回归系数来完成图像分类的工作。第二个,监督分类,需要创建独立的训练数据:概率模型可以用来发现观察条件和一组预测变量之间的关联的信息。
Google 地球引擎分类
器 在 Google 地球引擎ee.Classifier()功能的可用选项中,有几个属于“机器学习”的一般类别。算法函数从提供给它们的数据中“学习”,并根据学习到的信息进行预测。这些分类器特别擅长从大量遥感预测变量和(通常是高度非线性的)训练数据之间的关系构建统计模型。然后可以在大空间范围内应用这些模型,以生成地图输出形式的预测。近年来,分类和回归树 (CART)和 randomForest 等分类器已从计算机科学和统计学界引入生态研究。
randomForest
在 Google Earth Engine 中可用于监督分类的一种常用算法是 randomForest ( Breiman, 2001
// Import and filter Landsat 8 surface reflectance data. var LS8_SR1 = ee.ImageCollection('LANDSAT/LC08/C01/T1_SR') .filterDate('2015-08-01', '2015-11-01') //new date .filter(ee.Filter.eq('WRS_PATH', 35)) .filter(ee.Filter.eq('WRS_ROW', 33)) .filterMetadata('CLOUD_COVER', 'less_than', 20); // Create true color visualization parameters // to take an initial look at the study area. var visTrueColor = {bands: ["B4","B3","B2"], max:2742, min:0}; Map.addLayer(LS8_SR1, visTrueColor, 'LS8_SR1', false); Map.centerObject(ee.Geometry.Point(-107.8583, 38.8893), 8); // Define a cloud mask function specific to Landsat 8. var maskClouds = function(image){ var clear = image.select('pixel_qa').bitwiseAnd(2).neq(0); return image.updateMask(clear); }; // Apply the cloud mask function to the previously filtered image // collection and calculate the median. var LS8_SR2 = LS8_SR1 .map(maskClouds) .median(); Map.addLayer(LS8_SR2, visTrueColor, 'LS8_SR2 - masked')
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
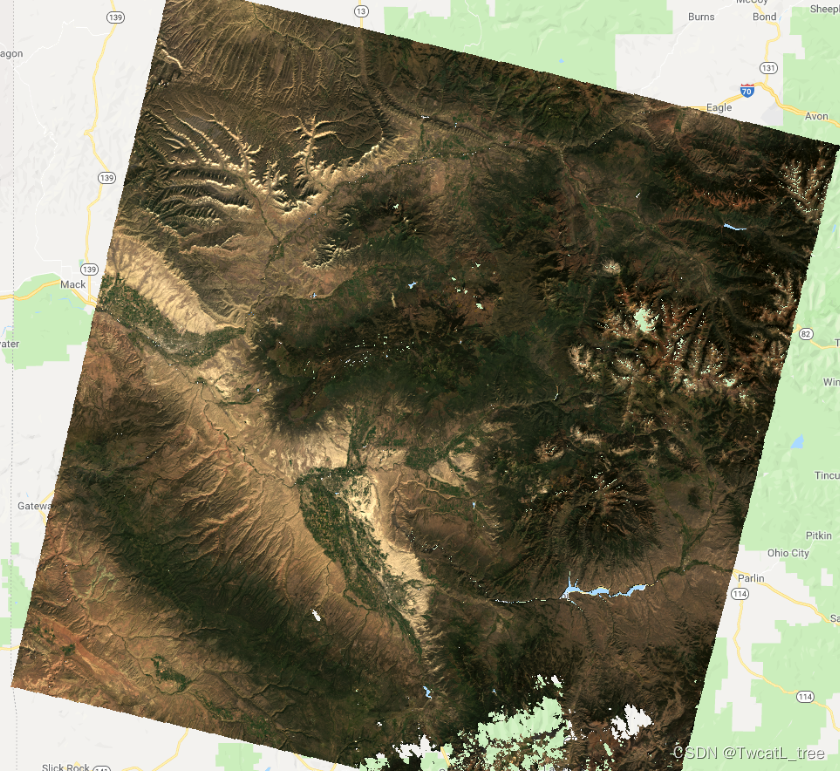
3.2制作预测列表
现在我们可以开始构建我们的预测器列表。您可能会阅读有关可以处理“高维”预测器列表的分类算法。这仅仅意味着可以包含大量潜在的解释变量。基于我们正在研究的生态系统的现有知识,我们可以选择一组初始变量,我们假设这些变量可以解释和预测景观中白杨的存在。将以下代码附加到您的脚本将构建一个多波段图像,其中包含我们所需的所有预测变量,包括一些与植被相关的光谱指数。打印predictors对象应该会在代码下方显示控制台输出。
// First define individual bands as variables. var red = LS8_SR2.select('B4').rename("red") var green= LS8_SR2.select('B3').rename("green") var blue = LS8_SR2.select('B2').rename("blue") var nir = LS8_SR2.select('B5').rename("nir") var swir1 = LS8_SR2.select('B6').rename("swir1") var swir2 = LS8_SR2.select('B7').rename("swir2") // Then, calculate three different vegetation indices: NDVI, NDWI, and TCB. var ndvi = nir.subtract(red).divide(nir.add(red)).rename('ndvi'); var ndwi = green.subtract(nir).divide(green.add(nir)).rename('ndwi'); var TCB = LS8_SR2.expression( "0.3029 * B2 + 0.2786 * B3 + 0.4733 * B4 + 0.5599 * B5 + 0.508 * B6 + 0.1872 * B7" , { 'B2': blue, 'B3': green, 'B4': red, 'B5': nir, 'B6': swir1, 'B7': swir1 }).rename("TCB"); // Combine the predictors into a single image. var predictors = nir .addBands(blue) .addBands(green) .addBands(red) .addBands(swir1) .addBands(swir2) .addBands(ndvi) .addBands(TCB) .addBands(ndwi) print('predictors: ', predictors);
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
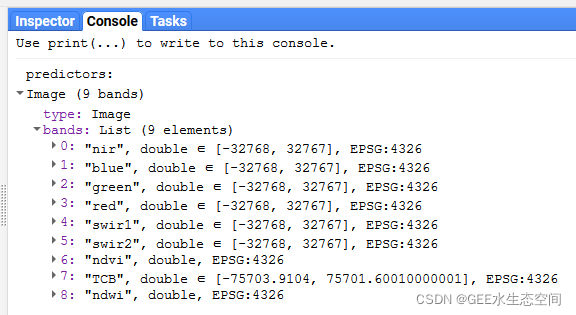
来自对象的打印语句的文本predictors,列出了每个波段以及相应的预测器名称。
3.3加载训练数据
要将值提取到点,我们必须首先导入我们的点数据集。幸运的是,我们已经准备好使用特定的 FeatureCollection ID 直接调用它。这些点表示白杨树存在和不存在的区域,因此我们将变量命名为PA。导入的训练数据可能要复杂得多,但为了我们的目的,一个简单的二元分类就可以解决问题。一旦我们加载我们的训练数据,我们将需要在每个点从我们的预测器中提取值。
将下面的代码添加到我们现有的脚本中,我们可以看到我们的训练数据已经加载完毕。随意根据您的个人喜好调整颜色,但结果应类似于下图所示。
var PA = ee.FeatureCollection('users/GDPE-GEE/Module7_PresAbs');
Map.addLayer(PA.style({color: 'red', pointSize: 3, width: 1, fillColor: 'white'}),{}, 'Merged_Presence_Absence');
var samples = predictors.sampleRegions({
collection: PA,
properties: ['presence'],
scale: 30 });
- 1
- 2
- 3
- 4
- 5
- 6
- 7
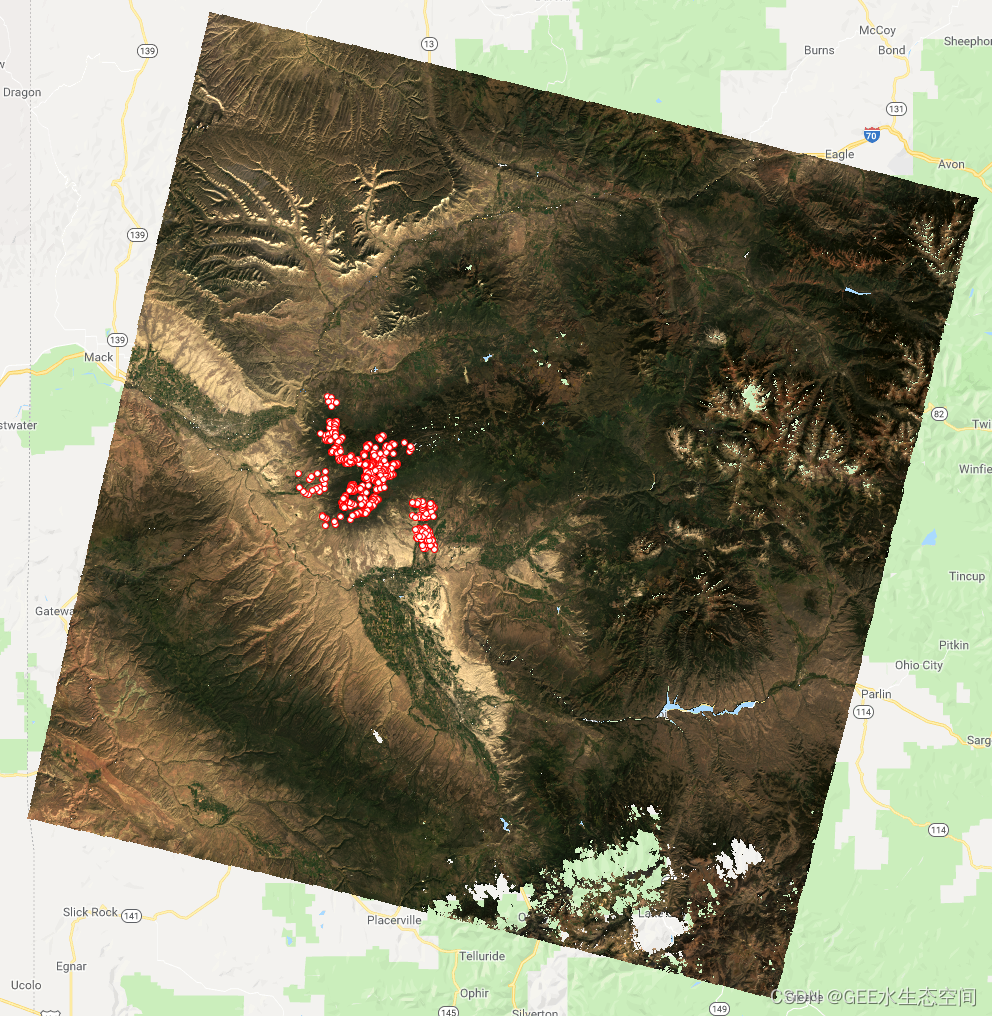
包含有关白杨林存在和不存在位置信息的点数据。
3.4构建模型
我们的训练数据现在包含为每个点位置记录的反射率值(来自我们的空间数据变量)。这就是我们的 RF 模型将用来了解 aspen 出现和不出现的地方。在我们这样做时,重要的是要了解 Google 地球引擎中的分类器算法应被视为对遥感潜力的初步探索,以增强您的工作。为什么是这样?让我们看一下您可以在 RF 分类器中调整的参数之一numberOfTrees:在这里,我们将这个数字保持在非常低的水平,以便相对快速地加载您的模型结果。将此数字从 10 增加到例如 1000,将导致 Google 地球引擎需要很长时间来处理。限制的问题numberOfTrees参数是研究表明,更多的树将生成统计上更稳健的 RF 模型(即 Evans 和 Cushman,2009 年)。正如 George Box 的名言所说,“所有模型都是错误的,但有些是有用的”,知道使用特定系统和算法的注意事项是很好的。
// Using the sampled data, build a randomForest model.
// Using a specific seed (random number) exactly replicates your model each time you run it.
var trainingclassifier = ee.Classifier.smileRandomForest({
numberOfTrees: 10,
seed: 7})
.train({
features: samples,
classProperty: 'presence'});
print(trainingclassifier);
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10

打印trainingclassifer对象后来自控制台选项卡的结果。请注意,我们可以验证我们选择了哪些模型选项,因为这些值是为numberOfTrees和打印的seed。
3.5精度评估
在承认 Google Earth Engine 中参数限制的警告之后,在我们使用模型的结果进行任何预测之前,了解我们对模型结果的信任程度仍然是一个好主意。评估分类器准确性的一种方法是查看混淆矩阵。请记住,这只是衡量我们训练数据的准确性!
将以下代码附加到您的脚本并重新运行以生成控制台输出,如下所示。这不是最漂亮的可视化,但它表明这似乎是一个高度准确的白杨存在和缺失模型。
// Print Confusion Matrix and Overall Accuracy.
var confusionMatrix = trainingclassifier.confusionMatrix();
print('Error matrix: ', confusionMatrix);
print('Training overall accuracy: ', confusionMatrix.accuracy());
- 1
- 2
- 3
- 4
3.6应用模型
对于可接受的模型精度,没有硬性规定。这将取决于您的数据集、研究领域和文献中设定的期望。话虽如此,我们的模型以非常高的精度生成,因此我们可以相当舒服地使用我们的模型对整个景观进行预测。不管模型的准确性如何,生态知识也可以帮助指导模型结果的解释。树木的预测不应出现在树线以上或峡谷底部的高山植被上。无论您是所在领域的专家还是仅使用常识,始终建议对模型输出进行目视检查。附加最后一段代码以完成建模演练并查看您的预测结果。
// Apply the model to the extent of the loaded predictor image.
var classified = predictors.classify(trainingclassifier);
Map.addLayer(classified, {min:0, max:1, palette:['white', 'blue']}, 'classified')
- 1
- 2
- 3

使用我们的 randomForest 模型对整个景观进行预测的结果。白杨存在以蓝色表示,不存在以白色表示。
4结论
在本单元中,我们介绍了 Google 地球引擎中的图像分类。我们讨论了分类方法的一些基本定义和一般特征,包括一种称为 randomForest 的机器学习算法。然后,我们使用 randomForest 来帮助我们通过结合来自遥感预测器和现场数据的信息来生成美国科罗拉多州西部白杨存在和不存在的景观尺度预测。
// Import and filter Landsat 8 surface reflectance data. var LS8_SR1 = ee.ImageCollection('LANDSAT/LC08/C01/T1_SR') .filterDate('2015-08-01', '2015-11-01') //new date .filter(ee.Filter.eq('WRS_PATH', 35)) .filter(ee.Filter.eq('WRS_ROW', 33)) .filterMetadata('CLOUD_COVER', 'less_than', 20); // Create true color visualization parameters // to take an initial look at the study area. var visTrueColor = {bands: ["B4","B3","B2"], max:2742, min:0}; Map.addLayer(LS8_SR1, visTrueColor, 'LS8_SR1', false); Map.centerObject(ee.Geometry.Point(-107.8583, 38.8893), 9); // Define a cloud mask function specific to Landsat 8. var maskClouds = function(image){ var clear = image.select('pixel_qa').bitwiseAnd(2).neq(0); return image.updateMask(clear); }; // Apply the cloud mask function to the previously filtered image // collection and calculate the median. var LS8_SR2 = LS8_SR1 .map(maskClouds) .median(); Map.addLayer(LS8_SR2, visTrueColor, 'LS8_SR2 - masked'); // First define individual bands as variables. var red = LS8_SR2.select('B4').rename("red") var green= LS8_SR2.select('B3').rename("green") var blue = LS8_SR2.select('B2').rename("blue") var nir = LS8_SR2.select('B5').rename("nir") var swir1 = LS8_SR2.select('B6').rename("swir1") var swir2 = LS8_SR2.select('B7').rename("swir2") // Then, calculate three different vegetation indices: NDVI, NDWI, and TCB. var ndvi = nir.subtract(red).divide(nir.add(red)).rename('ndvi'); var ndwi = green.subtract(nir).divide(green.add(nir)).rename('ndwi'); var TCB = LS8_SR2.expression( "0.3037 * B2 + 0.2793 * B3 + 0.4743 * B4 + 0.5585 * B5 + 0.5082 * B6 + 0.1863 * B7" , { 'B2': blue, 'B3': green, 'B4': red, 'B5': nir, 'B6': swir1, 'B7': swir1 }).rename("TCB"); // Combine the predictors into a single image. var predictors = nir .addBands(blue) .addBands(green) .addBands(red) .addBands(swir1) .addBands(swir2) .addBands(ndvi) .addBands(TCB) .addBands(ndwi) print('predictors: ', predictors); // Load the field sampling locations. var PA = ee.FeatureCollection('users/GDPE-GEE/Module7_PresAbs'); Map.addLayer(PA.style({color: 'red', pointSize: 3, width: 1, fillColor: 'white'}),{}, 'Merged_Presence_Absence'); // Determine the values of each predictor at each training data location. var samples = predictors.sampleRegions({ collection: PA, properties: ['presence'], scale: 30 }); // Using the sampled data, build a randomForest model. // Using a specific seed (random number) exactly replicates your model each time you run it. var trainingclassifier = ee.Classifier.smileRandomForest({ numberOfTrees: 10, seed: 7}) .train({ features: samples, classProperty: 'presence'}); print(trainingclassifier); // Print Confusion Matrix and Overall Accuracy. var confusionMatrix = trainingclassifier.confusionMatrix(); print('Error matrix: ', confusionMatrix); print('Training overall accuracy: ', confusionMatrix.accuracy()); // Apply the model to the extent of the loaded predictor image. var classified = predictors.classify(trainingclassifier); Map.addLayer(classified, {min:0, max:1, palette:['white', 'blue']}, 'classified')
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
- 54
- 55
- 56
- 57
- 58
- 59
- 60
- 61
- 62
- 63
- 64
- 65
- 66
- 67
- 68
- 69
- 70
- 71
- 72
- 73
- 74
- 75
- 76
- 77
- 78
- 79
- 80
- 81
- 82
- 83
- 84
- 85
- 86
- 87
- 88
- 89



