- 1小米员工爆料:公司为了规避年终奖,以低绩效为由提前裁员。
- 2MongoDB安全配置
- 3vscode常用插件
- 4信息量与熵_平均信息量与熵的关系
- 52022年全国职业院校技能大赛(中职组)网络安全赛项A模块解析_网络安全中职组技能大赛四个模块
- 6在Ruoyi中采用Ajax动态生成Echarts图表实践_若依框架图表
- 7docker-compose 命令不存在、未找到命令错误_docker-compose down 无效命令
- 8深入了解spark sql的join_spark sql full join
- 9搭建大型分布式服务(四十五)SpringBoot 单元测试Mock如何根据参数返回结果?_mock根据参数返回数据
- 10【渗透测试笔记】之【钓鱼姿势——Gophish平台钓鱼】_gopish
ATAC-seq学习记录_atac-seq优点
赞
踩
ATAC-seq意义
- 为何同样DNA序列的细胞的表型会不同,为何肝细胞是肝细胞,神经细胞是神经细胞?是什么造成了他们生产蛋白不同,决定蛋白生成的RNA不同呢?原因可以用
表观遗传来解释。

- DNA转录成RNA过程复杂,包括:
染色质可及性,DNA修饰,组蛋白修饰等等(选择性表达)。 染色质可及性即DNA开放区域,尤为重要。核小体由8个组蛋白组成复合物,每个核小体约147bpDNA。转录时DNA将从核小体复合物松开。许多因素,如染色质结构、核小体位置和组蛋白修饰,在染色质的组织和可及性起重要作用。致密核小体结构被破坏后,启动子、增强子、绝缘子、沉默子等顺式调控元件和反式作用因子可以接近的特性,叫染色质的可及性,也叫染色质开放性(chromatin accessibility ),这段区域叫开放染色质(open chromatin) 。- 什么是组蛋白修饰
- 定义:组蛋白包含5个部分,按分子量大小分别称为H1,H3,H2A,H2B和H4。组蛋白在相关酶作用下发生甲基化,乙酰化,磷酸化,腺苷酸化,泛素化,ADP核糖基化等修饰
- H3·H4乙酰化形成开放染色质结构,增加基因表达
- 组蛋白甲基化修饰多发生在H3H4,与基因抑制及激活相关,取决于被修饰的位置和程度
- 组蛋白磷酸化修饰一般与基因活化有关
- 组蛋白泛素化则是启动基因表达
- ATAC-seq
检测染色质可及性,确定基因表达调控机制。识别启动子区域、潜在的增强子或抑制子。启动子是靠近转录起始点(TSS)的DNA区域。包含转录因子的结合位点,转录因子招募RNA聚合酶。增强子是位于启动子下游或上游1Mb的DNA区域。当转录因子与增强子结合,并与启动子区域接触时,该基因的转录增加。相反抑制子会减少或抑制基因表达。 - ATAC-seq的峰往往是启动子,增强子序列以及一些反式调控因子结合位点。
- 2013年由斯坦福大学William J. Greenleaf和Howard Y. Chang实验室开发的ATAC-seq(
AssayforTransposase-AccessibleChromatinwith high throughput sequencing),一种捕获染色质可及性(染色质开放性)的测序方法。 - 为找到开放染色质区,基因组被
TN5转座酶处理。在ATAC-Seq中,修饰后的TN5将与NextEra接头相对应的DNA序列插入到基因组的开放区域,同时,DNA被转座酶活性剪切。 - 开放染色质的研究方法除了ATAC-seq,还有DNase-Seq,FAIRE-seq,MNase-seq 等。ATAC-Seq
所需样本少,建库快,重复性更高

- 故
ATAC-seq与Chip-seqcall出来的peak代表的意义不同。Chip-seqpeak是被目的蛋白结合拉下来的DNA,一般只有一个峰,而ATAC-seq是被Tn5转座酶切开、没有被组蛋白结合、染色质开放的DNA位点,如果是TF结合的区域,一般会有一个山谷般的存在。ChIP-seq和ATAC-seq在TF或者Tn5结合区域都会形成一个双峰的reads结合模式,但判断peak的时会有不同的标准。chip-seq是由于TF一起沉淀下来的DNA fragment一般会大于TF的结合区域,read的位置并不是真实TF结合位置,需要向内shift;而ATAC-seq一般是往两边shift。

应用上的区别
-
ATAC-Seq可
检测全基因组DNA结合蛋白,转录结合位点,一般用于不知道特定的转录因子,用此方法与其他方法结合筛查感兴趣的特定调控因子; -
ChIP-Seq是已知转录因子,根据
感兴趣的转录因子设计抗体去做ChIP实验富集结合的DNA片段。在测定转录因子的 ChIP-seq 中独有的峰可能是先驱转录因子,其先结合到封闭染色质,然后招募染色质重塑因子或其他转录因子起始转录。这些转录因子ATAC-seq检测不到。 -
得到DNA片段后,为测序准备建库,包括
用完整的NextEra接头和纯化、PCR扩增等。基于上述原因,ATAC-Seq推荐使用双端配对的方法。
应用
- 染色质
开放性图谱绘制,表观基因组图谱 - 找
调控生物学过程的关键转录因子 - 找
哪个转录因子调控了研究的基因 - 找转录因子调控的
靶基因 - 得到
不同组织或不同条件下对应可及性区域。 - 得到核小体位置
- 生成转录因子结合区域的特征(footprinting)
技术限制
- Tn5通过
插入剪断DNA 并将测序接头连接到剪断的两个DNA 片段的末端,因此对于一个DNA 片段而言,其两端的接头连接是随机的,导致同一片段两端的接头有50%的概率是同一接头。而只有连接不同接头的片段才可用于富集扩增及测序,因此一半的片段无法利用; 大量剪断的DNA 由于片段过大,无法进行PCR富集;- Tn5 的
活性受反应溶液的组成及反应条件影响,仍然需要优化以便提高剪切效果; - ATAC-seq在
植物细胞存在以下难点:细胞壁,叶绿体线粒体等细胞器污染,缺少稳定遗传的细胞系;
ATAC-Seq、Dnase-Seq、MNase-Seq、FAIRE-Seq
- 整体的分析思路一致,
找富集区域,对富集区域进行功能分析。 - ChIP-Seq是
揭示特定转录因子或蛋白复合物的结合区域,实际是研究DNA和蛋白质的相互作用,利用抗体将蛋白质和DNA一起富集,并对富集的DNA测序。 - DNase-Seq、ATAC-Seq、FAIRE-Seq都
研究开放染色质区域: - DNase-Seq用
DNase I内切酶识别开放染色质区域, - ATAC-seq用
Tn5转座酶,随后进行富集扩增; - FAIRE-Seq先超声裂解,后用酚-氯仿富集;
- MNase-Seq鉴定核小体区域。
下图是不同测序方法获取的峰形:

检测染色质可及性的方法中,ATAC-seq尤其受欢迎。

- ATAC-seq的优点:
Tn5转座酶的高活性使ATAC-seq简单,省时,而且只需500-50,000个细胞。灵敏度特异性与DNase-seq相当,优于FAIRE-seq。
整合分析
-
由于开放染色质是大多数TF结合的先决条件,因此
ATAC-seq峰通常与TF ChIP-seq峰重叠,但通常更宽。因此,TF ChIP-seq和ATAC-seq可以在同一实验系统中相互验证彼此的质量和可靠性。 -
ATAC-seq与 histone marker ChIP-seq集成,发现与活跃染色质标 H3K4me3,H3K4me1,H3K27ac等正相关,与不活跃的染色质标记 H3K27me3 负相关。
? -
ATAC-seq+RNA-seq: 一般RNA-seq会优先于ATAC-seq先测,但差异基因富集的基因通路只是一种相关性。要分析出其中谁调控目的基因,可通过ATAC-seq做motif分析,寻找潜在的调控因子,然后再后续的实验验证或者chip-seq验证。/ 看ATAC上丰度高的DNA序列区域是否对应转录本表达量增加,找到对应转录本相关基因的上游调控序列,整体分析转录。对基因功能分析,结合实验表型,推测表达调控-表达-功能-表型。

-
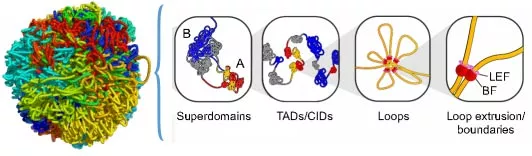
ATAC-seq+HiC: 对于一些想了解染色质高级结构对生命行为的作用的时候,通常会需要用到ATAC-seq等技术,因为Hi-C分析得到高级结构compartmentA/B、TADs、Loops等信息,通常只是相关性,但通过ATAC-seq,可以获得promoter、enhancer等信息,更能知道高级结构是如何影响启动子、增强子从而影响基因表达的。
-
ATAC-seq+组蛋白修饰: ATAC-seq预测一个位点的开放程度以及可能有某种转录因子的结合,但不知道该因子是促进基因表达,还是抑制,只通过基因层面鉴定来判断转录因子对基因的促进或者是不够的,它只是一种相关性。而这时候如果能提供像H3K27ac这类激活型组蛋白、H3K27me3这类抑制型组蛋白将能使数据结果可信。国内较早研究iPSCs的学者如裴端卿的工作可以看到,在解析iPSCs重编程中的染色质可及性的时候,不仅用到ATAC-seq来描述细胞的身份转变,还通过H3K27ac指征该区域的激活。其中一篇还通过调控成纤维细胞关键基因启动子区去乙酰化修饰,达到了促进重编程的进程。

-
scATAC-seq+scRNA-seq: 更前沿的技术一个细胞里同时进行RNA-seq和ATAC-seq,并且是单细胞水平的检测。SHARE-seq,能够实现在单细胞中同时高质量,高通量的检测基因表达和染色质可及性。该技术可以使用染色质潜力算法(chromatin potential),用ATAC和RNA的差异来预测细胞的变化方向。相对于以往仅依赖于RNA的预测手段,染色质潜力能够大大提前预测的时间。

- 思考:
- ATAC-Seq与ChIP-Seq的异同在哪里?
- 用和ChIP-Seq一样的参数Call peaks正确吗?
- 得到peaks后怎么进行质量评估?
- 样本内的重复怎么处理?
- 样本间的差异怎么分析?
- 怎么对peaks进行功能注释分析?
- 如何找motif?
- ATAC-Seq和ChIP-Seq和RNA-Seq的整合分析怎么做?
- 待学习:Harvard Chan Bioinformatics Core (HBC)深度NGS数据分析课程,第5部分关于ChIP-Seq,整体思路和绝大部分分析方法适合ATAC-seq。

待学习内容:
- ATAC-seq data analysis: from FASTQ to peaks
- ATAC-seq Data Standards and Processing Pipeline in ENCODE
- ATAC-seq数据分析实战
- Harvard FAS Informatics - ATAC-seq Guidelines
HBC深度NGS数据分析课程:
https://github.com/hbctraining/In-depth-NGS-Data-Analysis-Course
第五部分ChIP-Seq课程:
5. https://github.com/hbctraining/In-depth-NGS-Data-Analysis-Course/tree/master/sessionV/lessons
1:ATAC-seq的背景介绍以及与ChIP-Seq的异同
2:原始数据的质控、比对和过滤
3:用MACS2软件call peaks
4:对ATAC-Seq/ChIP-seq的质量评估(一)——phantompeakqualtools
5:对ATAC-Seq/ChIP-seq的质量评估(二)——ChIPQC
6:重复样本的处理——IDR
7:用Y叔的ChIPseeker做功能注释
8:用网页版工具进行motif分析
9:差异peaks分析——DiffBind
10:ATAC-Seq、ChIP-Seq、RNA-Seq整合分析
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
参考文献:
https://mp.weixin.qq.com/s?src=11×tamp=1633159169&ver=3349&signature=*MwqLr1J-qdZoNiKVxF32vEKh5-6TRystOXAJ3UOZ3Pl8XTBIB8Ly95IJM0L2EzGFVWOM-TdKnuhnb0gfMfsUTfahWJ5i3hcM2TcR9UDFSVWuyYw7CONzMjsMaYQG2Ca&new=1
https://mp.weixin.qq.com/s?src=11×tamp=1633159169&ver=3349&signature=rtYw5NsC62rUZvctQsUg3*w*NFFDdOHgSMu0pcp0HTQdCyqxpgril8yx7GWlJaID*lfd2HRLUWs59zuszSEFeean0jEwdRs4PzYy*T5b7nSpZRWqCs4SHcEQ2jyjDtwQ&new=1
- 1
- 2
- 3
简洁版ATACseq分析流程
- 数据预处理
- (1)比对前质量控制FastQC
- (2)原始序列比对
- (3)比对后处理和质量控制:去除重复序列,细胞器序列
- 序列比对后,Picard/SAMtools收集unique mapping reads/rate,duplicated rate百分比和片段大小分布
- 成功的ATACseq实验应生成
片段大小分布图(从bam文件得到),具有递减性和周期性的峰,对应于无核小体区域(NFR)(<100bp)和单核双核和三核小体(200,400,600bp)。大多数Linker DNA大小介于10-80bp间,故大多数片段都会是小于100bp。每个Nucleosome的DNA大小为180bp,加上两边插入的冗余,会得到大约200bp长度的mono-nucleosome的DNA。 无核小体区域的片段应该在基因的转录起始位点(TSS)周围富集,而核小体结合区域片段TSS处形成低谷,TSS周围侧翼区域稍微富集。ATACseqQC评估。
- Peak-calling:从比对得到的bam文件找出reads覆盖区,就是峰出现的位置。
- 高级分析
- (1)peak 差异分析:寻找不同分组差异peaks
- (2)peak注释:峰的注释可将染色质的可及性与基因调控联系。通常峰会被注释到最接近的基因或调控原件。获得最接近的基因列表后,使用GOKEGGReactome等数据库功能富集分析
- (3)motif富集分析:得到每个peak region里motif的位置和频率,再和随机背景或其他条件比较,可做motif富集分析
- (4)footprint分析 :ATACseq中footprint指一个TF结合在DNA上,组织Tn5切割,在染色质开放区域留下一个相对缺失的位置。而TF周围的组蛋白因为TF造成空间的推挤反而形成开放度较高区域。


