热门标签
热门文章
- 1C# Winform实现炫酷的透明动画界面(转载)
- 2ktt算法 约化_svm算法 最通俗易懂讲解
- 3职业规划(程序员)中 业务型 和 技术型 那个更重要?_程序员业务和技术哪个重要
- 4ajax的get,post请求和解决回调地狱的方法封装_ajax post 回调
- 5learn python是什么软件-Python中常用包——sklearn主要模块和基本使用方法
- 6Linux使用YUM安装程序_linux 安装yum
- 7js使用formData上传多个文件时,获取文件对象_formdata转file
- 8《Flutter开发从入门到实战》:为什么要学Flutter?_为什么 学习flutter
- 9Django 分页 & Vue父子组件 实现_子组件写个分页 ,请求接口在父组件 怎么写
- 10MySQL分页坑(limit+order by数据重复)_分页查询第0页是第一页的数据
当前位置: article > 正文
生信分析R语言助力作图----单基因批量相关性分析_单基因相关性分析
作者:很楠不爱3 | 2024-02-23 00:10:29
赞
踩
单基因相关性分析
单基因批量相关性分析
嘻嘻嘻~~~,晚上秒变生信分析小白,一个游走在生物学和计算机变成之间的小白,享受着里面的快乐和痛苦。不停的挣扎,不停的成长,多学习,多尝试,一定会有意想不到的收获。加油!!!
首先,需要先说明的是本作图经验,完全模仿于果子学生信的一篇博文,所以先把链接丢在这里啦,自己写这篇博文,也完全是为了记录这个操作过程,好在下次需要的时候,更好的去回忆。话不多说,开始表演。
https://mp.weixin.qq.com/s?__biz=MzIyMzA2MTcwMg==&mid=2650733008&idx=1&sn=b66e3fd527f99ddf19dcf6c2501e5be3&chksm=f029aa79c75e236f8951b87e17a51dc6a7dfeb555b983d8dc6d5a8c756b3eacc21829f19dc86&token=1035043907&lang=zh_CN&scene=21#wechat_redirect
首先,做这个相关性分析,在这里需要安装几个R包。
BiocManager::install(c("tidyr","dplyr","ggstatsplot"))
##加载的数据,就是我们常见的TCGA的表达矩阵,只是需要先将其进行转置。
##我们在此用得是自己的一个Test的数据。其格式如下:
- 1
- 2
- 3
注意,在此处,基因名一定要作为列名,因为后面需用ggstatsplot进行散点图绘制的时候,其需要用到列名中的基因名字。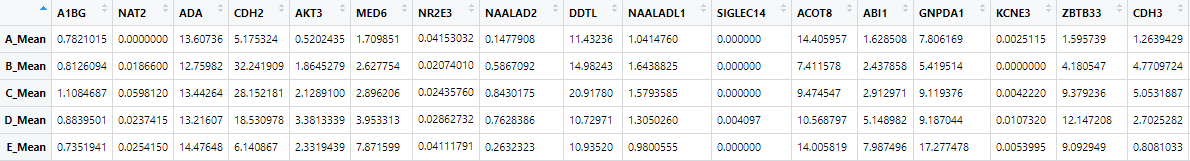
该数据,列名就是基因名,行名就是样本名。为了缩短数据处理时间,我们在此只取前500列作为后续的实验分析。
test3 <- test2[,1:500]
head(test3[1:3,1:3])
A1BG NAT2 ADA
A_Mean 0.7821015 0.000000 13.60736
B_Mean 0.8126094 0.018660 12.75982
C_Mean 1.1084687 0.059812 13.44264
y <- as.numeric(test3[,"A1BG"]) ## 本次操作中,选取A1BG基- 1
- 2
- 3
- 4
- 5
- 6
- 7
推荐阅读
相关标签


