- 1Metaio in Unity3d 教程--- 二. 创建自己的Application_unity 安卓自定义application
- 2数据中台驱动:高效交付之道
- 3编程语言字典(自用)_可供编程的词典机制是什么
- 4MySQL-窗口函数-排序_mysql窗口函数排序
- 5Android—内容提供者_android什么是内容提供者
- 6实现浏览器兼容版的window.getComputedStyle_getcomputedstyle的兼容写法
- 7【怎么注册小程序】小程序怎么注册?
- 8C++——一种特殊的二叉搜索树之红黑树_c++红黑二叉树
- 9html如何保存在文档桌面,如何把word文档保存到桌面
- 10逻辑思维训练——假设法
深度学习零基础学习之路——第四章 UNet-Family中Unet、Unet++和Unet3+的简介
赞
踩
Python深度学习入门
第一章 Python深度学习入门之环境软件配置
第二章 Python深度学习入门之数据处理Dataset的使用
第三章 数据可视化TensorBoard和TochVision的使用
第四章 UNet-Family中Unet、Unet++和Unet3+的简介
第五章 个人数据集的制作
前言
最近学习了Unet、Unet++和UNet3+模型,并且对这三者进行了一些研究,并将其作为组会上报告的内容,效果还是不错,因此趁自己还记得一些,写一个博客记录一下,方便后续复习,不得不说Unet模型还是很强大的,也难怪Unet模型现在很火,值得一学。
一、FCN全卷积网络模型
FCN网络模型全称为全卷积神经网络模型(Fully Convolution Network),该模型是2015年由Jonathan Long等人在一篇论文《Fully Convolutional Networks for Semantic Segmentation》中提出的语义分割模型。该模型算得上是深度学习用于语义分割领域的开山之作,在后续的语义分割模型中都可以看到FCN模型的影子。其模型结构和CNN非常相像,因此该模型也算是卷积神经网络CNN的升级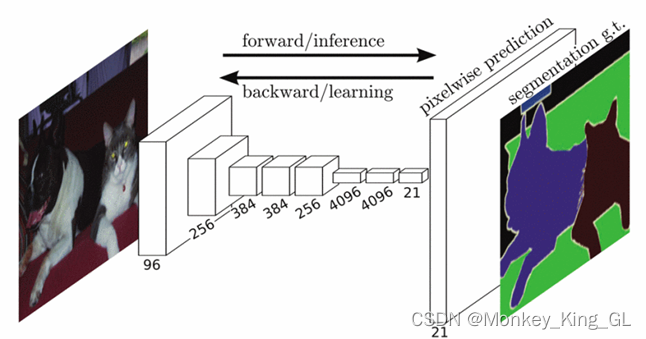
FCN核心思想:
1、 将CNN的全连接层换成了卷积层,这样FCN就适应任何尺寸的图片输入,也可以使得网络输出是一个热度图(heatmap),而非单个类别标签。
2、 加入上采样操作(反卷积) ,将卷积得到的feature map上采样到原图大小,然后这样就可以做像素级别的分类,这样就可以将分割任务变成了分类问题。
3、 采用跳跃连接,即在上采样过程中加入不同深度的feature map。这样既可以将下采样过程中丢失的细节数据补全,又保留了原始图像的空间信息,使得模型具备更高的精度与鲁棒性。这样就是为什么FCN网络模型有FCN32、FCN16、FCN8,如下图.

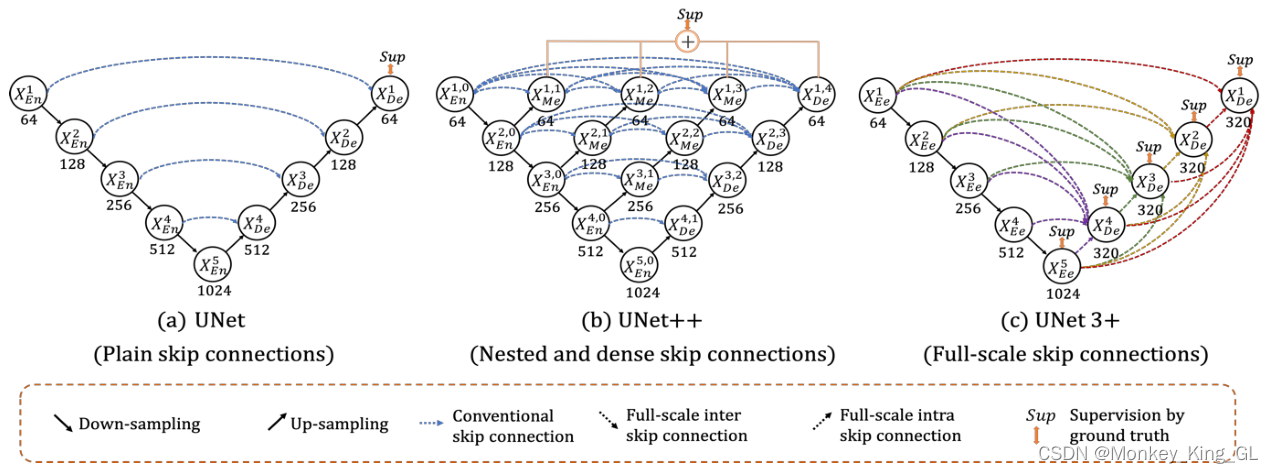
二、Unet编码模型
FCN模型的提出成为了深度学习解决分割问题的奠基石。但Unet模型的提出可以说是将深度学习解决分割问题推上了一个新的高度。论文《U-Net: Convolutional Networks for Biomedical Image Segmentation》中的Unet模型是与FCN同年2015年提出来的,但其还是晚于FCN。Unet模型可以算是医学图像分割领域的领头者,其也是通过下采样获取特征图,然后再上采样还原到原图,但Unet模型有很多独特的地方,正因这些特点才使得U-Net网络模型到现在还如此火热。
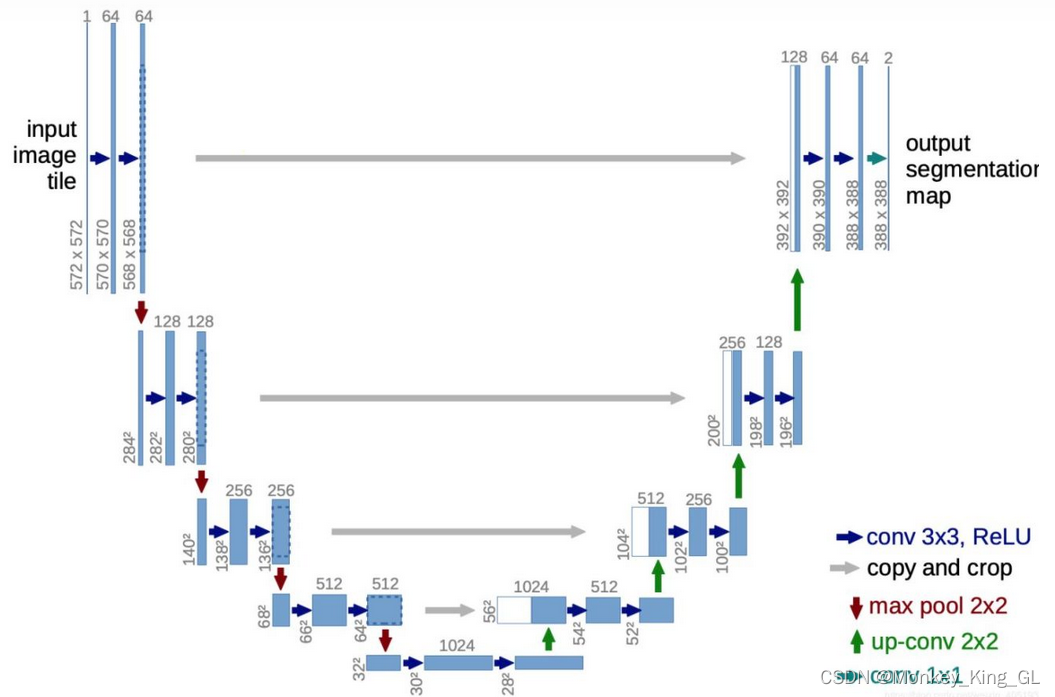
U-Net模型独特之处:
1、模型结构完全对称:
U-Net模型的结构完全异于CNN和FCN,左半边下采样,右半边进行对等的上采样。
2、采用编码和解码结构(Encoder-Decoder):
1)Encoder:编码器整体呈现逐渐缩小的结构,不断缩小特征图的分辨率,以捕获上下文信息。编码器共分为4个阶段,在每个阶段中,使用最大池化层进行下采样,然后使用两个卷积层提取特征,最终的特征图缩小了16倍;
2)Decoder:解码器呈现与编码器对称的扩张结构,逐步修复分割对象的细节和空间维度,实现精准的定位。解码器共分为4个阶段,在每个阶段中,将输入的特征图进行上采样后,与编码器中对应尺度的特征图进行拼接运算,然后使用两个卷积层提取特征,最终的特征图放大了16倍;
3、U-Net式的跳跃连接(skip connection):
该处的跳跃链接的作用和FCN处的跳跃连接作用是一样的,都是为了使得上采样恢复的特征图中包含更多low-level的语义信息,使得结果的精细程度更好。但它叫U-net式的跳跃连接是为了区分FCN式的跳跃连接,其区别在于Unet式的跳跃连接是channel维度的拼接融合,而FCN式的跳跃连接只是对应像素点的简单加和。
U-Net模型各方面看起来是很好,但是我学到这里的时候就心中有很多疑问疑问了:
- U-Net模型一定要按论文中的模型一样下采样四次才叫U-Net模型吗?
- 下采样对于分割网络是不是必须执行的呢?
- 上采样必须要等到下采样结束才可以开始上采样吗?
这些疑惑终于在UNet++网络模型的提出者写的一篇博客中解开了。链接:https://zhuanlan.zhihu.com/p/44958351。
1、U-Net模型一定要按论文中的模型一样下采样四次才叫U-Net模型吗?
答:我个人觉得这个说法是不正确的,U-Net模型应该指的是一种思想,一种架构,如将Encoder-Decoder结构运用到模型中、模型结构完全对称、采用U-Net式的跳跃连接等等,我们不应该拘束于用什么卷积,用几层,怎么降采样,学习率多少,优化器用什么,这些都是比较直观的参数,其实这些在论文中给出参数并不见得是最好的,所以关注这些的意义不大。
2、下采样对于分割网络是不是必须执行的呢?
答:我们先要了解下采样的作用是可以增加对输入图像的一些小扰动的鲁棒性,比如图像平移,旋转等,减少过拟合的风险,降低运算量,和增加感受野的大小。如果图片比较小、颜色、对象比较单一、很容易提取特征,下不下采样对于模型的预测影响不大的话,何必折腾下采样再上采样呢?
3、上采样必须要等到下采样结束才可以开始上采样吗?
答:这个问题我们可以这样理解,分别将U-Net模型下采样1次、2次、3次开始上采样会得到什么效果,一层、两层、三层、四层的Unet模型,这样我们就可以知道这个问题的答案了吧。
Unet的不足之处就在于它仅有同层之间的连接,上下层存在信息代沟现象。
三、Unet++模型
U-net++模型顾名思义就是U-Net模型的升级版,它出自论文《UNet++: A Nested U-Net Architecture for Medical Image Segmentation》,它既融合了Unet模型的结构思想,也解决了Unet模型存在的不足。作者当时就在想,既然Unet模型不一定要下采样四次才是最佳的,那下采样多少次才是做好呢?作者就进行了不同层模型对比实验(如下图),实验表明最佳的模型结构因数据集的不同而不同。
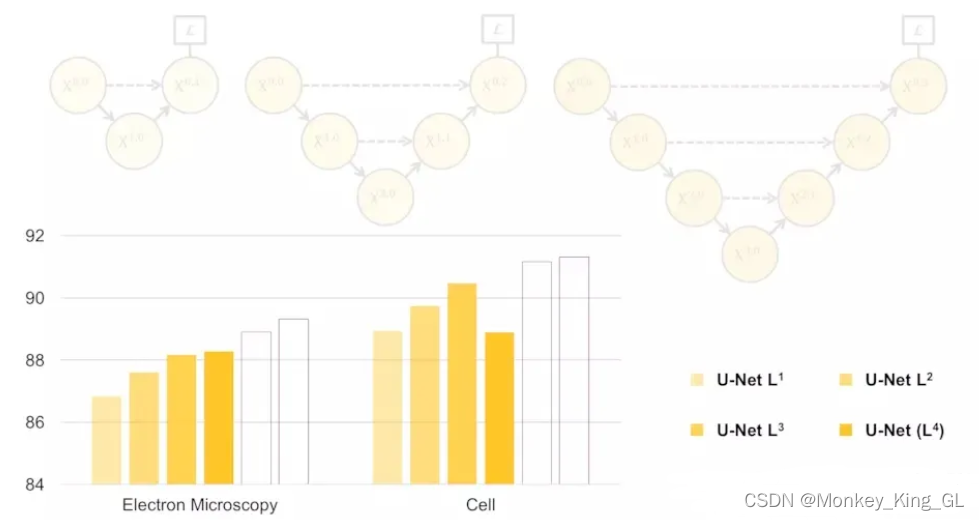 既然模型的下采样次数不是一个确定的值,那我们在训练模型前都需要把这些不同层的模型训练测试对比一下吗?我想应该是不需要的,不然这样太麻烦了,我们可以把这些模型融合到一个模型中去,让网络自己去学习不同深度的模型,这样就得到Unet++模型的基本结构了。
既然模型的下采样次数不是一个确定的值,那我们在训练模型前都需要把这些不同层的模型训练测试对比一下吗?我想应该是不需要的,不然这样太麻烦了,我们可以把这些模型融合到一个模型中去,让网络自己去学习不同深度的模型,这样就得到Unet++模型的基本结构了。
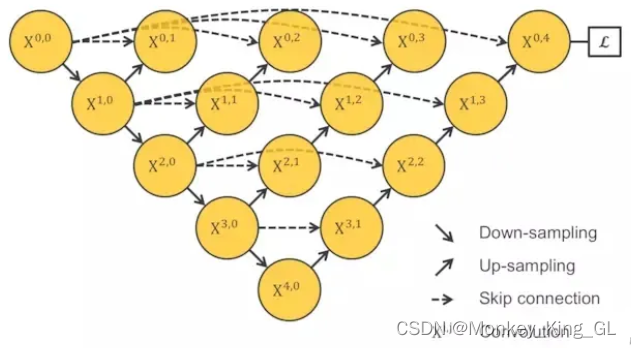
但是模型中间的X0,1,X0,2,X0,3,X1,1,X1,2,X2,1节点 因与最后计算损失值的函数LossFunction是断开的,导致模型在训练过程中的反向传播无法到达,进而导致模型无法训练。针对这个问题,有两种解决方式:
- 在模型的中间节点之间增加短连接,让模型训练过程中的反向传播可以到达每个节点。
- 将节点==X0,1X0,2X0,3,X0,4==后面加个1x1的卷积核,然后再与计算损失值的函数LossFunction相连去监督每个level的输出 (这也是Unet++的一个重要特点,深度监督Deep Supervision),这样整个模型就类似于是1、2、3、4层U-Net模型的叠加了。

U-Net++的创新点:
1、加入深度监督(Deep Supervision):
深度监督就是在每一层的末尾添加一个1x1的卷积核,然后再与计算损失值的损失函数LossFunction相连,这样有什么好处呢?
1)使模型更加完整,解决了模型反向传播时无法到达的情况。
2)使模型可以进行剪枝操作,因为我们Unet++模型融合了不同层的Unet模型,在测试的过程中模型太过大会影响测试效率。但是在测试过程中输入的图像只会前向传播,因此对于小图片的测试时扔掉深层部分模型对前面的输出完全没有影响的。但在训练阶段,因为既有前向,又有反向传播,被剪掉的部分是会帮助其他部分做权重更新的。所以加入剪枝操作可以提升模型的测试速度,这也是有实验证明了的。
2、多尺度跳跃连接:
可以抓取不同层次的特征,将它们通过特征叠加的方式整合,不同层次的特征,或者说不同大小的感受野,对于大小不一的目标对象的敏感度是不同的,比如,感受野大的特征,可以很容易的识别出大物体的,但是在实际分割中,大物体边缘信息和小物体本身是很容易被深层网络一次次的降采样和一次次升采样给弄丢的,这个时候就可能需要感受野小的特征来帮助.而UNet++就是拥有不同大小的感受野,所以效果好.
好吧,这个模型又经过其他大佬修改提出了U-Net3+模型,Unet++存在的不足就是增加了模型的参数量,从模型结构就可以看出,它比Unet多了很多中间节点;其次就是它缺乏全尺度探索足够信息的能力,即低层模型结构没有加入深层特征图的内容。这也就是UNet3+的创新点。
四、Unet3+模型
Unet3+根据Unet++的不足一一进行了改进,它的创新点就是Unet++存在的不足。
4.1、改进的跳跃连接(全尺度跳跃连接)
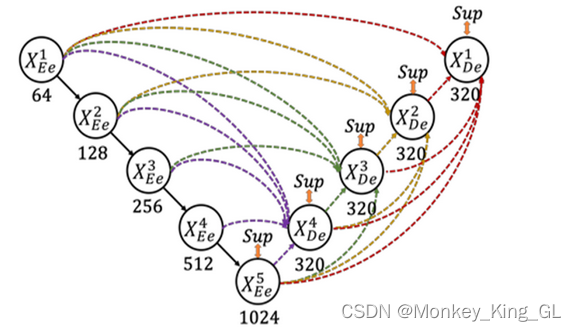
无论是连接简单的UNet,还是连接紧密嵌套的UNet++,都缺乏从全尺度探索足够信息的能力,未能明确了解器官的位置和边界。而Unet3+就去掉了Unet++的稠密卷积块,而是提出了一种全尺寸跳跃连接。全尺寸跳跃连接改变了编码器和解码器之间的互连以及解码器子网之间的内连接,让每一个解码器层都融合了来自编码器中的小尺度和同尺度的特征图,以及来自解码器的大尺度的特征图,这些特征图捕获了全尺度下的细粒度语义和粗粒度语义。
例如,下图是X3De特征图的生成过程,它融合了编码器的小尺度特征图X1Ee、X2Ee、解码器的大尺度特征图X4DeX5De和同尺度的特征图X3Ee。但因为这些特征图与X3Ee的特征图尺寸和通道数不一致,因此小尺度的特征图需要经过下采样相应的倍数,大尺度的特征图需要上采样相应的倍数才可以和X3Ee的特征图进行融合,然后再经过320个3*3的卷积核进行卷积,最后经过BN+ReLU操作得到X3De的特征图,实现全尺度特征融合。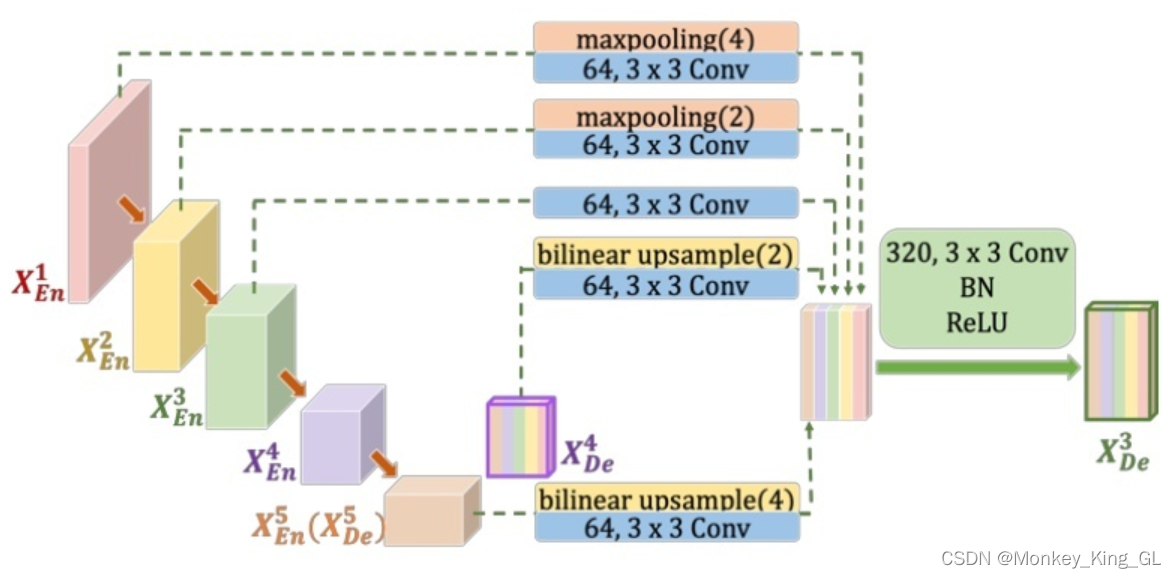 解码器其他部分的特征图也是按照同样方式得到的。具体的公式表示如下。其中,函数C表示卷积操作,函数H表示特征聚合机制(一个卷积层+一个BN+一个ReLU),函数D和函数U分别表示上采样和下采样操作,[ ]表示通道维度拼接融合。
解码器其他部分的特征图也是按照同样方式得到的。具体的公式表示如下。其中,函数C表示卷积操作,函数H表示特征聚合机制(一个卷积层+一个BN+一个ReLU),函数D和函数U分别表示上采样和下采样操作,[ ]表示通道维度拼接融合。
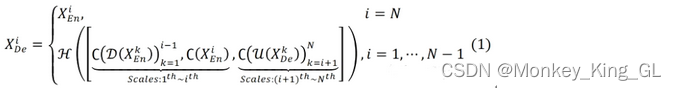
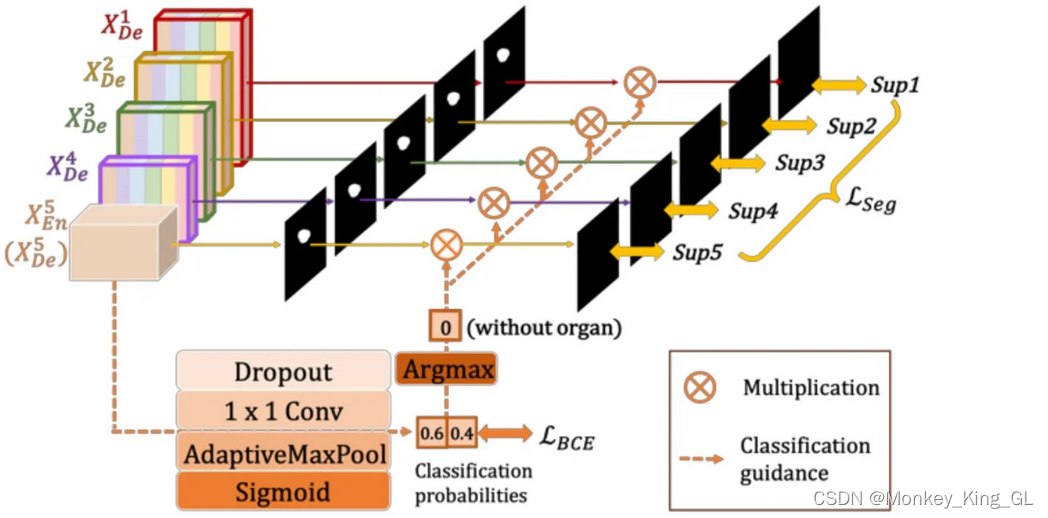
4.2 全尺度深度监督
Unet3+的全尺度深度监督与UNet++中的深度监督不同之处在于监督的位置不同,前者监督的是网络解码器每个阶段输出的特征图,后者监督的是网络第一层中的四张特征图(其中三张为跳跃连接中卷积块的输出特征图,一张为解码器最后输出的特征图)。此外,在UNet3+中,为了实现深度监督,每个解码器阶段的最后一层被送入一个普通的3×3卷积层,然后是一个双线性上采样和一个sigmoid函数(这里的上采样是为了放大到全分辨率)。
4.3 分类指导模块(Classification-guided Module,CGM)
该模块的提出是为了解决医学图像分割过程中非器官图像出现假阳性的现象(意思就是输入一张没有目标器官的图像,经过模型测试,结果显示存在目标器官的假象)。这个分类指导模块就是将模型的最深层经过dropout,卷积,sigmoid等一系列的操作之后得到一个是否有目标器官的概率,再通过Argmax函数的帮助下得到一个{0,1}的单个输出,进而指导每一个切分侧边的输出。
U-Net3+的创新点:
1、降低了模型参数量,模型结构更加简洁:
Unet++模型虽然较Unet模型融入了更多特征信息,使模型更加精准,但其也增加了网络结构的参数量,导致模型训练和运行速度降低了很多。而UNet3+不仅保留了Unet++的优秀特性,还删除了中间节点减少了模型参数量,使模型结构更加简洁。
2、全尺度跳跃连接:
全尺寸跳跃连接改变了编码器和解码器之间的互连以及解码器子网之间的内连接,让每一个解码器层都融合了来自编码器中的小尺度和同尺度的特征图,以及来自解码器的大尺度的特征图,这些特征图捕获了全尺度下的细粒度语义和粗粒度语义。
3、分类指导模块:
分类指导模块给模型又提供了一个参考对象,通过该模块可以避免因噪声数据和过度分割导致的假阳现象。
五、总结
 从上面的学习我们可以知道Unet3+的性能是优于Unet和Unet++的,但是无论是Unet、Unet++还是Unet3+他们都是作者们经过多次实验得出的结论,都是值得我们学习的,他们都有各自的闪光点。Unet除了这两个变形之外还有很多很多其他的变形模型,他们一起被统称为UNet-Family。
从上面的学习我们可以知道Unet3+的性能是优于Unet和Unet++的,但是无论是Unet、Unet++还是Unet3+他们都是作者们经过多次实验得出的结论,都是值得我们学习的,他们都有各自的闪光点。Unet除了这两个变形之外还有很多很多其他的变形模型,他们一起被统称为UNet-Family。
UNet是一个经典的网络设计方式,在图像分割任务中具有大量的应用。也有许多新的方法在此基础上进行改进,融合更加新的网络设计理念,但目前几乎没有人对这些改进版本做过比较综合的比较。由于同一个网络结构可能在不同的数据集上表现出不一样的性能,在具体的任务场景中还是要结合数据集来选择合适的网络。


