热门标签
热门文章
- 1机器学习---迁移学习
- 2MATLAB中FFT_fft得到特定频率信号的复数是什么
- 3再升级!视频理解大模型 CogVLM2 开源
- 4微软重磅开源 GraphRAG:新一代 RAG 技术来了!
- 5mongoDB教程(十二):分页操作
- 6基于node.js和hexo框架搭建的个人博客项目_hexo 支持doc
- 7python matplotlib模块 savefig图像清晰度差方法(本人实测有效)_matplotlib savefig分辨率太低
- 8Python前沿技术,机器学习与人工智能的应用_python在人工智能和机器学习中的应用
- 9github.io搭配(阿里云/xx)域名部署_github.io配置域名
- 10接口测试工具Apifox介绍_apifox的 定时任务卡
当前位置: article > 正文
limma 小样本量的差异表达分析教程_微阵列数据做limma差异分析
作者:知新_RL | 2024-07-21 07:30:34
赞
踩
微阵列数据做limma差异分析
本教程将使用R中的simulated数据集,模拟100个探针和6个微阵列的基因表达数据。这些微阵列分为两组,其中前两个探针在第二组中呈差异表达。基因的标准差在不同基因之间变化,先验的自由度为4。
1. 导入必要的库和数据
# 导入必要的库
library(limma)
# 设定随机数生成种子以确保结果可重复
set.seed(123)
# 模拟基因表达数据
sd <- 0.3 * sqrt(4 / rchisq(100, df = 4))
y <- matrix(rnorm(100 * 6, sd = sd), 100, 6)
rownames(y) <- paste("Gene", 1:100)
y[1:2, 4:6] <- y[1:2, 4:6] + 2
# 创建设计矩阵
design <- cbind(Grp1 = 1, Grp2vs1 = c(0, 0, 0, 1, 1, 1))
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
2. 普通的线性模型拟合
# 拟合线性模型
fit <- lmFit(y, design)
# 进行贝叶斯调整
fit <- eBayes(fit)
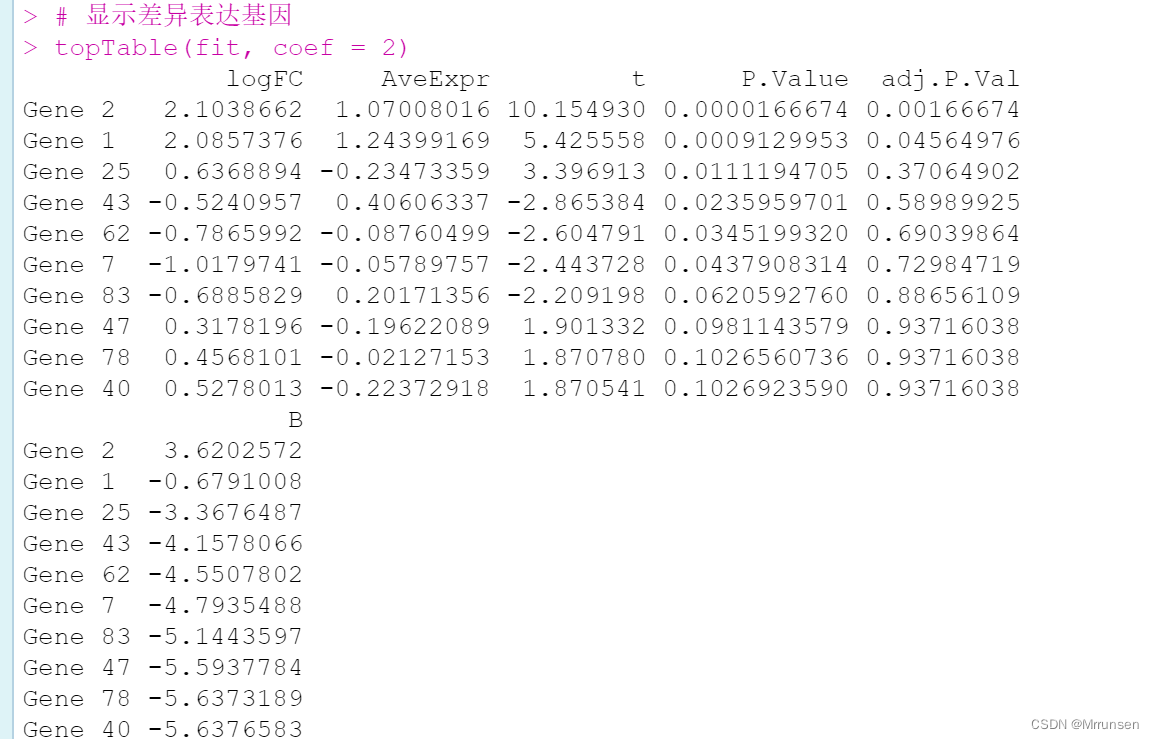
# 显示差异表达基因
topTable(fit, coef = 2)
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
3. 折叠变化阈值法
# 使用折叠变化阈值法拟合
fit2 <- treat(fit, lfc = 0.1)
# 显示差异表达基因
topTreat(fit2,- 1
- 2
- 3
- 4
声明:本文内容由网友自发贡献,不代表【wpsshop博客】立场,版权归原作者所有,本站不承担相应法律责任。如您发现有侵权的内容,请联系我们。转载请注明出处:https://www.wpsshop.cn/w/知新_RL/article/detail/860043
推荐阅读
相关标签


