- 1[错误解决]Ubuntu中使用dpkg安装deb文件提示依赖关系问题,仍未被配置
- 2Java接入ChatGPT API
- 3Huggingface使用_tokenizer checkpoint是
- 4SpringBoot和SpringCloud版本问题_spring-cloud-starter-netflix版本2.3
- 5IDEA导入项目无法识别pom.xml文件,Java类为黄色J标识,找不到maven视图_idea中的maven读不到pom
- 6iOS——分类、扩展和关联对象_ios 分类
- 7乐信—高级Java开发工程师二面(偏业务)_乐信集团面试题
- 8mac静态路由设置_mac route
- 9CImage 类及其常用成员函数用法实例详解 一
- 10大语言模型微调框架Unsloth:简化模型微调流程,提升模型性能
TILs 评分:TCGA 肿瘤浸润淋巴细胞病理切片深度学习评分!图片下载与可视化
赞
踩
生信碱移
病理切片的TILs评分
TCGA 数据库是最大的肿瘤组学公开数据库之一。尽管如此,更多的研究往往仅局限于关注 TCGA 中各类肿瘤样本的上游组学信息或基本病理特征,而忽略了对样本数字化 H&E 病理染色图像的进一步应用。
▲ TCGA中肿瘤样本的病理染色切片
早在 2018 年一篇的 Cell Reports [IF:7.5] 文章中,研究人员便针对 13 种 TCGA 肿瘤类型的 H&E 图像,使用卷积神经网络对图像块进行建模,预测了切片区域内肿瘤浸润淋巴细胞(TILs)的映射强度。在他们的结果中,不同肿瘤类型、免疫亚型和肿瘤分子亚型的 TIL 密度和空间结构存在差异,这表明空间浸润 TILs 评分可能反映特定肿瘤细胞的异常状态。

▲ DOI: 10.1016/j.celrep.2018.03.086
最近看到不少文章使用这个 TILs 评分,但只是看起来唬人。由于13 种肿瘤的 TILs 评分都已经计算出来了,所以直接应用就好。这 13种肿瘤分别是:
-
BRCA: 乳腺癌 (Breast Invasive Carcinoma)
-
LUAD: 肺腺癌 (Lung Adenocarcinoma)
-
UCEC: 子宫内膜癌 (Uterine Corpus Endometrial Carcinoma)
-
COAD: 结肠癌 (Colon Adenocarcinoma)
-
LUSC: 肺鳞癌 (Lung Squamous Cell Carcinoma)
-
SKCM: 皮肤黑色素瘤 (Skin Cutaneous Melanoma)
-
STAD: 胃腺癌 (Stomach Adenocarcinoma)
-
PRAD: 前列腺癌 (Prostate Adenocarcinoma)
-
BLCA: 膀胱癌 (Bladder Urothelial Carcinoma)
-
CESC: 宫颈癌和子宫颈鳞状细胞癌 (Cervical Squamous Cell Carcinoma and Endocervical Adenocarcinoma)
-
PAAD: 胰腺癌 (Pancreatic Adenocarcinoma)
-
READ: 直肠癌 (Rectum Adenocarcinoma)
-
UVM: 葡萄膜黑色素瘤 (Uveal Melanoma)
这篇文章中,小编将讲讲如何下载指定样本的图片,以及计算不同亚型间的 TILs评分差异(注意,仅适用于上面的13种肿瘤)。示例数据+代码的网盘链接就放在公众号后台,铁子们发送 TIL病理评分 即可获得。可以产生的结果如下
▲ 最终的示例结果如上图所示。
一、计算亚型间的TIL差异
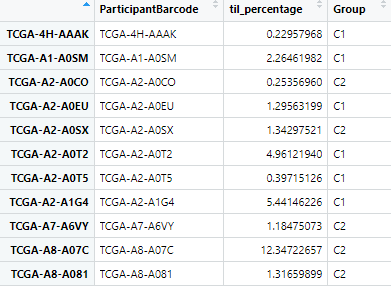
① 这里给大家举的是一个例子:如果我们做分型研究,分好了不同的肿瘤样本,如何比较 TIL 得分在不同分组病人中的差异。提供的示例文件名称分别是sample.txt、til_percentage.txt,内容展示如下图:
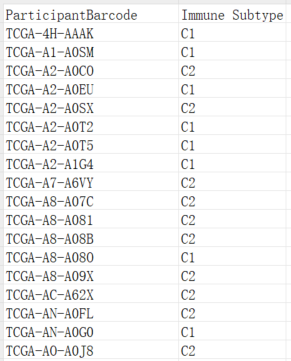
▲ Tab 键分隔的样本属性文件sample.txt:第一列是病人的ID,第二列是我们预先分好的病人亚型,每一列的标题不需要修改。

▲ Tab键分隔的TIL得分文件til_percentage.txt:这个是从网站中直接下载的,第一列是样本ID,第二列是TIL得分,总共包含上述提到的13个癌种,大家直接使用就好。
② 可视化以及差异检验代码如下:
- library(ggpubr)
-
- # 读入数据
- til <- read.table("til_percentage.txt", header = T, sep = "\t")
- sample <- read.table("sample.txt", header = T, sep = "\t")
- com_id <- intersect(til$ParticipantBarcode, sample$ParticipantBarcode)
- rownames(til) <- til$ParticipantBarcode
- rownames(sample) <- sample$ParticipantBarcode
- sample <- sample[com_id, ]
- til <- til[com_id, ]
- til$Group <- sample$Immune.Subtype
-
- # 设置比较组
- my_comparisons <- list( c("C1", "C2"))
- ggviolin(til, x = "Group", y = "til_percentage", fill = "Group",
- palette = c("#00AFBB", "#E7B800"),
- add = "boxplot", add.params = list(fill = "white"))+
- stat_compare_means(comparisons = my_comparisons, label = "p.signif")+ # Add significance levels
- stat_compare_means(label.y = 50)
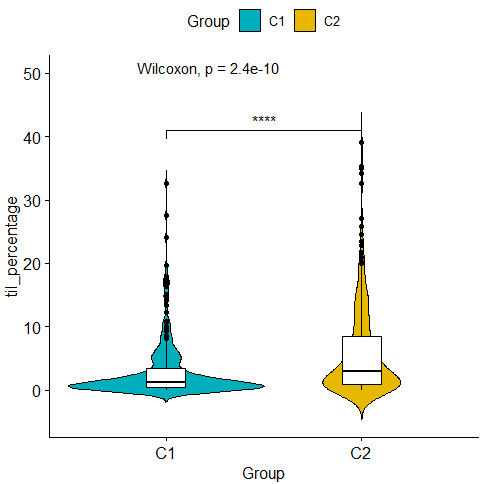
③ 看看不同分组病人的具体得分(这里可以看到哪些病人得分高,选几个差异大的等会下载其结果图片):
View(til) # 可以用来看看不同分组病人的id
二、下载指定样本的病理图片
① 浏览器中进入下述网址:
-
https://cancerimagingarchive.net/datascope/TCGA_TilMap/
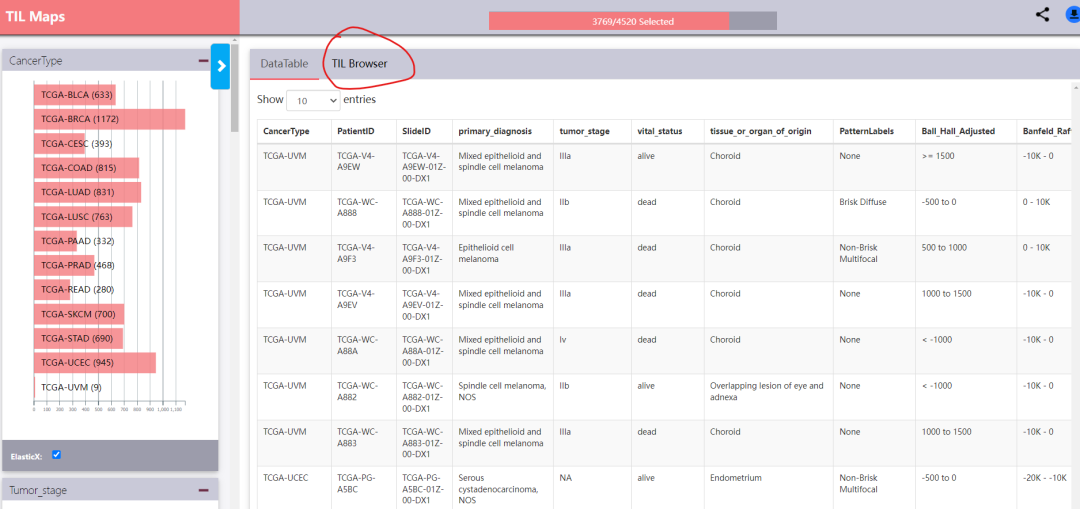
② 点击下方截图中的"TIL Browser":
③ 然后在搜索框中输入病人id:
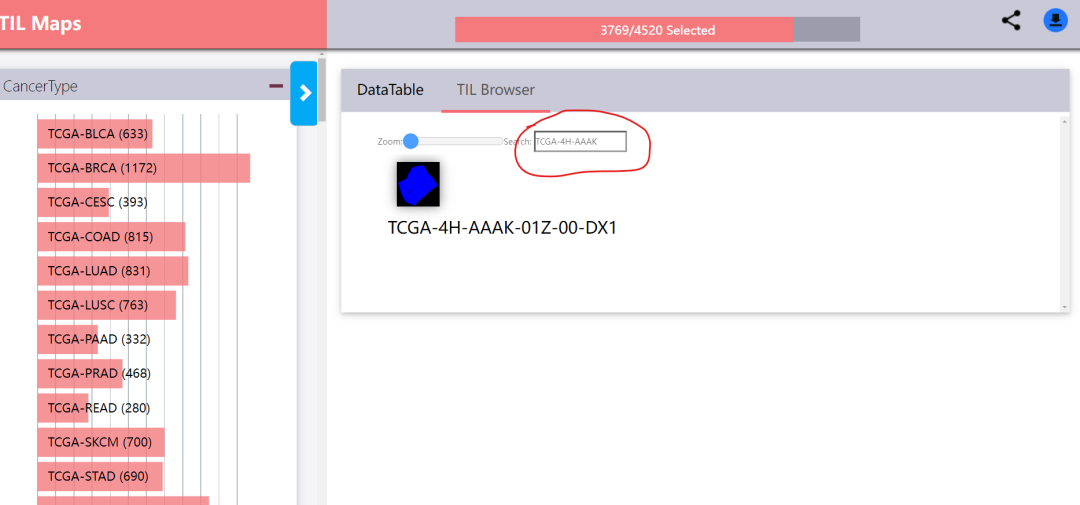
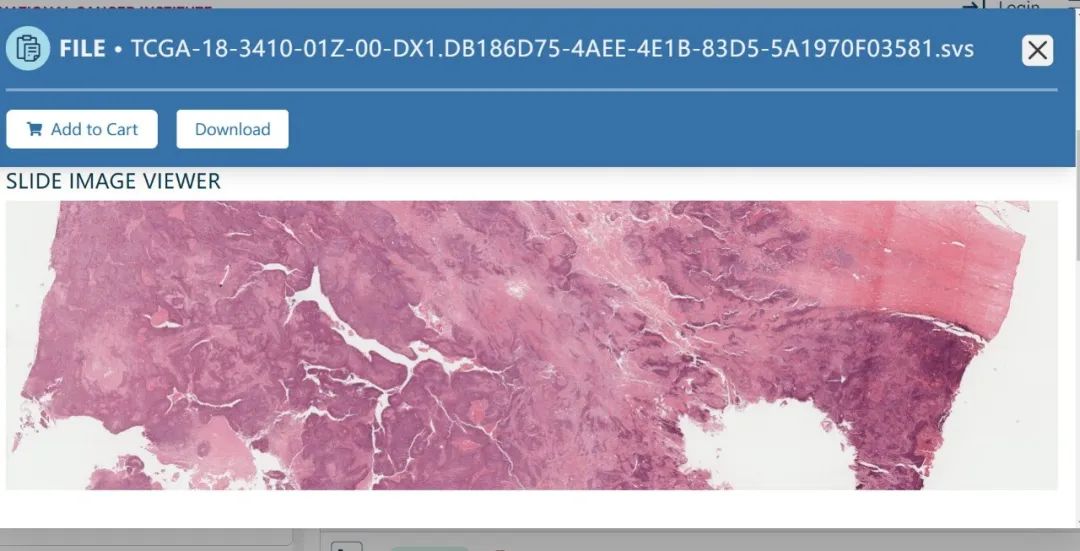
④ 鼠标移至图片后,右键点击"图片另存为"即可进行下载:

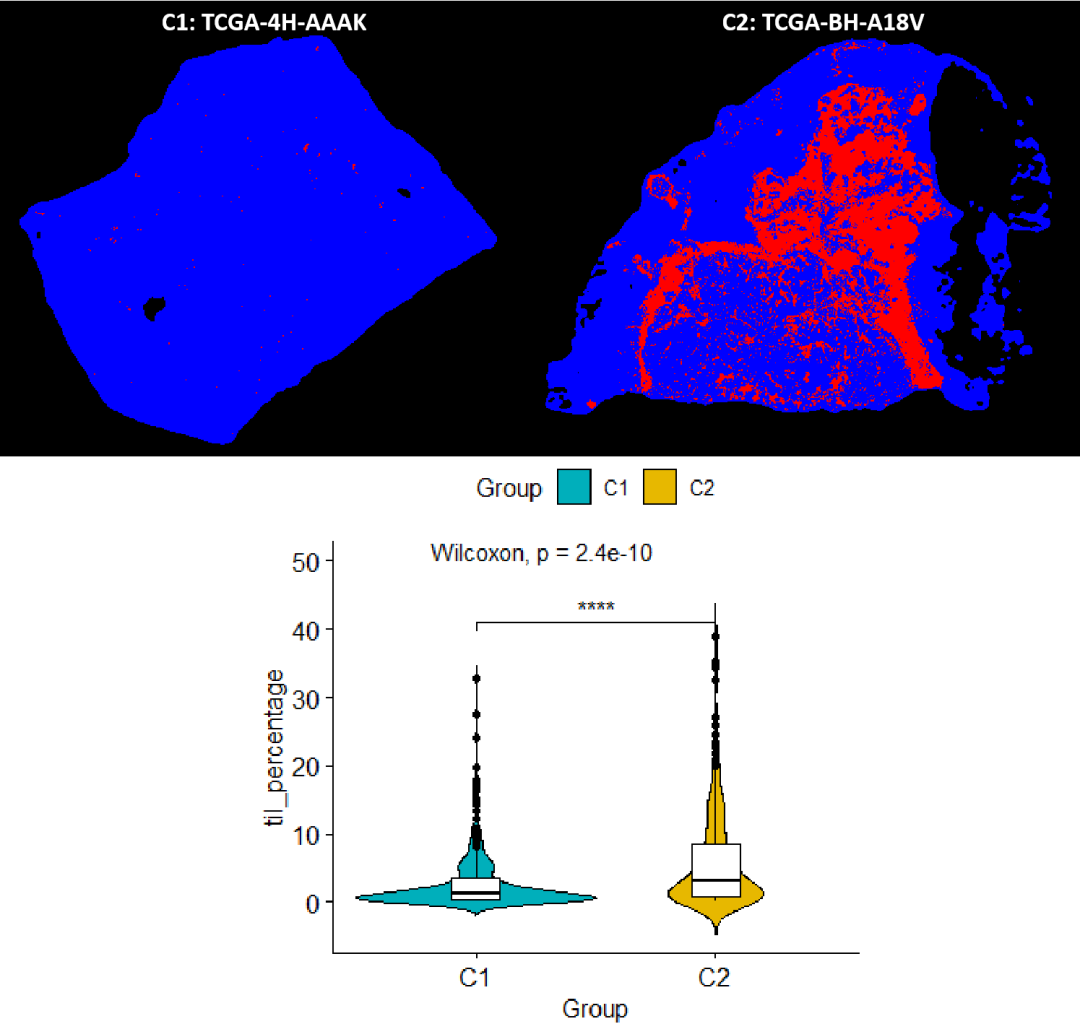
⑤ 这里我的C1、C2分别下载的是TCGA-4H-AAAK与TCGA-BH-A18V,将两张图片简单排版即可展示如下:

从图片来看,我们的结论是 C2 的样本肿瘤浸润淋巴细胞更多,而 C1 相对更少。
欢迎各位关注
就分享到这里了
