- 1ElasticSearch 搜索引擎_es搜索引擎
- 2计算机二级office学习之Excel操作题考点整理_计算机二级考试excel操作题
- 3【24届数字IC秋招总结】提前批面试经验1——小米、百度昆仑芯、长鑫存储
- 4linux下的shell脚本(基本)
- 5在一个py文件里调用另一个py文件,使调用的py文件后台运行_在.py里面运行另外一个.py
- 6详解 async/await :应用场景,示例代码,注意事项
- 7kafka监控工具Kafka-eagle安装_kafka 监控工具
- 8亚信安慧AntDB:开启数据洞察的新视野
- 9Mysql各种特殊时间段查询方法_mysql 时间段查询
- 10某东员工面试被问自动化测试,11:00进去的,11:10就出来······
【项目实战】Python实现深度神经网络RNN-LSTM分类模型(医学疾病诊断)
赞
踩
说明:这是一个机器学习实战项目(附带数据+代码+视频+文档),如需数据+完整代码可以直接到文章最后获取。
1.项目背景
随着互联网+的不断深入,我们已步入人工智能时代,机器学习作为人工智能的一个分支越来越多地被应用于各行各业,其中在临床医学检测中也得到了越来越多的应用。基于临床医学越来越多的检测数据,通过建立一个机器学习模型来进行更加智能地预测已成为当今时代的使命。本模型也是基于一些历史的疾病数据进行建模、预测。
2.收集数据
本数据是模拟数据,分为两部分数据:
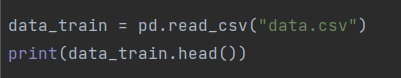
训练数据集:data.csv
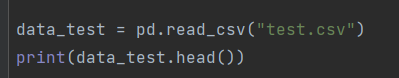
测试数据集:test.csv
在实际应用中,根据自己的数据进行替换即可。
特征数据:age、gender、body_mass_index、heart_failure hypertension、 chronic_obstructic_pulmonary_disease、 chronic_liver_disease、……renal_toxic_drug
标签数据:acute_kidney_disease
3.数据预处理
1)原始数据描述:
2)数据完整性、数据类型查看:
3)数据缺失值个数:

可以看到数据不存在缺失值。
4.探索性数据分析
1)显示age特征的分布情况:
2)显示gender特征的分布情况:
3)显示heart_failure特征的分布情况:
剩下的其它特征,可以自行分析。
4)相关性分析

说明:正值是正相关、负值时负相关,值越大变量之间的相关性越强。
5.特征工程
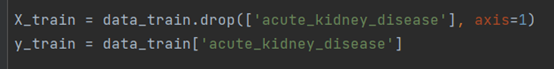
1)特征数据和标签数据拆分,acute_kidney_disease为标签数据,除acute_kidney_disease之外的为特征数据;
2)数据集拆分,分为训练集和尝试集
数据集已提前分好,直接读取即可。
6.LSTM建模
1)神经网路LSTM简单介绍:
LSTM网络是RNN的一个变体,也是目前更加通用的循环神经网络结构,全程为Long Short-Term Memory,翻译成中文叫作”长 ‘短记忆’”网络。读的时候,”长”后面要稍作停顿,不要读成”长短”记忆网络,因为那样的话,就不知道记忆到底是长还是短。本质上,它还是短记忆网络,只是用某种方法把”短记忆”尽可能延长了一些。
简而言之,LSTM就是携带一条记忆轨道的循环神经网络,是专门针对梯度消失问题所做的改进。它增加的记忆轨道是一种携带信息跨越多个时间步的方法。可以先想象有一条平行于时间序列处理过程的传送带,序列中的信息可以在任意位置”跳”上传送带,然后被传送到更晚的时间步,并在需要时原封不动地”跳”过去,接受处理。这就是LSTM原理:就像大脑中的记忆存储器,保存信息以便后面使用,我们回忆过去,较早期的信息就又浮现在脑海中,不会随着时间的流逝而消失得无影无踪。
这个思路和残差连接非常相似,其区别在于,残差连接解决的是层与层之间得梯度消失问题,而LSTM解决的是循环层与神经元层内循环处理过程中的消息消失问题。
简单来说,C轨道将携带着跨越时间步的信息。它在不同的时间步的值为Ct,这些信息将与输入连接和循环连接进行运算(即与权重矩阵进行点积,然后加上一个偏置,以及加一个激活过程),从而影响传递到下一个时间步的状态如右图所示。
LSTM-增加了一条记忆轨道,携带序列中较早的信息
2)建立LSTM分类模型,模型参数如下:
编号
参数
1
loss='binary_crossentropy'
2
optimizer='adam'
3
metrics=['acc']
其它参数根据具体数据,具体设置。
3)神经网络结构及概要
神经网络结构图:
神经网络概要:
可以看到每层网络的类型、形状和参数。
7.模型评估
1)评估指标主要采用查准率、查全率、F1
编号
评估指标名称
评估指标值
1
查准率
98.74%
2
查全率
100.00%
3
F1
99.37%
通过上述表格可以看出,此模型效果良好。
2)损失和准确率图
3)ROC曲线绘制
训练集ROC曲线图:
测试集ROC曲线图:
8.临床应用
根据测试集的特征数据,来预测这些患者是否会有相关疾病;根据预测结果:针对将来可能会患有此种疾病的人员,提前进行预防。
预测结果如下:
- features = ['age']
- fig = plt.subplots(figsize=(15, 15))
- for i, j in enumerate(features):
- plt.subplots_adjust(hspace=1.0)
- sns.countplot(x=j, data=data_train)
- plt.title("No. of age")
-
- # 本次机器学习项目实战所需的资料,项目资源如下:
-
-
-
- 链接:https://pan.baidu.com/s/1PE58j5RizuobkojAsSFwGg
- 提取码:5gnl
-
-
-
-
- fig = plt.subplots(figsize=(15, 15))
- for i, j in enumerate(features):
- plt.subplots_adjust(hspace=1.0)
- sns.countplot(x=j, data=data_train)
- plt.title("No. of gender")