- 1Win7系统怎么改变Alt+Tab切换窗口数量,改变Alt+Tab切换窗口数量的方法_win10系统怎么改变alt+tab切换窗口数量,改变alt+tab切换窗口数量的方法
- 2AltiumDesigner VS 立创EDA —— PCB绘图软件选择_初学者选立创eda还是ad
- 3Hadoop大数据技术课程设计说明_大数据技术与应用课程设计
- 4学校教职工用计算机台数指标,陕西省义务教育学校办学基本标准的主要指标
- 5mac 版 Nginx 的使用及 web 项目的实际部署--最简单的方式_mac 启动nginx
- 6前端工程化 构造简单的微信小程序开发脚手架工具_plop temp
- 7人脸识别算法学习eigenfaces理论分析
- 8Android studio——Logcat(日志)的使用_android studio logcat
- 9日常学习总结_运维学习每日报告
- 10职高计算机专业c语言_职高计算机专业都学什么
nnUNet(代码)-预处理
赞
踩
任务一:(论文)Automated Design of Deep Learning Methods for Biomedical Image Segmentation // nnU-Net: a self-configuring method for deep learning-based biomedical image segmentation Fabian
1.背景动机:
简单来说医疗数据尤其是在3D医学图像领域,遇到图片质量、图片模态、图片大小、体素大小、类别比率差别都会产生巨大的效果落差。大部分专业的人士做的事情,都是从网络结构的优化设计,到数据增强和后处理。还有另一个问题就是训练和推理对于硬件的需求。费劲研究寻找更好的模型不如好好调参。
最后,我们提出了nnUNet,最终这个网络使得医学研究的实际应用成为了可能,至于这个框架为什么可以适应任意的数据集并且能够开箱即用,主要缘于以下两点:
1.我们用公式来明确表示与数据的关键属性有关的“管道”(方法)优化问题和分割算法的一些关键的设计选择;
数据指纹(data fingerprint):(表示数据集的关键属性)
管道指纹(pipeline fingerprint):(表示‘管道’关键的优化设计)
2.我们通过将大多数的理论知识集成为一种启发性规则,来使得上面两者的关系更加的准确,这个规则可以从原有的data fingerprint中生成出pipeline fingerprint(即我可以通过数据的性质来推出它可能需要进行什么样子的训练),同时将硬件的局限条件考虑进去。
学习内容:看懂nnunet体系化的调参过程
···(加一步预处理simpleitk):转变成nnunet可识别的nii.gz格式:
包括标签修改,格式转换
import cv2 import SimpleITK as sitk import matplotlib.pyplot as plt import numpy as np import os # train_path = 'subset0' label_path = 'seg-lungs-LUNA16' paths = os.listdir(train_path) lpaths = os.listdir(label_path) #print(paths) def Md_image(data): # 获取图像参数信息 messagesize = data.GetSize() spacing = data.GetSpacing() # print("Image spacing:", spacing) direction = data.GetDirection() # print("Image direction:", direction) origin = data.GetOrigin() # print("Image origin:", origin) # Numpy矩阵数据转成SimpleITK图像数据 a = sitk.GetArrayFromImage(data) a[a==3]=1 a[a==4]=2 a[a==5]=3 image_a = sitk.GetImageFromArray(a) image_a.SetOrigin(origin) image_a.SetSpacing(spacing) image_a.SetDirection(direction) return image_a for i, path in enumerate(paths): if path.find('mhd')>=0: # 训练集部分 data = sitk.ReadImage(os.path.join(train_path,path)) # 读取每一个mhd # 标签部分 ldata = sitk.ReadImage(os.path.join(label_path,path)) ldata_images = Md_image(ldata) # 存入不同文件夹 i=int(i/2) #sitk.WriteImage(data, 'imagesTr/'+str(i)+'_0000.nii.gz') # _0000是ct模式的格式 sitk.WriteImage(ldata_images, 'labelsTr/' + str(i) + '.nii.gz') print(i)
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
- 54
2.nnU-Net automatically adapts to any new dataset:
· nnUNet将数据集标准化(数据指纹):图像大小,体素间距信息或类比等关键属性,以及在方法设计过程中做出的全部选择中的流水线指纹。
· nnU-Net旨在为给定的数据集指纹生成成功的管道指纹(一系列的启发规则)
管道指纹:
1.蓝图参数:代表基本的设计选择(例如,使用简单的类似U-Net的体系结构模板)以及超参数,可以针对这些超参数简单地选择可靠的默认值(如图包括,损失函数,最优化,训练计划,结构模板,数据增强和训练时间)。
2.推断参数:对新数据集的必要适应进行编码,并包括对确切的网络拓扑,图像重采样,图像归一化,补丁大小,批次大小和图像预处理的修改。(数据指纹和推断的参数之间的链接是通过执行一组启发式规则建立的,当应用于看不见的数据集时,无需进行昂贵的重新优化)
3.经验参数:通过对训练案例进行交叉验证,可以自动确定经验参数
· 默认情况下,nnU-Net会生成三种不同的U-Net配置:
① 2D、3D和3D_Cascaded三个网络分别训练,得出各自的模型(三个网络结构共享一个“管道指纹”,五折交叉验证)
在进行训练时,经验参数会通过交叉验证被确定
② 每进行一次训练,nnUNet都会为此创建三个不同的配置:
2D、3D、3D_cascade
2D_Unet: 普通的2D_Unet
3D_Unet: 对一整张图片像素进行操作
3D_cascade: 级联网络:第一个网络对下采样图片进行操作,而第二个网络对前一个网络产生的结果在整个图片的像素上进行调整。
· 选择出最优的模型进行推理
在进行完交叉验证之后,nnUNet会经验性的选择表现最好的参数,可能是独个的推理结果,也可能是一起推理的结果。在结果可以评估的情况下,把对次优效果的抑制作为一项后处理操作。



提出了基于深度学习的生物医学图像分割的自动方法配置。给定一个新的分段任务,数据集属性以“数据集指纹”(粉红色)的形式提取。一组启发式规则为参数相互依赖性(如细箭头所示)建模,并对该指纹进行操作,以推断管道中依赖于数据的“基于规则的参数”(绿色)。这些由“固定参数”(蓝色)补充,这些参数是预定义的,不需要调整。在五重交叉验证中,最多培训了三种配置。最后,nnU网络自动对这些模型的最优集合进行经验选择,并确定是否需要后处理(“经验参数”,黄色)。底部的表格显示了所有配置参数的显式值以及总结的规则公式。
任务二.:结合代码
1.(预处理)experiment_planning/nnUNet_plan_and_preprocess.py:
step 1 : crop(task_name, False, tf)# 剪枝操作
Crop操作(可以大量减少数据size):数据,是否重写(override),线程数
设置多线程用进程池Pool函数
1.零点填充:需要对data进行分割训练部分(True)和不需要训练的背景部分(False),需要对不同通道进行融合,生成nonzero_mask,主要用到binary_fill_holes,函数create_nonzero_mask。
2.裁剪边缘为0的数值:get_bbox/crop_bbox
总结:用处主要是为了适应一些不规则的数据集,包含了对不同模态的合并,还有一些设置,比如返回存储的data要是float32
step 2 : dataset_analyze(数据分析)
dataset_analyzer = DatasetAnalyzer(cropped_out_dir, overwrite=False, num_processes=tf) # 得到数据指纹
两种对于数据指纹的处理方式
if planner_3d is not None:
exp_planner = planner_3d(cropped_out_dir, preprocessing_output_dir_this_task)
exp_planner.plan_experiment()
if not dont_run_preprocessing: # double negative, yooo
exp_planner.run_preprocessing(threads)
if planner_2d is not None:
exp_planner = planner_2d(cropped_out_dir, preprocessing_output_dir_this_task)
exp_planner.plan_experiment()
if not dont_run_preprocessing: # double negative, yooo
exp_planner.run_preprocessing(threads)
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
planner_3d相当于模块experiment_planner_baseline_3DUNet_v21.py(就3d过程进行分析):
- 分析一部分超参数
##############参数dataset_properties.pkl里面的内容(预处理过程需要用的):
all_sizes:保存经过crop后每个样本的size
all_spacings:保存经过crop后每个样本的spacing
intensityproperties:密度信息,这个很重要
关键步骤是获取每个样本的前景像素信息:
1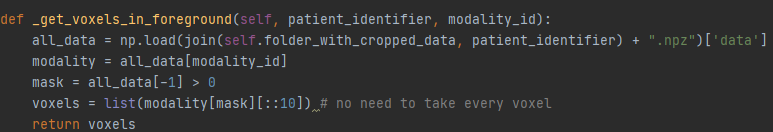
median:基于前景的中值
mean:均值
sd, mn, mx:标准差,最小值,最大值
percentile_99_5, percentile_00_5:百分位为99.5和0.05的数
并且基于每个样本的这些密度数据和基于所有数据的这些密度信息都求一份进行保存。
###############关注experiment_planner_baseline_3DUNet.py(构建网络需要用的,plan里面的内容):
batch_size:基准是2,根据ref/here,如果网络需要的内存here过小,batch_size才会增大,再检查一遍它是否过大。
network_num_pool_per_axis:对于patch_size:[24,504,512]。计算它每个维度的值除以min_feature_map_size=4,然后求其是2的i次方(i向下取整,计算规则在下面的网络模型选择里面)
input_patch_size:首先与基准的512×512×512投影到图片上patch比较不能比基准大,后面计算here(当前网络需要的显存)与ref(8g的基准显存)比较然后更新。
current_spacing:统一后的体素间距,也就是target_spacing,取所有spacing的百分之五十的位数
original_spacing:初始的体素间距
do_dummy_2D_data_aug:阈值判断是否需要数据增强 - 网络模型选择:
根据一种规则计算出网络最深的层数:common_utils.py/get_pool_and_conv_props_poolLateV2

i是patch_size每个维度的值,min_f=4(最小的featrure_map确认长宽在4以上),可以根据patch_size值得出每个维度需要的pool次数,kernel次数等于pool次数加一。
后续计算需要用到的是根据三个维度的patchsize计算每个维度需要进行网络训练的深度,然后根据这些深度进行迭代计算poolsize以及kernelsize。
pool_op_kernel_sizes:循环根据规则值得出每个维度需要的pool_size大小(2/1)
由于是三维的,对每个维度上的核大小都进行选择:
pool = [2 if num_pool_per_axis[i] + p >= net_numpool else 1 for i in range(dim)]
p是网络的深度,每个维度上,对于这个维度计算的深度(num_pool_per_axis[i])加上当前深度如果大于计算的最大深度(net_numpool),那么这个维度上pool_size为2否贼为1。
conv_kernel_sizes:根据体素反应出的感受野进行选择每个维度上的kernel_size(3/1)
对每个维度进行选择核的大小:
[current_spacing[i] / reach > 0.5 for i in range(dim)]
conv = [3 if not reached[i] else 1 for i in range(dim)]
每个维度上,对于这个维度的spacing(current_spacing[i])除以三个维度中最大的spacing(reach),如果大于0.5那么卷积核为3否则为1。
current_spacing = [i * j for i, j in zip(current_spacing, pool)]
对于每个深度的current_spacing值是变动的,迭代完一个深度的计算后,spacing的每个维度值将乘以前面做的pool的大小。
feature_map_num
初始通道数为30pooling一次乘以对应的pool值
step 3: resample_and_normalize
重采样
需要的输入:数据(data),标签(seg),之前数据分析的一些参数。
真实的图片大小=体素间距(sapcing)*像素值
1.重采样过程就是统一体素间距的过程,也可以说是统一分辨率的过程,确定了target_spacing,根据公式容易计算出新的像素值大小。
2.再进一步根据skimage的resize函数对图像的大小进行改变:这里对于重新计算出的每个维度的像素值进行判断,对于最大维度值大于最小维度值的三倍以上的情况进行特殊的分割处理(这种特异性情况会影响训练效果)。
3.对与data与seg都进行resize操作:但是不同处在于seg的值是确定的012等情况,resize后难免出现小数,这时候会不好判断阈值,这里的思想是对于每类标签进行二分类的resize判断(跟网络的分割思想相似),对于大于0.5的为本次迭代的标签值。
4.注意重采样后的data可能出现nan,以及seg可能出现小于0的值,这时候需要进行排除。
归一化
输入:数据(data),前景的密度信息(dataset_properties.pkl)
mean_intensity = self.intensityproperties[c]['mean']
std_intensity = self.intensityproperties[c]['sd']
lower_bound = self.intensityproperties[c]['percentile_00_5']
upper_bound = self.intensityproperties[c]['percentile_99_5']
data[c] = np.clip(data[c], lower_bound, upper_bound)
data[c] = (data[c] - mean_intensity) / std_intensity
if use_nonzero_mask[c]:
data[c][seg[-1] < 0] = 0
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
先进行窗宽窗位调整,后进行归一化。
学习结果与计划:
1.还有很多训练过程代码细节完成,以及拿到训练plan再进行推理生成结果。
2.继续看代码,以及论文增加见识,后面两周到考试前开始复习矩阵论和组合数学。
3.这篇内容还没完成以后逐渐完善。



