- 1【2023华为od机考】算法题答案记录100分题_华为前端od算法题
- 2复制Anaconda虚拟环境_copy anaconda huanjing
- 3DNS(UOS)_uos dns
- 4云主机安装宝塔面板注意事宜_宝塔的node终端换源
- 5Windows 11 的C盘User(用户)文件夹下的用户文件夹名称的修改_win11更改c盘user用户名
- 6PDD根据ID取商品详情 API_funimg pddpic
- 7CKA认证,开启您的云原生之旅!
- 8Linux命令200例:mc一个十分实用的文件管理器_linux mc
- 9iframe嵌套其它网站页面及相关知识点详解_iframe跨域问题 嵌入别人的网站
- 10SQL SELECT完整语法_sql select语法
R语言学习 - 非参数法生存分析_r 利用寿命表法绘制生存曲线
赞
踩
微信公众号:http://blog.genesino.com
生存分析指根据试验或调查得到的数据对生物或人的生存时间进行分析和推断,研究生存时间和结局与众多影响因素间关系及其程度大小的方法,也称生存率分析或存活率分析。常用于肿瘤等疾病的标志物筛选、疗效及预后的考核。
简单地说,比较两组或多组人群随着时间的延续,存活个体的比例变化趋势。活着的个体越少的组危险性越大,对应的基因对疾病影响越大,对应的药物治疗效果越差。
生存分析适合于处理时间-事件数据,如下
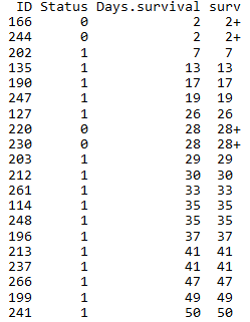
生存时间数据有两种类型:
- 完全数据 (complete data)指被观测对象从观察起点到出现终点事件所经历的时间; 一般用状态值1或TRUE表示。
- 截尾数据 (consored data)或删失数据,指在出现终点事件前,被观测对象的观测过程终止了。由于被观测对象所提供的信息是不完全的,只知道他们的生存事件超过了截尾时间。截尾主要由于失访、退出和终止产生。一般用状态值0或FALSE表示。
- TCGA中的临床数据标记也符合这个规律,在下面软件运行时也可修改状态值的含义, 但一般遵循这个规律。
生存概率 (survival probability)指某段时间开始时存活的个体至该时间结束时仍然存活的可能性大小。
生存概率=某人群活过某段时间例数/该人群同时间段期初观察例数。
生存率 (Survival rate),用S(t)表示,指经历t个单位时间后仍存活的概率,若无删失数据,则为活过了t时刻仍然存活的例数/观察开始的总例数。如果有删失数据,分母则需要按时段进行校正。
生存分析一个常用的方法是寿命表法。
寿命表是描述一段时间内生存状况、终点事件和生存概率的表格,需计算累积生存概率即每一步生存概率的乘积 (也可能是原始生存概率),可完成对病例随访资料在任意指定时点的生存状况评价。
R做生存分析
R中做生存分析需要用到包survival和survminer。输入数据至少两列,存活时间和生存状态,也就是测试数据中的Days.survial和vital_status列。如果需要比较不同组之间的差异,也需要提供个体的分组信息,如测试数据中的PAM50列。对应TCGA的数据,一般根据某个基因的表达量或突变有无对个体进行分组。
读入数据
library(survival)
BRCA <- read.table('BRCA.tsv', sep="\t", header=T)
head(BRCA)- 1
- 2
- 3
ID SampleType PAM50Call_RNAseq Days.survival pathologic_stage
1 TCGA-E9-A2JT-01 Tumor_type LumA 288 stage iia
2 TCGA-BH-A0W4-01 Tumor_type LumA 759 stage iia
3 TCGA-BH-A0B5-01 Tumor_type LumA 2136 stage iiia
4 TCGA-AC-A3TM-01 Tumor_type Unknown 762 stage iiia
5 TCGA-E9-A5FL-01 Tumor_type Unknown 24 stage iib
6 TCGA-AC-A3TN-01 Tumor_type Unknown 456 stage iib
vital_status
1 0
2 0
3 0
4 0
5 0
6 0- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
简单地看下每一列都有什么内容,方便对数据整体有个了解,比如有无特殊值。
summary(BRCA)- 1
ID SampleType PAM50Call_RNAseq Days.survival
TCGA-3C-AAAU-01: 1 Tumor_type:1090 Basal :138 Min. : 0.0
TCGA-3C-AALI-01: 1 Her2 : 65 1st Qu.: 450.2
TCGA-3C-AALJ-01: 1 LumA :415 Median : 848.0
TCGA-3C-AALK-01: 1 LumB :194 Mean :1247.0
TCGA-4H-AAAK-01: 1 Normal : 24 3rd Qu.:1682.8
TCGA-5L-AAT0-01: 1 Unknown:254 Max. :8605.0
(Other) :1084
pathologic_stage vital_status
stage iia :359 Min. :0.0000
stage iib :259 1st Qu.:0.0000
stage iiia:156 Median :0.0000
stage i : 90 Mean :0.1394
stage ia : 85 3rd Qu.:0.0000
stage iiic: 67 Max. :1.0000
(Other) : 74 
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
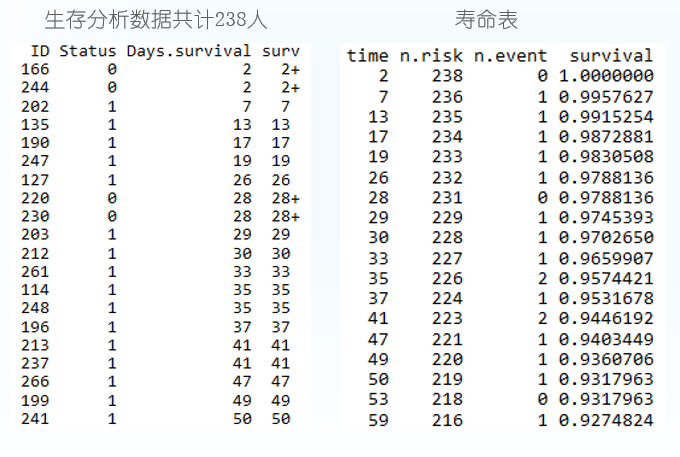
计算寿命表
# Days.survival:跟踪到的存活时间
# vital_status: 跟踪到的存活状态
# ~1表示不进行分组
fit <- survfit(Surv(Days.survival, vital_status)~1, data=BRCA)
# 获得的survial列就是生存率
summary(fit)
Call: survfit(formula = Surv(Days.survival, vital_status) ~ 1, data = BRCA)
time n.risk n.event survival std.err lower 95% CI upper 95% CI
116 1021 1 0.999 0.000979 0.997 1.000
158 1017 1 0.998 0.001386 0.995 1.000
160 1016 1 0.997 0.001697 0.994 1.000
172 1010 1 0.996 0.001962 0.992 1.000
174 1008 1 0.995 0.002195 0.991 0.999
197 1003 1 0.994 0.002406 0.989 0.999
224 993 1 0.993 0.002604 0.988 0.998
227 990 1 0.992 0.002788 0.987 0.998
239 987 1 0.991 0.002961 0.985 0.997
255 981 1 0.990 0.003125 0.984 0.996
266 978 1 0.989 0.003282 0.983 0.996
295 965 1 0.988 0.003435 0.981 0.995
302 962 1 0.987 0.003581 0.980 0.994
304 958 1 0.986 0.003723 0.979 0.993
320 948 1 0.985 0.003862 0.977 0.993
322 946 1 0.984 0.003995 0.976 0.992
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
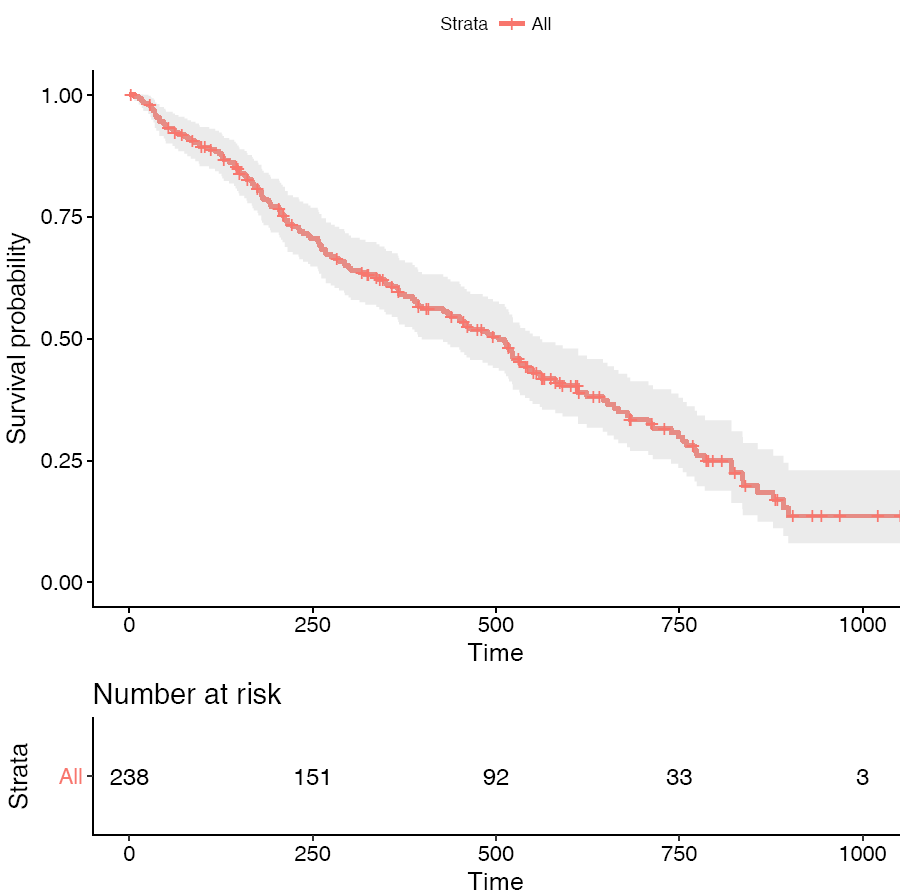
绘制生存曲线,横轴表示生存时间,纵轴表示生存概率,为一条梯形下降的曲线。下降幅度越大,表示生存率越低或生存时间越短。
library(survminer)
# conf.int:是否显示置信区间
# risk.table: 对应时间存活个体总结表格
ggsurvplot(fit, conf.int=T,risk.table=T)- 1
- 2
- 3
- 4
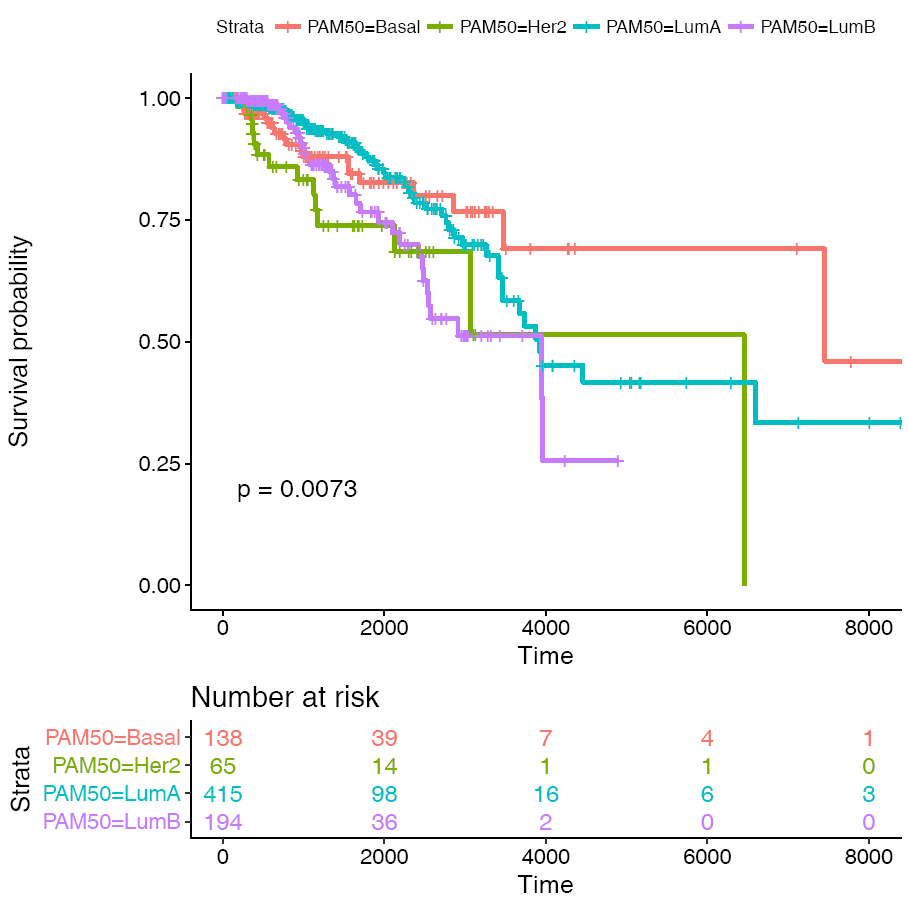
PAM50是通过50个基因的表达量把乳腺癌分为四种类型 (Luminal A, Luminal B, HER2-enriched, and Basal-like)作为预后的标志。根据PAM50属性对病人进行分组,评估比较两组之间生存率的差别。
# 这三步不是必须的,只是为了方便,选择其中的4个确定了的分组进行分析
# 同时为了简化图例,给列重命名一下,使得列名不那么长
BRCA_PAM50 <- BRCA[grepl("Basal|Her2|LumA|LumB",BRCA$PAM50Call_RNAseq),]
BRCA_PAM50 <- droplevels(BRCA_PAM50)
colnames(BRCA_PAM50)[colnames(BRCA_PAM50)=="PAM50Call_RNAseq"] <- 'PAM50'
# 按PAM50分组
fit <- survfit(Surv(Days.survival, vital_status)~PAM50, data=BRCA_PAM50)
# 绘制曲线
ggsurvplot(fit, conf.int=F,risk.table=T, risk.table.col="strata", pval=T)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
简化Stage信息,先只查看大的阶段
BRCA_PAM50$pathologic_stage <- gsub('(i+v*).*', "\\1", BRCA_PAM50$pathologic_stage)
BRCA_PAM50$pathologic_stage <- as.factor(BRCA_PAM50$pathologic_stage)
colnames(BRCA_PAM50)[colnames(BRCA_PAM50)=="pathologic_stage"] <- 'PS'
fit <- survfit(Surv(Days.survival, vital_status)~PS, data=BRCA_PAM50)
# 绘制曲线
ggsurvplot(fit, conf.int=F,risk.table=T, risk.table.col="strata", pval=T)- 1
- 2
- 3
- 4
- 5
- 6


