- 1Blender在Ubuntu上的编译
- 2fluentd 使用笔记_concat fluentd 时间开头
- 3Spark--Spark编程基础和编程进阶知识总结(第三章和第四章)
- 4three.js 交互_如何使用Three.js创建交互式3D角色
- 5Flink 流处理流程 API详解_flink流api
- 610G_Ethernet_02 10G Ethernet Subsystem 简介
- 7信息学奥赛一本通 1080:余数相同问题_1080:余数相同问题
- 8linux虚拟机搭建kafka(单节点、使用kafka自带zookeeper)_linux安装单节点kafka
- 9华为OD机试真题-游戏分组-2023年OD统一考试(C卷)_王者荣耀游戏分组绝对值
- 10idea链接不上虚拟机的docker里的mongodb_虚拟机内的mongodb访问不了
医学YOLOv8 | 脑肿瘤检测实战
赞
踩
在医疗保健领域,准确和高效地识别脑肿瘤是一个重大挑战。本文中,我们将探讨一种使用 YOLOv8,一种先进的目标检测模型,将脑肿瘤进行分类的新方法,其准确率达到了 99%。通过将深度学习与医学图像相结合,我们希望这种方法将提高脑肿瘤识别的速度和准确性。
首先,我们将从 Kaggle 获取脑肿瘤分类数据集。然后,我们将利用各种数据清理方法来准备数据,以输入到我们的模型中。接下来,我们将从 Ultralytics 下载 YOLOv8,并对其进行调整以适应我们的具体情况。最后,我们将创建一个 FastAPI 应用程序以实现简化的使用。
1. 探索数据集
让我们仔细看看刚刚从 Kaggle 下载的数据集。数据集已经被分为训练集和测试集。数据集中有四种不同的肿瘤类别:胶质瘤、脑膜瘤、垂体瘤和无肿瘤。每个类别在训练集中约有 1300 个示例,在测试集中约有 300 个示例。
2. 数据准备
在此过程中,我们将利用各种技术来清理数据集图像。这些技术包括识别和删除损坏的图像,以及消除尺寸明显较小的图像。
2.1 删除损坏的图像
在这个过程中,我们必须检查每个类别中的所有图像,并验证它们是否可以使用 cv.imread 打开。如果无法打开,就必须从数据集中删除它,因为它可能是损坏的图像或根本不是图像文件。
2.2 删除尺寸不合格的图像
另一个数据清理技术涉及检查图像是否低于某个尺寸阈值并将其删除。这很重要,因为冗余数据可能会对我们的模型性能产生负面影响。下面的代码可以一次删除所有损坏的和低于阈值的图像。
- import os
- import cv2
-
-
- train_dir = "BrainTumor/train"
-
-
- categories = ["glioma", "meningioma", "notumor", "pituitary"]
-
-
- size_threshold = (10,10)
- valid_extensions=('.jpg', '.png', '.jpeg')
-
-
- def is_image_corrupt(image_path):
- try:
- img = cv2.imread(image_path)
- if img is None:
- return True
- return False
- except:
- return True
-
-
-
-
-
- def is_image_below_threshold(img_path):
- img = cv2.imread(image_path)
- if img.shape <= size_threshold:
- print(img.shape)
- return True
- return False
-
-
-
- for each_category in categories:
- folder_path = os.path.join(train_dir, each_category)
- for each_file in os.listdir(folder_path):
- image_path = os.path.join(folder_path, each_file)
- if os.path.isfile(image_path) and each_file.lower().endswith(valid_extensions):
- if is_image_corrupt(image_path) or is_image_below_threshold(image_path):
- os.remove(image_path)
- print(f"Removed corrupt image: {each_file}")

3. 数据分析
作为分析的一部分,我们将检查数据集以确定总记录数和每个类别的图像数量。我们还将评估类别的分布,并生成图表以增进对数据的理解。
这种方法允许我们从数据中获得洞察,以防止在将其输入模型时出现过拟合和欠拟合问题。
- import matplotlib.pyplot as plt
- import os
-
-
- train_dir = "/BrainTumor/train"
- valid_extensions=('.jpg', '.png', '.jpeg')
-
-
- categories = ["glioma", "meningioma", "notumor", "pituitary"]
- category_count = {}
-
-
- for each_category in categories:
- folder_path = os.path.join(train_dir, each_category)
- valid_images = [file for file in os.listdir(folder_path) if file.lower().endswith(valid_extensions)]
- category_count[each_category] = len(valid_images)
-
-
- fig, ax = plt.subplots(figsize=(10, 4))
-
-
- # Bar chart
- bar_plot = plt.barh(list(category_count.keys()), list(category_count.values()), 0.5)
- plt.title('Tumor Type Distribution')
- plt.xlabel('Count')
- plt.ylabel('Tumor Type')
- for i, bar in enumerate(bar_plot):
- plt.text(bar.get_width(), bar.get_y() + bar.get_height() / 2, str(list(category_count.values())[i]), ha='left', va='center')
-
-
- plt.show()
-
-
-
-
-
-
- sample_size = sum(category_count.values())
-
-
- class_dist = {key : val/sample_size for key, val in category_count.items()}
-
-
-
-
- fig, ax = plt.subplots(figsize=(10, 4))
-
-
-
-
- # Bar chart
- bar_plot = plt.barh(list(class_dist.keys()), list(class_dist.values()), 0.6)
- plt.title('Class Distribution')
- plt.xlabel('Class')
- plt.ylabel('Percentage')
-
-
- for i, bar in enumerate(bar_plot):
- plt.text(bar.get_width(), bar.get_y() + bar.get_height() / 2, str(round(list(class_dist.values())[i], 3)), ha='left', va='center')
-
-
- plt.show()

所展示的代码将创建两个条形图,表示每个类别的图像数量和类别分布。图表显示我们的数据分布均匀,虽然在“无肿瘤”类中的图像数量稍多,但与其他类别相比仍然相对平衡。
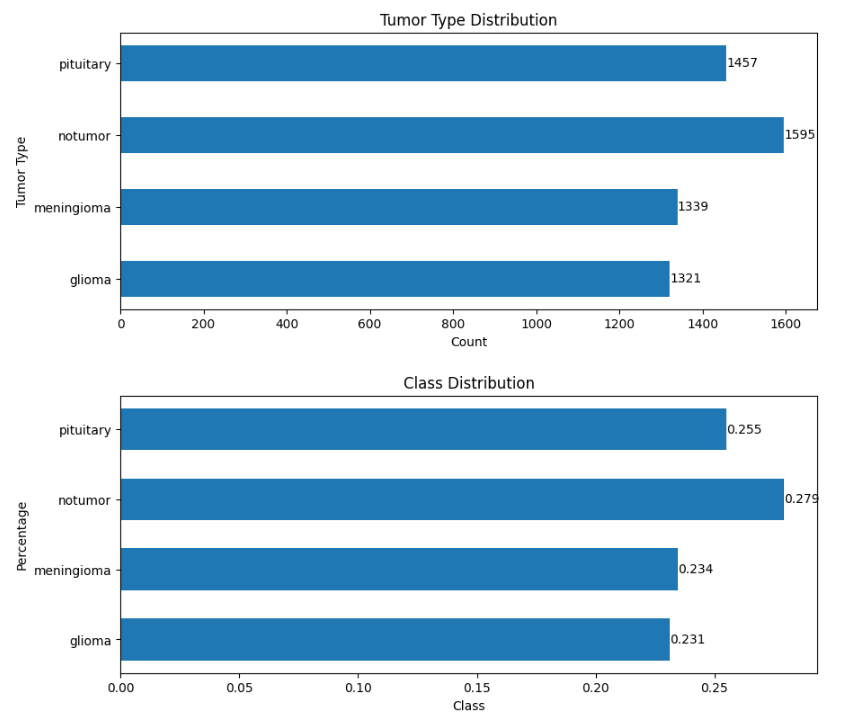
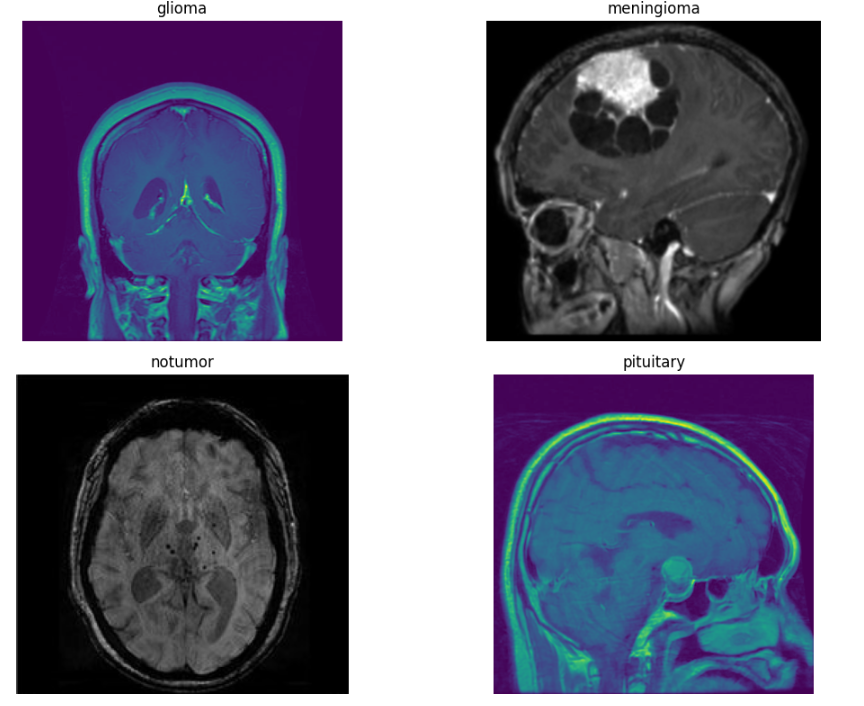
4. 数据可视化
在将数据输入之前,用肉眼查看它对于更好的理解是很重要的。下面提供的代码显示了每个类别的一张图像。
- import matplotlib.pyplot as plt
- import os
- train_dir = "/BrainTumor/train"
- valid_extensions=('.jpg', '.png', '.jpeg')
-
-
- categories = ["glioma", "meningioma", "notumor", "pituitary"]
-
-
- plt.figure(figsize=(12, 8))
- for i, category in enumerate(categories):
- folder_path = os.path.join(train_dir, category)
- image_path = os.path.join(folder_path, os.listdir(folder_path)[0])
- if not image_path.lower().endswith(valid_extensions):
- continue
- img = plt.imread(image_path)
- plt.subplot(2, 2, i+1)
- plt.imshow(img)
- plt.title(category)
- plt.axis("off")
- plt.tight_layout()
- plt.show()

输出:
5. 训练模型
在脑肿瘤分类项目中,我们将使用 YOLOv8 预训练模型。我们的第一步将是将这个模型导入项目中。接下来,我们将用我们的数据集微调预训练模型。最后,我们将在测试数据上评估模型,以确定每个类别的准确性。
- from ultralytics import YOLO
-
-
- model = YOLO('yolov8m-cls.pt') # load a pretrained YOLOv8n classification model
-
-
- # train/pre-tuned the model on our dataset
- model.train(data='BrainTumor', epochs=3)
-
-
- # run the model on test data
- res = model.val()
-
-
- # Result saved to runs/classify/val
要获得归一化混淆矩阵,导航到当前目录中的 runs/classify/val 文件夹。一旦进入那里,您将能够以以下图像的形式查看它。
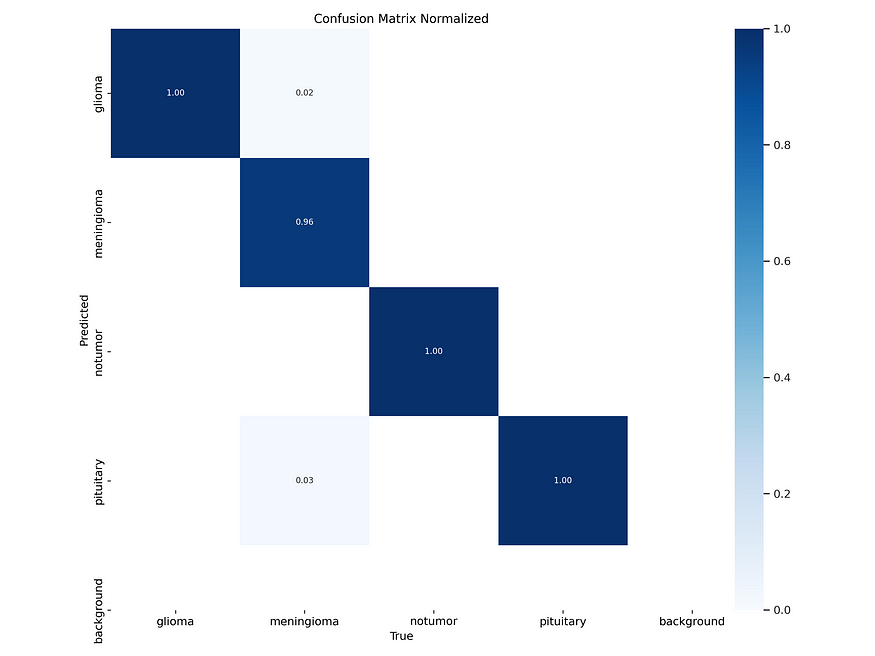
从所提供的数据中,模型在三个类别上达到了 100% 的性能,而在一个类别(脑膜瘤)上达到了 96%。因此,总准确性可以计算如下:(100 x 3 + 96) / 4 = 99%。
6. 测试自定义图像
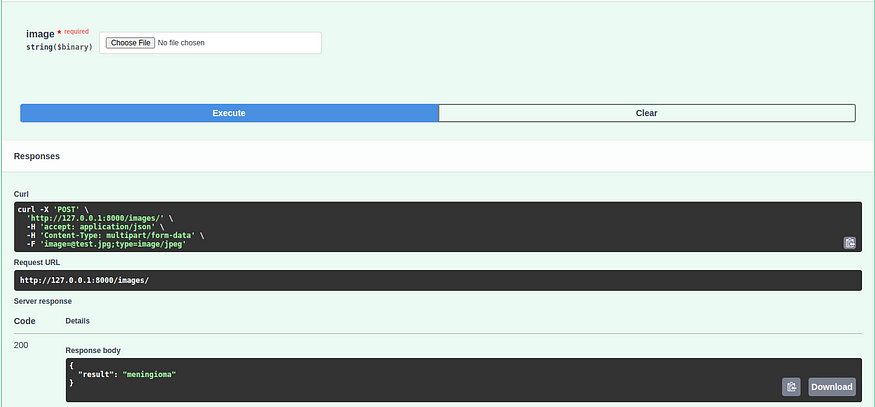
在项目的最后一步,我们将在 FastAPI 中建立一个端点。这个端点将接受图像作为输入,并返回图像的标签预测。有了这个功能,我们可以轻松地在任何选择的图像上测试我们的模型。在微调模型后,它将生成另一个预训练模型文件(.pt),位于/run/classify/train/weights/best.pt 中。我们将将此文件集成到我们的 FastAPI 项目中。
以下是运行在端口 8000 上的 FastAPI 代码,它有一个 /images 的端点。这个端点将以图像作为输入,并返回由我们的模型(best.pt)预测的图像标签。
- import subprocess
- from fastapi import FastAPI, UploadFile, File
- from ultralytics import YOLO
-
-
-
-
- def model_train():
- model = YOLO('./runs/classify/train/weights/best.pt') # load a pretrained YOLOv8n classification model
- return model
-
-
-
-
- app = FastAPI()
-
-
- model_data = None
-
-
-
-
- @app.post("/images/")
- def create_upload_file(image: UploadFile = File(...)):
- global model_data
- if model_data is None:
- model_data = model_train()
-
-
- with open(f"./images/{image.filename}", "wb+") as f:
- f.write(image.file.read())
-
-
- result = model_data(f"./images/{image.filename}")
-
-
- return {"result": result[0].names[result[0].probs.top1]}
-
-
-
-
- def run_uvicon():
- uvicorn_command = [
- "uvicorn",
- "main:app",
- "--host", "127.0.0.1",
- "--port", "8000",
- "--reload",
- ]
-
-
- subprocess.run(uvicorn_command, check=True)
-
-
-
-
- if __name__ == "__main__":
- run_uvicon()

输出:
结论
总之,我们文章中使用的数据是从 Kaggle 平台获取的。在此之后,我们对数据进行了清理处理,然后将其输入到模型中,最终生成了归一化混淆矩阵。作为最后一步,我们使用 FastAPI 建立了一个端点,使我们能够以高度准确和高效的方式进行预测,并随后返回输入系统的任何肿瘤图像的类别。
· END ·
HAPPY LIFE

本文仅供学习交流使用,如有侵权请联系作者删除


