- 1Java面试题总结(四)-mysql篇、redis篇
- 2五种开源协议的比较(BSD、Apache、GPL、LGPL、MIT)_开源协议对比
- 3go 解压和压缩包
- 4交换机中对于tagged帧和untagged帧的处理
- 5双目测距python_python+openCV实现双目视差图及测距
- 606_Python游戏编程(Pygame)_制作一 个跳跃的热气球游戏 创建一个游戏窗口,然后在窗口内创建一个热 气球。
- 7华为云幻兽帕鲁服务器搭建教程(windows平台)_帕鲁winserver服务器容器架设
- 8C语言 fclose 函数 - C语言零基础入门教程_fclose()
- 9Mysql连接超时的问题与解决方法_failoverreadonly
- 10Android 逆向调用so(一)_android 逆向 so
【Python VTK】读取二维序列医学图像分割结果并进行三维重建_vtk三维重建
赞
踩
一、问题描述
最近在开发过程中遇到了这样的问题:
在医学图像开发过程中,我们将医学图像通过深度学习算法进行分割,现在想要通过这一套二维图像进行三维重构。
以下是分割结果:
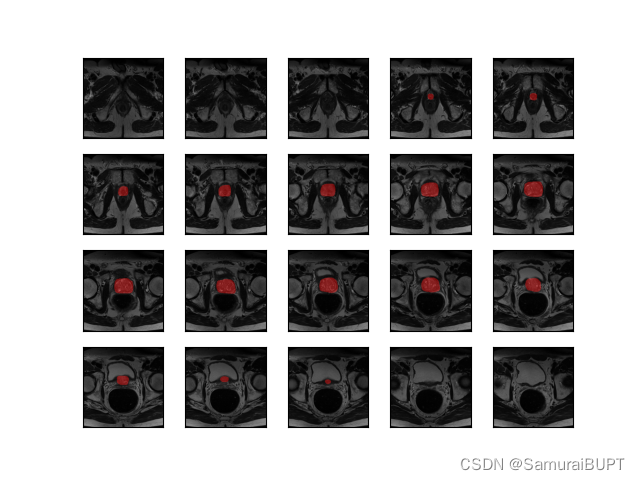
图一:前列腺核磁图像分割结果 图一:前列腺核磁图像分割结果 图一:前列腺核磁图像分割结果
以下是读取的遮罩mask:

图二:图像分割遮罩
图二:图像分割遮罩
图二:图像分割遮罩
如何将这些二维图像进行三维重建,是个棘手问题,笔者通过vtk进行建模操作。
二、解决方案
0. 写在前面
医学图像的三维重建本身就是热点技术,这项技术也并非新鲜技术,笔者调研多份前者的博客与其余资料,整理出了自己的解决方案,旨在与大家共同交流,如果您有更好的建模方案,欢迎随时与我交流!
1. 准备工作
进行医学图像的三维重建,首先需要提供清晰可见的轮廓与遮罩。(如图二所示)
所用到的库:
- vtk,您可以通过
pip install vtk直接安装
2. 文件结构
- mask文件夹 (用于存放分割结果遮罩,图片名为
mask_0.png,mask_1.png,mask_2.png等20张图片) - vtk_gaussian.py (python脚本,用于执行并进行三维重建)
如图所示:
图三:项目采用的文件结构 图三:项目采用的文件结构 图三:项目采用的文件结构
3. 代码讲解
3.0 完整代码
我知道有的朋友比较急,这里先给出完整代码:
import vtk # 定义渲染窗口、交互模式 aRender = vtk.vtkRenderer() Renwin = vtk.vtkRenderWindow() Renwin.AddRenderer(aRender) iren = vtk.vtkRenderWindowInteractor() iren.SetRenderWindow(Renwin) # 定义个图片读取接口 # 读取PNG图片就换成PNG_Reader = vtk.vtkPNGReader() PNG_Reader = vtk.vtkPNGReader() PNG_Reader.SetNumberOfScalarComponents(1) PNG_Reader.SetFileDimensionality(2) # 说明图像是三维的 # 定义图像大小,本行表示图像大小为(512*512*240) PNG_Reader.SetDataExtent(0, 256, 0, 256, 0, 19) # 设置图像的存放位置 name_prefix = ['mask/mask_'] PNG_Reader.SetFilePrefix(name_prefix[0]) # 设置图像前缀名字 # 表示图像前缀为数字(如:0.jpg) PNG_Reader.SetFilePattern("%s%d.png") PNG_Reader.Update() PNG_Reader.SetDataByteOrderToLittleEndian() spacing = [1.0, 1.0, 2.5] # x, y 方向上的间距为 2 像素,z 方向上的间距为 2.5 像素 PNG_Reader.GetOutput().SetSpacing(spacing) # 高斯平滑 gauss = vtk.vtkImageGaussianSmooth() gauss.SetInputConnection(PNG_Reader.GetOutputPort()) gauss.SetStandardDeviations(1.0, 1.0, 1.0) gauss.SetRadiusFactors(1.0, 1.0, 1.0) gauss.Update() # 计算轮廓的方法 contour = vtk.vtkMarchingCubes() gauss.GetOutput().SetSpacing(spacing) contour.SetInputConnection(gauss.GetOutputPort()) contour.ComputeNormalsOn() contour.SetValue(0, 100) mapper = vtk.vtkPolyDataMapper() mapper.SetInputConnection(contour.GetOutputPort()) mapper.ScalarVisibilityOff() actor = vtk.vtkActor() actor.SetMapper(mapper) renderer = vtk.vtkRenderer() renderer.SetBackground([1.0, 1.0, 1.0]) renderer.AddActor(actor) window = vtk.vtkRenderWindow() window.SetSize(512, 512) window.AddRenderer(renderer) interactor = vtk.vtkRenderWindowInteractor() interactor.SetRenderWindow(window) # 开始显示 if __name__ == '__main__': window.Render() interactor.Initialize() interactor.Start()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
- 54
- 55
- 56
- 57
- 58
- 59
- 60
- 61
- 62
- 63
- 64
- 65
- 66
3.1 定义渲染窗口、交互模式
import vtk
# 定义渲染窗口、交互模式
aRender = vtk.vtkRenderer()
Renwin = vtk.vtkRenderWindow()
Renwin.AddRenderer(aRender)
iren = vtk.vtkRenderWindowInteractor()
iren.SetRenderWindow(Renwin)
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
本人的所有三维重建脚本中几乎都包含这一块内容,同时这也是进行vtk交互窗口初始化的部分,更多信息您可以查阅vtk官方文档或者其他技术博客。
3.2 读取二维图像序列 - 定义读取接口
要将mask_0.png到mask_19.png的图像全部读取,需要先定义个图片读取接口 (vtk.vtkXxxReader)。
笔者的图像为.png格式,因此使用vtk.vtkPNGReader() 进行图像读取。
PNG_Reader = vtk.vtkPNGReader()
PNG_Reader.SetNumberOfScalarComponents(1)
PNG_Reader.SetFileDimensionality(2) # 说明图像是二维的
- 1
- 2
- 3
在这里,可以根据不同的图片格式选取不同的vtkReader,如果是 .jpg 格式的图像,可以有如下更改:
JPG_Reader = vtk.vtkJPEGReader()
# your code here...
- 1
- 2
3.3 读取二维图像序列 - 前置设置 & 图像读取
# 定义图像大小,本人表示图像大小为(256*256)
# 后两个参数是图片的数目,本人这里所用的图像共20张,所以就输入0, 19
# 在后续读取的时候,会根据这个序列进行读取
PNG_Reader.SetDataExtent(0, 256, 0, 256, 0, 19)
# 设置图像的存放位置
name_prefix = ['mask/mask_']
PNG_Reader.SetFilePrefix(name_prefix[0])
# 表示图像前缀为数字(如:0.jpg)
PNG_Reader.SetFilePattern("%s%d.png")
PNG_Reader.Update()
PNG_Reader.SetDataByteOrderToLittleEndian()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
这段代码是进行图像读取的一些前置设置。SetDataExtent()函数的参数设置会对后续图像的处理会有一定影响,请正确填写您所使用的图片的大小和数目!
SetFilePrefix() 函数会根据传入的字符串进行锁定。在这里一定要特别注意:
- 本人的图像存放在
mask文件夹下,每张图片的名字为:mask_0.png,mask_1.png等 - 在这里设置Prefix时,就要输入
mask/mask_ - 后面的
SetFilePattern()函数会自动读取数字,因为前缀已经设置好,不需要再在此处进行一些正则运算符操作
3.4 图像数据量少,三维建模的结果很扁平
解决方案:您可以增大图像之间的间距来解决这个问题,紧接着上面的代码:
PNG_Reader.SetDataByteOrderToLittleEndian()
spacing = [1.0, 1.0, 2.5] # x, y 方向上的间距为 2 像素,z 方向上的间距为 2.5 像素
PNG_Reader.GetOutput().SetSpacing(spacing)
- 1
- 2
- 3
在读取好图片之后,可以设置图像之间的 spacing 这个列表,分别代表x, y, z 三个维度的间距,其中我将z维度的间距增大为2.5, 这样的操作在后面的建模中有显著效果。具体内容如下:


图四:两种不同间距的建模结果,左图为 z = 1.0 ,右图为 z = 2.5 图四:两种不同间距的建模结果,左图为z=1.0,右图为z=2.5 图四:两种不同间距的建模结果,左图为z=1.0,右图为z=2.5
为了使建模结果更接近与器官的形状,我建议设置好二维图像之间的间距。
3.5 三维重建结果的平滑—高斯平滑
原本建模的结果“层次分明”,并不是特别美观。笔者采用高斯平滑的方案对图像进行平滑。
如果您有更好的平滑方案,欢迎您与我交流!
# 高斯平滑
gauss = vtk.vtkImageGaussianSmooth()
gauss.SetInputConnection(PNG_Reader.GetOutputPort())
gauss.SetStandardDeviations(1.0, 1.0, 1.0)
gauss.SetRadiusFactors(1.0, 1.0, 1.0)
gauss.Update()
- 1
- 2
- 3
- 4
- 5
- 6


图五:两种不同间距的平滑结果,左图为未平滑,右图为使用高斯平滑 图五:两种不同间距的平滑结果,左图为未平滑,右图为使用高斯平滑 图五:两种不同间距的平滑结果,左图为未平滑,右图为使用高斯平滑
3.6 计算轮廓与边缘提取
在进行高斯平滑之后,进行边缘提取。
# 计算轮廓的方法
contour = vtk.vtkMarchingCubes()
gauss.GetOutput().SetSpacing(spacing)
contour.SetInputConnection(gauss.GetOutputPort())
contour.ComputeNormalsOn()
contour.SetValue(0, 100)
- 1
- 2
- 3
- 4
- 5
- 6
3.7 管道操作与可视化展示
后面这段代码是vtk的显示部分,笔者一般不去动它,也是每一份脚本中的固有内容,如果您对该部分感兴趣,您应该查阅vtk官方文档。
mapper = vtk.vtkPolyDataMapper() mapper.SetInputConnection(contour.GetOutputPort()) mapper.ScalarVisibilityOff() actor = vtk.vtkActor() actor.SetMapper(mapper) renderer = vtk.vtkRenderer() renderer.SetBackground([1.0, 1.0, 1.0]) renderer.AddActor(actor) window = vtk.vtkRenderWindow() window.SetSize(512, 512) window.AddRenderer(renderer) interactor = vtk.vtkRenderWindowInteractor() interactor.SetRenderWindow(window) # 开始显示 if __name__ == '__main__': window.Render() interactor.Initialize() interactor.Start()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
三、一些vtk库的使用经验分享
1. python vtk
个人感觉python vtk的开发并没有pythonic风格,开发者有一些将cpp的开发思路带入python库/接口的设计,让我写起来味如嚼蜡,从上面的代码亦可以看出,具有浓厚的cpp风格。
不过代码能跑就行,不得不说vtk仍然是很强大的三维重建工具!
2. 数据传入的两种方式
2.1 .GetOutputPort() 和 .SetInputConnection()
您会看见诸如 contour.SetInputConnection(gauss.GetOutputPort()) 的句子。
这两个函数一般是成对出现,上下传递的。
2.1 .GetOutput() 和 .SetInputData()
笔者在参阅其他博主的博客时,同样看见这样的写法,例如:
contour.SetInputData(gauss.GetOutput())
这两个函数一般是成对出现的,进行上下传递处理结果。
3. vtkImageData 和 vtkPolyData
在开发过程中,您可能会遇到不少报错,其中肯定会有vtk数据类型报错的问题。我整理了一份表格:
| Name | Input Type | Return Type | Variable |
|---|---|---|---|
| vtk.vtkPNGReader() | ? | vtkImageData | PNG_Reader |
| vtk.vtkImageGaussianSmooth() | vtkImageData | vtkImageData | gauss |
| vtk.vtkMarchingCubes() | vtkImageData | vtkPolyData | contour |
| vtk.vtkPolyDataNormals() | vtkImageData | vtkPolyData | normfilter |
希望能够帮助您解决开发过程中的一些疑惑。
后记
医学图像的三维重建工作部分博客较少,笔者希望提供一些星星之火,大家共同进步!
笔者采用的重建代码已经打包至百度云盘,您可以通过下面的链接下载:
提取码:jpt3



