- 1目标识别项目实战:基于Yolov7-LPRNet的动态车牌目标识别算法模型(二)_基于lprnet车牌识别算法
- 2Git实用教程 4.0:回到过去_git reset: moving to head
- 3docker安装步骤_docker 安装
- 4大模型日报2024-04-14
- 5明月之刃:armbian巧借nmtui管理网络连接_armbian wifi
- 6取证工具volatility插件版学习记录_volatility 取证
- 7频谱、能谱、功率谱、倍频程谱、1/3 倍频程谱_1/3倍频程振动加速度级
- 8linux v3h平台开发系列详解(GMSL摄像头篇):1.3 两路GMSL相机_gmsl多通道摄像头
- 9linux抓包tcpdump并保存,linux下抓包命令--tcpdump的使用
- 10Unreal Engine 常用组件介绍
SCS【2】单细胞转录组 之 cellranger
赞
踩

点击关注,桓峰基因
前言
单细胞RNA-seq使基因表达的研究达到了前所未有的高度。这项技术的前景正在吸引越来越多的用户使用单细胞分析方法。随着越来越多的分析工具可用,导航这一景观和产生一个最新的工作流来分析一个人的数据变得越来越困难。在这里,我们详细介绍了一个典型的scRNA-seq分析的步骤,包括预处理(质量控制、归一化、数据校正、特征选择和降维)和细胞和基因水平的下游分析。我们基于独立的比较研究制定了这些步骤的当前最佳实践建议。我们已经将这些最佳实践建议集成到一个工作流中,并将其应用于一个公共数据集,以进一步说明这些步骤在实践中如何工作。
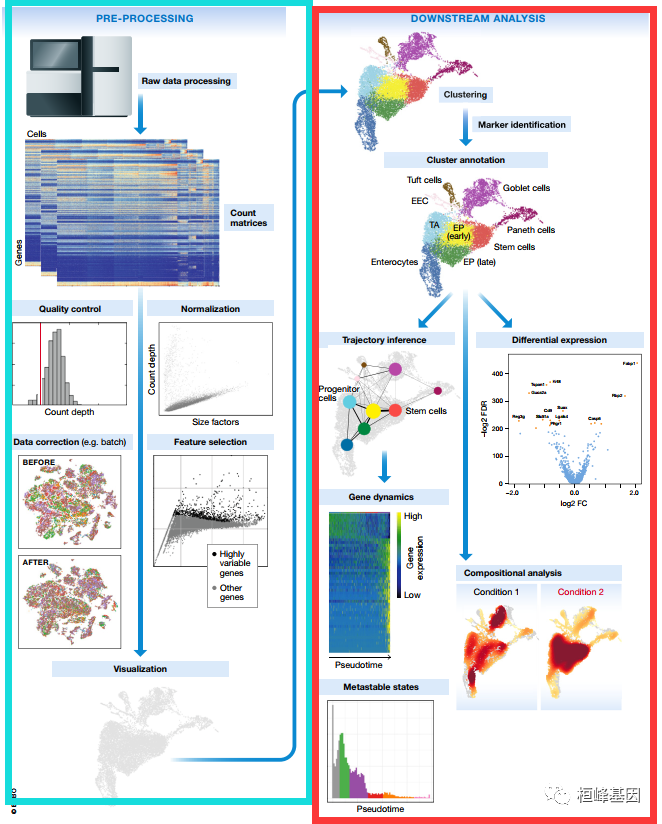
What is Cell Ranger?
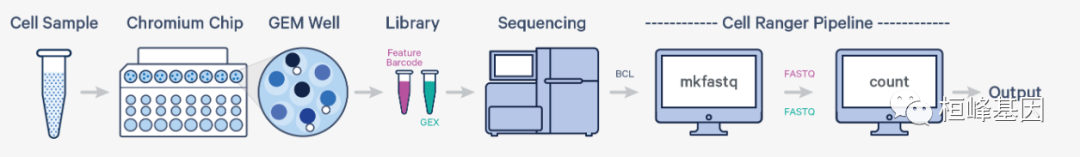
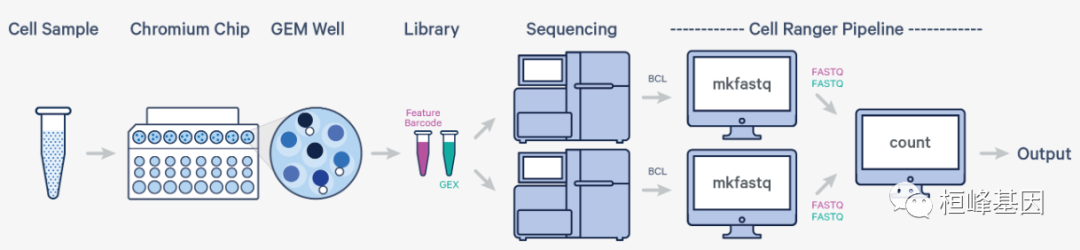
Cell Ranger是10X genomics公司为单细胞RNA测序分析量身打造的数据分析软件,可以直接输入Illumina 原始数据(raw base call,BCL)输出表达定量矩阵、降维(pca),聚类(Graph-based& K-Means)以及可视化(t-SNE)结果,结合配套的Loupe Cell Browser给予研究者更多探索单细胞数据的机会。cellranger的高度集成化,使得单细胞测序数据探索变得更加简单,研究者有更多的时间来做生物学意义的挖掘。Cell Ranger包括5个与3’单细胞基因表达解决方案和相关产品的分析流程:
1. cellranger mkfastq
将Illumina测序仪生成的原始基调用(BCL)文件分解为FASTQ文件。它是Illumina的bcl2fastq的包装,具有10x Genomics库特有的附加功能和简化的样本表格式。
2. cellranger count
cellranger count从cellranger mkfastq中获取FASTQ文件,并执行对齐、过滤、条形码计数和UMI计数。它使用Chromium细胞条形码生成特征条形码矩阵,确定簇,并执行基因表达分析。计数管道可以从同一口GEM井的多次排序运行中获取输入。cellranger count还处理特征条形码数据和基因表达读取。
3. cellranger multi
cellranger multi用于分析Cell Multiplexing和Fixed RNA Profiling数据。它从cellranger mkfastq中获取FASTQ文件,并执行对齐、过滤、条形码计数和UMI计数。它使用Chromium细胞条形码生成特征条形码矩阵,确定簇,并执行基因表达分析。cellranger多管道还支持特征条码数据的分析。
4. cellranger aggr
Cellranger aggr聚合了多个Cellranger count或Cellranger multi运行的输出,将这些运行归一化到相同的测序深度,然后重新计算特征条形码矩阵并对组合数据进行分析。aggr管道可用于将来自多个样本的数据组合成一个实验范围内的特征条形码矩阵和分析。
5. cellranger reanalyze
Cellranger再分析采用由Cellranger计数、Cellranger或Cellranger聚合产生的特征条形码矩阵,并使用可调参数设置重新运行降维、聚类和基因表达算法。
工作流程
如果您从原始基本调用(BCL)文件开始,Cell Ranger工作流将从为每个流单元目录解码BCL文件开始。10x基因组公司建议使用生成FASTQs中描述的cellranger mkfastq。如果您从已经通过bcl2fastq直接解复用的FASTQ文件开始,或者从SRA等公共源文件开始,您可以跳过cellranger mkfastq,并从cellranger count开始。请参阅指定输入FASTQ页面(count, multi),以了解在您的场景中使用哪些参数的具体指导方针。工作流程的具体步骤取决于您有多少样本、GEM孔和流式细胞,以及您是否包括来自特征条形码、细胞多路复用或固定RNA分析试剂盒的数据。本节描述了一些可能的工作流程。
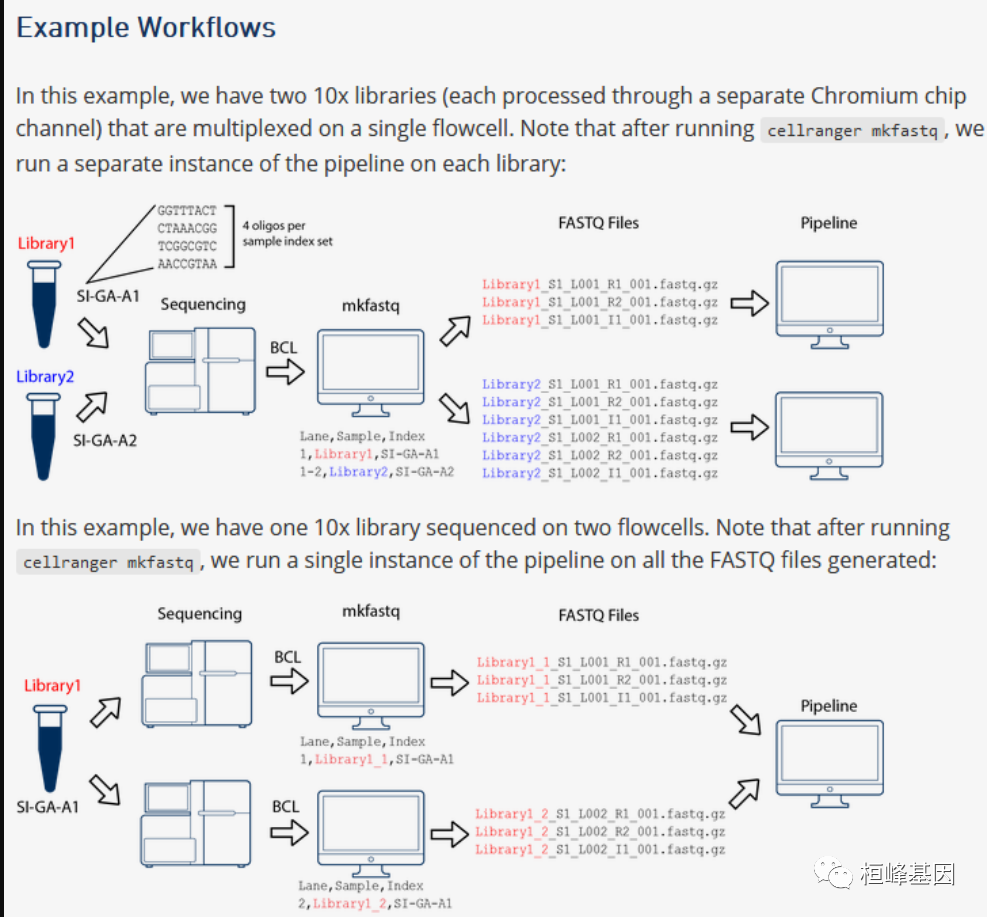
一个样品,一个GEM孔,一个流孔
在本例中,一个样品通过一个GEM阱处理,并在一个流单元上测序。在这种情况下,使用cellranger mkfastq生成FASTQs,并按照单样本分析中的描述运行cellranger count。这个例子还演示了两个测序库。一个GEM井可以产生多个物理库:一个基因表达库和一个或多个特征条码库。
一个样品,一个GEM孔,多个流动细胞
在本例中,一个样本通过一个GEM井处理,产生一个跨多个流单元测序的库。这种工作流程通常用于增加排序深度。在这种情况下,可以将所有读取合并到一个cellranger计数实例或多管道中。在指定Input FASTQ页面(count, multi)中描述了这个过程。
一个样品,多个GEM孔,一个流动槽
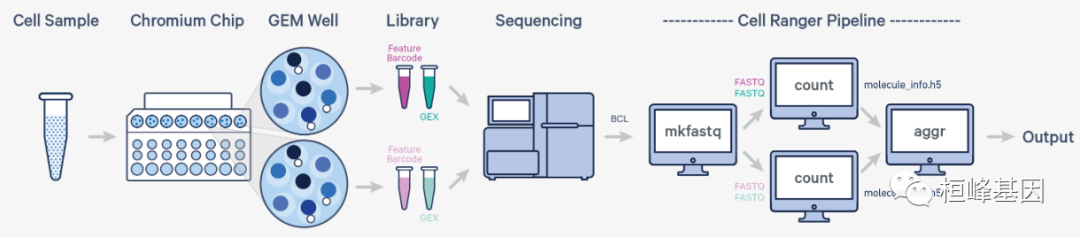
在这里,一个样品通过多个GEM孔处理。这通常是在进行技术复制实验时进行的。然后将来自GEM井的库汇集到一个流单元并进行测序。在这种情况下,使用cellranger mkfastq对测序运行的数据进行多路分离,然后通过单独的cellranger计数实例从每个GEM井运行库。然后,您可以使用cellranger aggr执行组合分析,如多库聚合中所述。
多个样品,多个GEM孔,一个流动槽
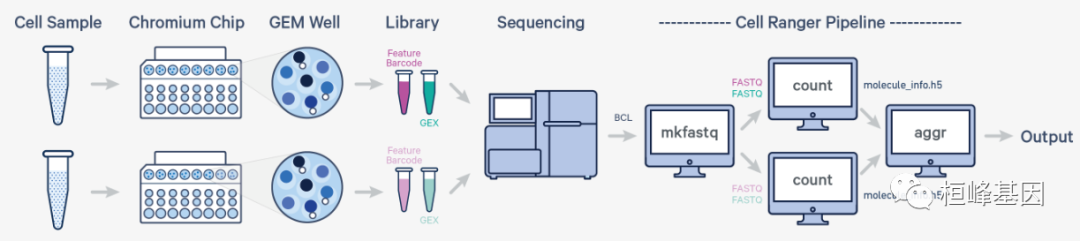
在本例中,多个样本通过多个GEM孔处理,这些GEM孔生成多个库,并被池放在一个流单元上。解复用后,必须对每个GEM井分别运行cellranger计数;如果你有两个GEM井,那么运行cellranger count两次。然后,您可以使用cellranger aggr的单个实例聚合它们,如多库聚合中所述。
多个样品,一个GEM孔,一个流孔(cell Multiplexing)
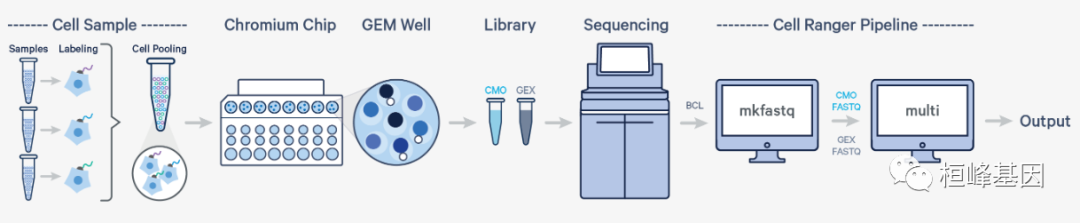
Cell Ranger 6.0引入了对分析Cell多路数据的支持。在这种情况下,多个样本使用Cell Multiplexing Oligos (CMOs)进行唯一标记,使多个样本可以集中在单个GEM孔中。这就产生了每个GEM孔的CMO和基因表达(GEX)库。运行cellranger mkfastq来生成FASTQ文件之后,在GEX和CMO库的组合FASTQ数据上运行cellranger多管道。
多个样本,一个GEM孔,一个流式细胞(固定RNA分析)
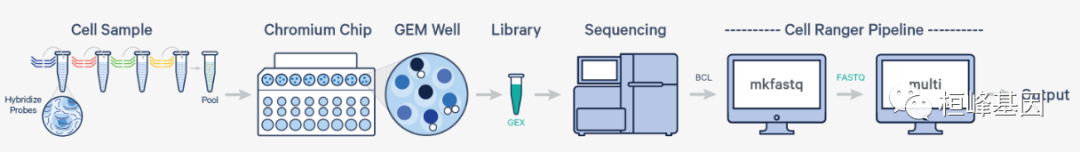
Cell Ranger 7.0引入了对分析固定RNA分析(FRP)基因表达数据的支持。在这种情况下,多个样本被唯一地标记为探针条形码,使样本能够集中在一个GEM孔中,并产生一个基因表达库。运行cellranger mkfastq来生成FASTQ文件之后,在GEX库的FASTQ数据上运行cellranger多管道。
软件安装
我们这里选择最新版Cell Ranger 7.0,每个版本都对应着参考数据以及例子数据,7.0版本的数据下载地址如下:
https://support.10xgenomics.com/single-cell-gene-expression/software/downloads/latest
在这个网页上下载软件以及参考基因组还有就是测试数据。
1. cellranger 7.0 下载
curl -o cellranger-7.0.0.tar.gz "https://cf.10xgenomics.com/releases/cell-exp/cellranger-7.0.0.tar.gz?Expires=1656187885&Policy=eyJTdGF0ZW1lbnQiOlt7IlJlc291cmNlIjoiaHR0cHM6Ly9jZi4xMHhnZW5vbWljcy5jb20vcmVsZWFzZXMvY2VsbC1leHAvY2VsbHJhbmdlci03LjAuMC50YXIuZ3oiLCJDb25kaXRpb24iOnsiRGF0ZUxlc3NUaGFuIjp7IkFXUzpFcG9jaFRpbWUiOjE2NTYxODc4ODV9fX1dfQ__&Signature=FEFSd-63q1ZuJwFOGD5KlH-OpL7Rzpj9UqBC7lOgRM8v4C1fJ9ndTwv8gz~zXDmJpzNJPIvO7GBazi1rMaV1XEp1yyC0VHZN3H6F1rAWF4fiQVJHzmDJ3Wja1eQmIGJlFm46Z0rQsPeaUAAOM-5dYAkP8VA39x3uXEgRuwKyQ-4nfOLPsSqc3obPuTzUZBMGErvpuDVnO8QhRN5UBNRo~wJGORevD3vn7hyN9ZCLzkxPpCXzq6fMGEWtPpxauN1OaeI1Wgpikpps3e2rosZ2QEl73wCaORZddIwURn2LTJKODkbZh-1ETgpJ2ShqGt1v69tMvlSTgO-9k5Wjd31fUA__&Key-Pair-Id=APKAI7S6A5RYOXBWRPDA"
#或者
wget -O cellranger-7.0.0.tar.gz "https://cf.10xgenomics.com/releases/cell-exp/cellranger-7.0.0.tar.gz?Expires=1656187885&Policy=eyJTdGF0ZW1lbnQiOlt7IlJlc291cmNlIjoiaHR0cHM6Ly9jZi4xMHhnZW5vbWljcy5jb20vcmVsZWFzZXMvY2VsbC1leHAvY2VsbHJhbmdlci03LjAuMC50YXIuZ3oiLCJDb25kaXRpb24iOnsiRGF0ZUxlc3NUaGFuIjp7IkFXUzpFcG9jaFRpbWUiOjE2NTYxODc4ODV9fX1dfQ__&Signature=FEFSd-63q1ZuJwFOGD5KlH-OpL7Rzpj9UqBC7lOgRM8v4C1fJ9ndTwv8gz~zXDmJpzNJPIvO7GBazi1rMaV1XEp1yyC0VHZN3H6F1rAWF4fiQVJHzmDJ3Wja1eQmIGJlFm46Z0rQsPeaUAAOM-5dYAkP8VA39x3uXEgRuwKyQ-4nfOLPsSqc3obPuTzUZBMGErvpuDVnO8QhRN5UBNRo~wJGORevD3vn7hyN9ZCLzkxPpCXzq6fMGEWtPpxauN1OaeI1Wgpikpps3e2rosZ2QEl73wCaORZddIwURn2LTJKODkbZh-1ETgpJ2ShqGt1v69tMvlSTgO-9k5Wjd31fUA__&Key-Pair-Id=APKAI7S6A5RYOXBWRPDA"
- 1
- 2
- 3
- 4
2. 参考基因组下载
Cell Ranger引用通常是向后兼容的,匹配正确的版本即可。
curl -O https://cf.10xgenomics.com/supp/cell-exp/refdata-gex-GRCh38-2020-A.tar.gz
#或者通过wget下载
wget -c https://cf.10xgenomics.com/supp/cell-exp/refdata-gex-GRCh38-2020-A.tar.gz
tar -zxvf refdata-gex-GRCh38-2020-A.tar.gz
- 1
- 2
- 3
- 4
- 5
3. 下载例子数据
下载地址:
https://www.10xgenomics.com/resources/datasets?menu%5Bproducts.name%5D=Single%20Cell%20Gene%20Expression&query=&page=1&configure%5Bfacets%5D%5B0%5D=chemistryVersionAndThroughput&configure%5Bfacets%5D%5B1%5D=pipeline.version&configure%5BhitsPerPage%5D=500
进入页面后发现这里有全部的数据类型,根据自己需要分析的内容选择合适的测试数据。
# 4k PBMCs from a Healthy Donor(4,340 cells)36.38 GB (Analysis run with --expect-cells=5000)
nohup wget -c 'http://s3-us-west-2.amazonaws.com/10x.files/samples/cell-exp/2.1.0/pbmc4k/pbmc4k_fastqs.tar' &
# 1k PBMCs from a Healthy Donor (996 cells ,v2 chemistry) 5.94 GB (run with --expect-cells=1000)
wget -c http://cf.10xgenomics.com/samples/cell-exp/3.0.0/pbmc_1k_v2/pbmc_1k_v2_fastqs.tar
- 1
- 2
- 3
- 4
- 5
4. 单细胞固定RNA分析探针装置
确定这些输入参数并自定义红色代码后,运行cellranger:
cd /home/jdoe/runs
cellranger count --id=sample345 \
--transcriptome=/opt/refdata-gex-GRCh38-2020-A \
--fastqs=/home/jdoe/runs/HAWT7ADXX/outs/fastq_path \
--sample=mysample \
--localcores=8 \
--localmem=64
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
在一系列验证输入参数的检查之后,cellranger count管道阶段将开始运行:
Martian Runtime - v7.0.0
Running preflight checks (please wait)...
Checking sample info...
Checking FASTQ folder...
Checking reference...
Checking optional arguments...
...
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
默认情况下,Cell Ranger将使用系统上所有可用的核心来执行管道阶段。你可以用——localcores选项指定不同数量的内核;例如——localcores=16将限制Cell Ranger一次最多使用16个核心。同样,——localmem将限制Cell Ranger使用的内存量(以GB为单位)。该管道将为其输出创建一个以您指定的样例ID命名的新文件夹(例如:/home/jdo /run /sample345)。如果这个文件夹已经存在,Cell Ranger将假设它是一个现有的管道,并试图恢复运行它。
输出文件 一个成功的cellranger count运行应该以类似这样的消息结束
Outputs: - Run summary HTML: /opt/sample345/outs/web_summary.html - Run summary CSV: /opt/sample345/outs/metrics_summary.csv - BAM: /opt/sample345/outs/possorted_genome_bam.bam - BAM index: /opt/sample345/outs/possorted_genome_bam.bam.bai - Filtered feature-barcode matrices MEX: /opt/sample345/outs/filtered_feature_bc_matrix - Filtered feature-barcode matrices HDF5: /opt/sample345/outs/filtered_feature_bc_matrix.h5 - Unfiltered feature-barcode matrices MEX: /opt/sample345/outs/raw_feature_bc_matrix - Unfiltered feature-barcode matrices HDF5: /opt/sample345/outs/raw_feature_bc_matrix.h5 - Secondary analysis output CSV: /opt/sample345/outs/analysis - Per-molecule read information: /opt/sample345/outs/molecule_info.h5 - CRISPR-specific analysis: null - Loupe Browser file: /opt/sample345/outs/cloupe.cloupe - Feature Reference: null - Target Panel File: null Waiting 6 seconds for UI to do final refresh. Pipestance completed successfully! yyyy-mm-dd hh:mm:ss Shutting down. Saving pipestance info to "tiny/tiny.mri.tgz"
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
当数据是单细胞靶向基因表达分析,需要注意一下我们多输入一个文件,设置 --target-panel 参数。
cd /home/jdoe/runs
cellranger count --id=sample345 \
--target-panel=/opt/cellranger-5.0.0/target_panels/immunology_v1.0_GRCh38-2020-A.target_panel.csv \
--transcriptome=/opt/refdata-gex-GRCh38-2020-A \
--fastqs=/home/jdoe/runs/HAWT7ADXX/outs/fastq_path \
--sample=mysample \
--localcores=8 \
--localmem=64
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
当数据是单细胞靶向基因表达和抗体捕获与CRISPR指南捕获分析,需要注意一下我们多输入两个文件,设置 --target-panel 和 --feature-ref 参数。
cellranger count --id=sample345 \
--target-panel=/opt/cellranger-5.0.0/target_panels/immunology_v1.0_GRCh38-2020-A.target_panel.csv \
--transcriptome=/opt/refdata-gex-GRCh38-2020-A \
--libraries=/path/to/library.csv \
--feature-ref=/path/to/feature_ref.csv \
--localcores=8 \
--localmem=64
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
这些都需要在服务器上使用,资源要求蛮高的,自己一般不具备这样的条件,可以联系桓峰基因。
桓峰基因,铸造成功的您!
有想进生信交流群的老师可以扫最后一个二维码加微信,备注“单位+姓名+目的”,有些想发广告的就免打扰吧,还得费力气把你踢出去!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
有想进生信交流群的老师可以扫最后一个二维码加微信,备注“单位+姓名+目的”,有些想发广告的就免打扰吧,还得费力气把你踢出去!
References:
- Luecken MD, Theis FJ. Current best practices in single-cell RNA-seq analysis: a tutorial. Mol Syst Biol. 2019;15(6):e8746. Published 2019 Jun 19. doi:10.15252/msb.20188746