- 1Python 打包配置文件 setup.py 详解_python 中setup.py在哪
- 2【云原生 | 从零开始学istio】二、Istio核心特性与架构
- 3unity karting导入时的编译问题_all compiler errors have to be
- 4小程序实现无限级树形菜单_小程序 树菜单
- 5“IT小百科”之“Windows自带的服务和系统进程详解”_windows服务 打开exef进程
- 6DRF学习之权限验证(十五)_permission_classes=[isauthenticated]
- 7分布式事务概念及理论
- 8#每日一题# 25. K 个一组翻转链表 - 20191021_给你一个链表,每 k 个节点一组进行翻转,请你返回翻转后的链表。 k 是一个正整数,
- 9开源大型语言模型概览:多语种支持与中文专注
- 10Unity pc端内嵌网页插件Embedded Browser基本使用流程(转载)
利用随机森林对特征重要性进行评估 方法一_可变重要性度量vim
赞
踩
https://hal.archives-ouvertes.fr/file/index/docid/755489/filename/PRLv4.pdf
前言
随机森林是以决策树为基学习器的集成学习算法。随机森林非常简单,易于实现,计算开销也很小,更令人惊奇的是它在分类和回归上表现出了十分惊人的性能,因此,随机森林也被誉为“代表集成学习技术水平的方法”。
本文是对随机森林如何用在特征选择上做一个简单的介绍。
随机森林(RF)简介
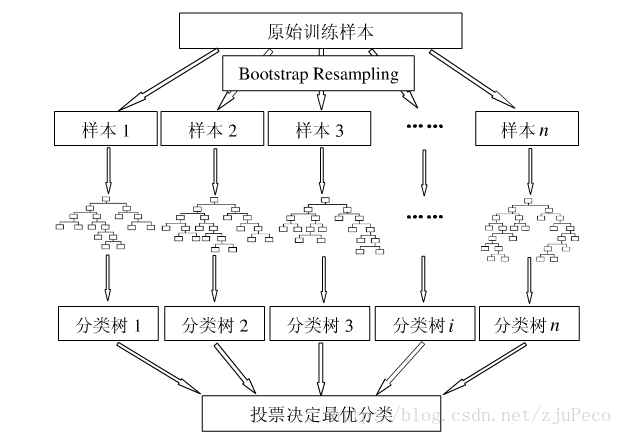
只要了解决策树的算法,那么随机森林是相当容易理解的。随机森林的算法可以用如下几个步骤概括:
- 用有抽样放回的方法(bootstrap)从样本集中选取n个样本作为一个训练集
- 用抽样得到的样本集生成一棵决策树。在生成的每一个结点:
- 随机不重复地选择d个特征
- 利用这d个特征分别对样本集进行划分,找到最佳的划分特征(可用基尼系数、增益率或者信息增益判别)
- 重复步骤1到步骤2共k次,k即为随机森林中决策树的个数。
- 用训练得到的随机森林对测试样本进行预测,并用票选法决定预测的结果。
下图比较直观地展示了随机森林算法(图片出自文献2):
图1:随机森林算法示意图
没错,就是这个到处都是随机取值的算法,在分类和回归上有着极佳的效果,是不是觉得强的没法解释~
然而本文的重点不是这个,而是接下来的特征重要性评估。
特征重要性评估
现实情况下,一个数据集中往往有成百上前个特征,如何在其中选择比结果影响最大的那几个特征,以此来缩减建立模型时的特征数是我们比较关心的问题。这样的方法其实很多,比如主成分分析,lasso等等。不过,这里我们要介绍的是用随机森林来对进行特征筛选。
用随机森林进行特征重要性评估的思想其实很简单,说白了就是看看每个特征在随机森林中的每颗树上做了多大的贡献,然后取个平均值,最后比一比特征之间的贡献大小。
好了,那么这个贡献是怎么一个说法呢?通常可以用基尼指数(Gini index)或者袋外数据(OOB)错误率作为评价指标来衡量。
我们这里只介绍用基尼指数来评价的方法,想了解另一种方法的可以参考文献2。
我们将变量重要性评分(variable importance measures)用VIMVIM来表示,将Gini指数用GIGI来表示,假设有mm个特征X1,X2,X3,...,XcX1,X2,X3,...,Xc,现在要计算出每个特征XjXj的Gini指数评分VIM(Gini)jVIMj(Gini),亦即第jj个特征在RF所有决策树中节点分裂不纯度的平均改变量。
Gini指数的计算公式为
GIm=∑|K|k=1∑k′≠kpmkpmk′=1−∑|K|k=1p2mkGIm=∑k=1|K|∑k′≠kpmkpmk′=1−∑k=1|K|pmk2
其中,KK表示有KK个类别,pmkpmk表示节点mm中类别kk所占的比例。
直观地说,就是随便从节点mm中随机抽取两个样本,其类别标记不一致的概率。
特征XjXj在节点mm的重要性,即节点mm分枝前后的GiniGini指数变化量为
VIM(Gini)jm=GIm−GIl−GIrVIMjm(Gini)=GIm−GIl−GIr
其中,GIlGIl和GIrGIr分别表示分枝后两个新节点的GiniGini指数。
如果,特征XjXj在决策树ii中出现的节点在集合MM中,那么XjXj在第ii颗树的重要性为
VIM(Gini)ij=∑m∈MVIM(Gini)jmVIMij(Gini)=∑m∈MVIMjm(Gini)
假设RFRF中共有nn颗树,那么
VIM(Gini)j=∑ni=1VIM(Gini)ijVIMj(Gini)=∑i=1nVIMij(Gini)
最后,把所有求得的重要性评分做一个归一化处理即可。
VIMj=VIMj∑ci=1VIMiVIMj=VIMj∑i=1cVIMi
举个例子
值得庆幸的是,sklearnsklearn已经帮我们封装好了一切,我们只需要调用其中的函数即可。
我们以UCI上葡萄酒的例子为例,首先导入数据集。
- import pandas as pd
- url = 'http://archive.ics.uci.edu/ml/machine-learning-databases/wine/wine.data'
- df = pd.read_csv(url, header = None)
- df.columns = ['Class label', 'Alcohol', 'Malic acid', 'Ash',
- 'Alcalinity of ash', 'Magnesium', 'Total phenols',
- 'Flavanoids', 'Nonflavanoid phenols', 'Proanthocyanins',
- 'Color intensity', 'Hue', 'OD280/OD315 of diluted wines', 'Proline']
- 1
- 2
- 3
- 4
- 5
- 6
- 7
然后,我们来大致看下这时一个怎么样的数据集
- import numpy as np
- np.unique(df['Class label'])
- 1
- 2
输出为
array([1, 2, 3], dtype=int64)- 1
可见共有3个类别。然后再来看下数据的信息:
df.info()- 1
输出为
- <class 'pandas.core.frame.DataFrame'>
- RangeIndex: 178 entries, 0 to 177
- Data columns (total 14 columns):
- Class label 178 non-null int64
- Alcohol 178 non-null float64
- Malic acid 178 non-null float64
- Ash 178 non-null float64
- Alcalinity of ash 178 non-null float64
- Magnesium 178 non-null int64
- Total phenols 178 non-null float64
- Flavanoids 178 non-null float64
- Nonflavanoid phenols 178 non-null float64
- Proanthocyanins 178 non-null float64
- Color intensity 178 non-null float64
- Hue 178 non-null float64
- OD280/OD315 of diluted wines 178 non-null float64
- Proline 178 non-null int64
- dtypes: float64(11), int64(3)
- memory usage: 19.5 KB

- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
可见除去class label之外共有13个特征,数据集的大小为178。
按照常规做法,将数据集分为训练集和测试集。
- from sklearn.cross_validation import train_test_split
- from sklearn.ensemble import RandomForestClassifier
- x, y = df.iloc[:, 1:].values, df.iloc[:, 0].values
- x_train, x_test, y_train, y_test = train_test_split(x, y, test_size = 0.3, random_state = 0)
- feat_labels = df.columns[1:]
- forest = RandomForestClassifier(n_estimators=10000, random_state=0, n_jobs=-1)
- forest.fit(x_train, y_train)
- 1
- 2
- 3
- 4
- 5
- 6
- 7
好了,这样一来随机森林就训练好了,其中已经把特征的重要性评估也做好了,我们拿出来看下。
- importances = forest.feature_importances_
- indices = np.argsort(importances)[::-1]
- for f in range(x_train.shape[1]):
- print("%2d) %-*s %f" % (f + 1, 30, feat_labels[indices[f]], importances[indices[f]]))
- 1
- 2
- 3
- 4
输出的结果为
- 1) Color intensity 0.182483
- 2) Proline 0.158610
- 3) Flavanoids 0.150948
- 4) OD280/OD315 of diluted wines 0.131987
- 5) Alcohol 0.106589
- 6) Hue 0.078243
- 7) Total phenols 0.060718
- 8) Alcalinity of ash 0.032033
- 9) Malic acid 0.025400
- 10) Proanthocyanins 0.022351
- 11) Magnesium 0.022078
- 12) Nonflavanoid phenols 0.014645
- 13) Ash 0.013916
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
对的就是这么方便。
如果要筛选出重要性比较高的变量的话,这么做就可以
- threshold = 0.15
- x_selected = x_train[:, importances > threshold]
- x_selected.shape
- 1
- 2
- 3
输出为
(124, 3)- 1
瞧,这不,帮我们选好了3个重要性大于0.15的特征了吗~
参考文献
[1] Raschka S. Python Machine Learning[M]. Packt Publishing, 2015.
[2] 杨凯, 侯艳, 李康. 随机森林变量重要性评分及其研究进展[J]. 2015.