- 1渗透测试之内网渗透(四):端口转发_socat 端口转发
- 2如何在VSCode上新建Flutter项目【两步搞定】_vscode fulltter项目
- 3内卷了!阿里 Java 八股文面试题“惨遭”泄露,导致 132 人面进大厂_阿里java八股文
- 4苹果手机支持鸿蒙,全球第三大手机系统「鸿蒙」上线,这19款能抢先用…
- 5Win10系统更新后【开机黑屏/白屏,不显示桌面,但是鼠标和任务管理器都好使】问题的解决_csdn win10更新黑屏
- 6java 子线程定时去更改主线程的变量
- 7大数据在金融行业的应用_大数据技术在商务银行的应用图表
- 8python坐标字符串_字符串坐标的numpy mgrid shapely
- 9linux kernel 5.1编译8723ds报错_vfs_internal_i_am_really_a_filesystem_and_am_not_a
- 10IOS MJRefresh:下拉刷新与上拉加载_ios开发 mjrefresh上拉加载
Nature Methods | 张阳团队开发远超AlphaFold2精度的蛋白互作结构预测算法
赞
踩
基因是构造生命的基本蓝图,而蛋白质则是生命功能的执行者和生命现象的体现者。细胞中的蛋白质主要是通过与细胞内其它蛋白质的相互作用来实现其绝大部分生物学功能。因此,蛋白质-蛋白质相互作用(简称“蛋白质互作”)在生命功能的实现以及生物的进化过程中都扮演极其重要的角色。例如,抗体和抗原蛋白相互作用可以帮助生命个体识别和抵御外界病原体的入侵;受体和配体蛋白相互作用可以触发细胞信号传导通路;酶蛋白和底物相互作用可以催化新陈代谢的进程等等。它们在生物功能上的这种特殊的重要性,也使得蛋白质互作成为许多现代药物设计的关键靶点。
相应地,如何确定蛋白质复合物的三维结构、利用蛋白质互作的结构信息指导蛋白质功能注解和药物设计,一直是现代生物医学的一个重要问题。在传统的结构生物学研究中,人们可以使用X射线晶体衍射、核磁共振及冷冻电镜等实验手段去解析蛋白质复合物的原子结构。但是结构生物学实验往往耗费大量人力物力,无法用于大规模获取蛋白质组学级别的分子结构信息。并且,因为技术上的限制(如结构的稳定性以及高阶复合物的尺度等),某些蛋白质相互作用的结构并不能通过传统的结构生物学实验手段获得。
近年来,随着AlphaFold2等AI算法在蛋白质结构预测问题上的突破,通过计算手段获得高质量的蛋白质及复合物结构已经成为可能。然而,目前绝大多数AI算法都是通过对已知蛋白结构和基因进化关系的训练和学习来构造蛋白结构,而基因进化的信息主要是从现有基因序列的多序列比对(MSA)来获得。因此,这些AI算法的实际预测精度,往往依赖于目标蛋白MSA的质量,尤其在同源序列较少的孤儿蛋白(orphan proteins)和蛋白质复合物上的表现并不尽如人意。
近日,新加坡国立大学和美国密歇根大学张阳教授团队在《Nature Methods》杂志上发表了题为《Improving deep learning protein monomer and complex structure prediction using DeepMSA2 with huge metagenomics data》的论文(图1)。该研究工作开发了两种新的算法来提高蛋白质互作的结构预测精度。
首先,作者开发了DeepMSA2,利用递推动态规划和隐马尔科夫模型算法,从海量宏基因组(metagenome)序列库中快速提取高质量的MSA数据。然后又利用新开发的DMFold算法构建蛋白质复合物的三维结构。实验结果显示:DMFold/DeepMSA2对蛋白质复合物的结构预测精度要显著优于AlphaFold2等算法,且在抗体-抗原复合物结构预测问题上表现尤为突出。特别地,DMFold(https://zhanggroup.org/DMFold)算法在最近一届蛋白质结构预测大赛(CASP15)的蛋白质复合物结构预测比赛中获得冠军。密歇根大学郑伟博士为该论文的第一作者,张阳教授和P. Lydia Freddolino教授为论文共同通讯作者。

图1. 张阳团队在《Nature Methods》的发表论文
一、背景介绍
在蛋白质及其复合物预测中,一个重要的问题就是如何构建多重序列比对,即MSA。因为MSA所包含的基因和蛋白质的共进化信息,可以作为深度学习模型空间约束预测的特征输入。具体来讲,如果在进化过程中,蛋白质的其中一个氨基酸位点发生了突变,且破坏了氨基酸残基间的相互作用,则该蛋白会变得不稳定,拥有该突变的物种则不容易存活下来。但是如果与其存在空间相互作用的氨基酸同时也发生了突变,且这两个突变又能够很好的作用在一起,使得蛋白质结构继续稳定下来,这种生物也就可以继续存活下来。这种现象被称为基因和蛋白质共进化。所以,利用多序列比对可以有效推导出蛋白质共进化以及氨基酸空间距离的信息。因此,MSA及共进化信息被广泛应用于基于AI的蛋白质三级结构预测中。
对于蛋白质互作形成的复合物,其MSA可以通过“合理”拼接蛋白质单体的MSA获得(图2)。但是,构造有效合理的蛋白质互作MSA需要满足一定条件。首先,单体MSA的同源序列必须要达到一定的数目,以保证推导单体内部有效共进化信息的统计量;第二,更重要的是,蛋白质互作MSA需要提供蛋白质-蛋白质之间的空间演化信息。具体来讲,两个配对单体MSA的同源序列必须来自于同一个复合体才能配对。否则来自非互作或相互独立的同源序列之间不存在相互作用的共进化关系,它们的错误配对会对整体的互作MSA以及最后的蛋白质互作建模精度产生负面的影响。因为目前缺少大标度高精度的蛋白质互作序列数据库供互作MSA的直接搜索,如何从单体MSA构造有效的互作MSA,进而构建高精度蛋白质复合物结构预测仍然是一个巨大的挑战。
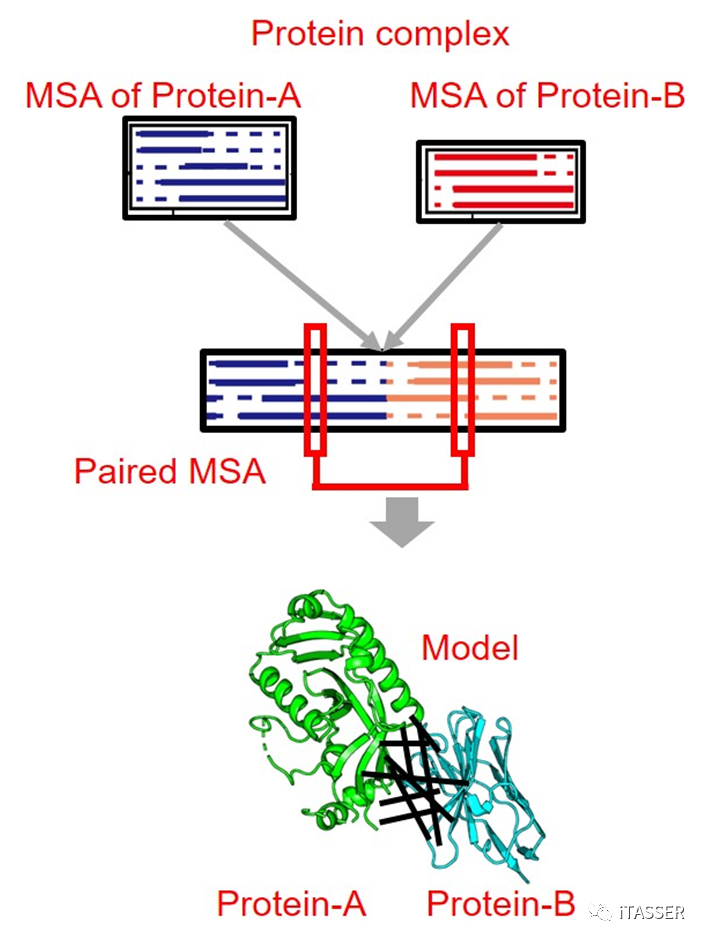
图2. 通过合理拼接单体MSA构建复合物MSA,进而预测蛋白质复合物结构
在这项研究中,张阳团队基于他们此前开发的DeepMSA迭代MSA构建算法,提出了改进版的DeepMSA2算法。DeepMSA2结合了一系列基于大标度宏基因组序列数据库的MSA生成策略,同时该方法结合了新的AI驱动的MSA打分策略来筛选高质量的MSA模型。定量的基准和双盲测试结果都表明,使用了DeepMSA2的结构预测算法DMFold可以构建远超AlphaFold2精度的蛋白质-蛋白质复合物结构。
二、方法简介
DeepMSA2算法包括DeepMSA2-Monomer单体MSA构建算法和DeepMSA2-Multimer复合物MSA构建算法。对于蛋白质单体MSA构建(图3),该方法利用三个基于不同搜索策略构建的并行模块(dMSA、qMSA和 mMSA)从不同的数据库中获取原始MSA。这些搜索策略均基于动态规划(dynamic programming)和隐马尔科夫模型(hidden Markov model)算法,而这些数据库则由多个全基因组和宏基因组序列库组装而成。三个MSA生成模块都遵循相似的逻辑:首先,从序列数据库中检索初始的查询序列;如果未获得足够的有效序列,则对更大的数据库进行迭代搜索;最后,利用AI深度学习预测的蛋白质结构置信打分对三个模块收集到的原始MSA进行排序,选择出最佳MSA。
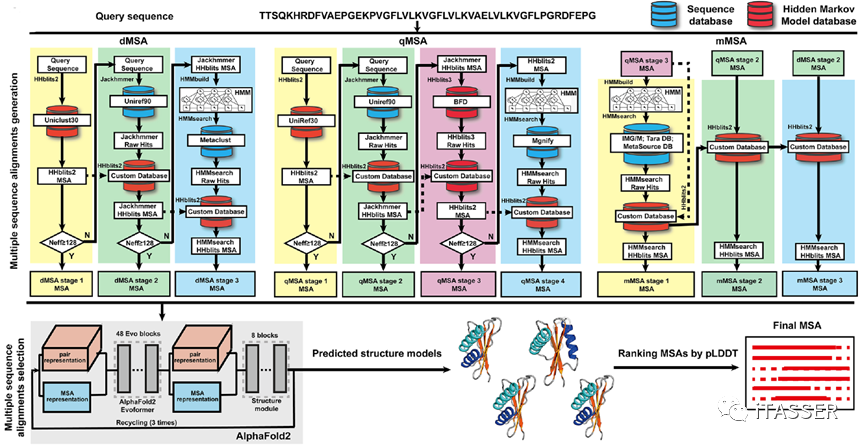
图3. DeepMSA2蛋白质单体MSA构建算法(DeepMSA2-Monomer)
对于蛋白质复合物MSA 构建(图 4),每条组分链(组成蛋白质复合物的每个子链)都可以通过单体MSA构建步骤得到10个排好序的MSA,可选取每条组分链的前M个排名最高的单体MSA与所有其他链的单体MSA配对,从而得到MN个联合MSA(N是复合体中不同组分单链的数量)。然后,每个联合MSA中的序列通过连接来自不同组分单链的具有相同起源(物种)的蛋白质单体序列来创建复合物的MSA。最后根据联合MSA深度的和单体MSA置信分数的综合得分来选择最佳复合物MSA。
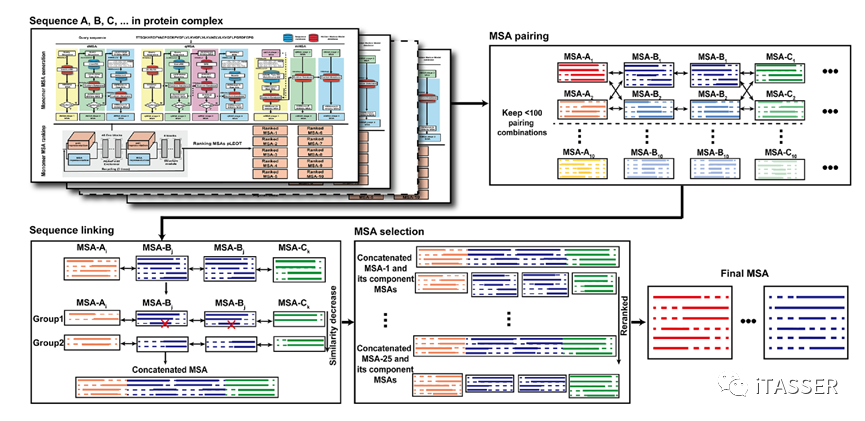
图4. DeepMSA2蛋白质复合物MSA构建的算法(DeepMSA2-Multimer)
通过将DeepMSA2算法与AlphaFold2端到端结构训练模块结合,该研究提出了蛋白质单体及复合物结构预测算法DMFold。DMFold算法包括两个部分,蛋白质单体结构预测算法DMFold-Monomer和蛋白质复合物结构预测算法DMFold-Multimer。
三、结果分析
(1) 提升蛋白质单体结构预测精度
这项研究首先在蛋白质单体预测问题上与AlphaFold2算法进行了比较。图5展示了DMFold(DMFold-Monomer,后文简称DMFold)与AlphaFold2在132个非冗余的困难蛋白质单体上的TM-score比较。TM-score是由张阳实验室首先提出、后被业界广泛使用的一种用来评估蛋白质结构预测准确与否的指标。TM-score=0代表最差预测,而TM-score=1代表预测的和真实的结构相同。TM-score=0.5表明预测的模型和真实结构有着相同的全局拓扑结构(fold或topology)。总的来说,DMFold生成的模型的TM-score比AlphaFold2高出5%(0.82 vs 0.78)。对于特别困难的蛋白质(图5所示的红色框内),DMFold的精度要显著地优于AlphaFold2(TM-score=0.63 vs 0.52)。
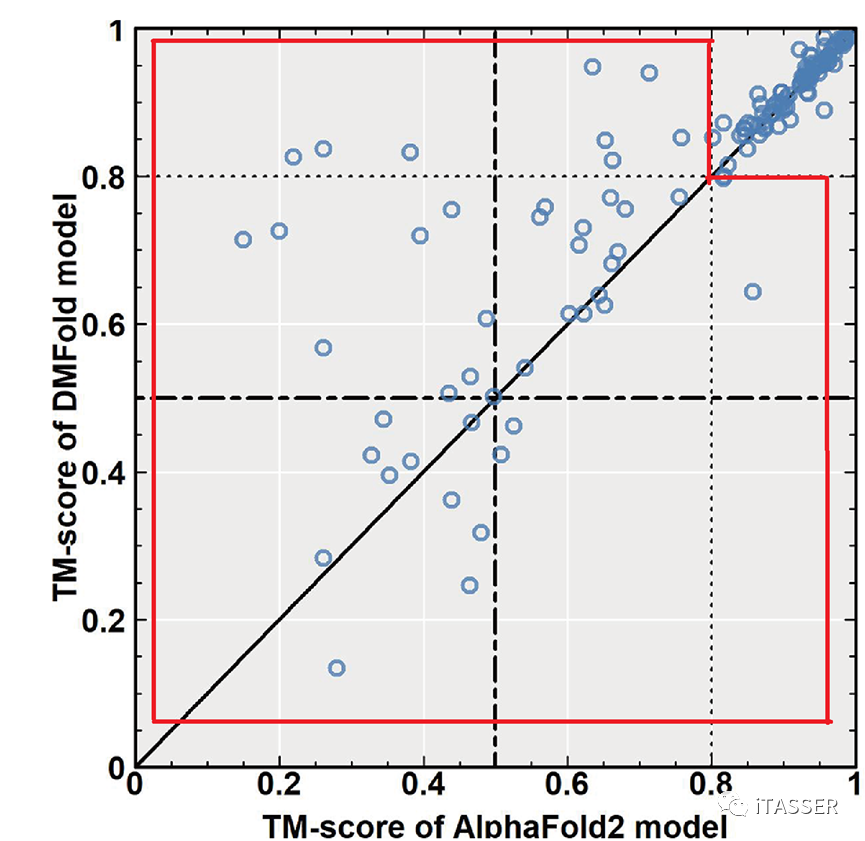
图5. DMFold算法(Y轴)与AlphaFold2算法(X轴)预测蛋白质单体结构结果的比较
图6以一种RNA聚合酶(PDB ID: 6vr4)作为示例比较了DMFold与AlphaFold2。AlphaFold2预测出的结构模型的TM-score为0.20,全局pLDDT分数为0.40。而DMFold结构模型的TM-score为0.73,全局pLDDT分数为0.71。pLDDT分数是AlphaFold2用来评估结构预测质量的置信系数,pLDDT≥0.7表示预测的结构可能正确,pLDDT<0.7表示预测的结构可能错误。从结果来看,DMFold预测出的结构模型能得到更高的TM-score精度以及pLDDT置信系数。AlphaFold2预测结果较差的一个主要原因就是因为其MSA构建算法只能检索到2条同源序列,导致氨基酸位点上能够检测到的共进化信息不足。相反,DMFold使用DeepMSA2构建的MSA,其包含了42条同源序列,所以每个位点上的共进化信息较强。进一步地研究结果表明,氨基酸层面的pLDDT打分与每个氨基酸位点的同源比对氨基酸数量(Nr)呈正相关。这一结果反映了MSA的质量与蛋白质单体结构预测的精度和置信系数是紧密相关的。
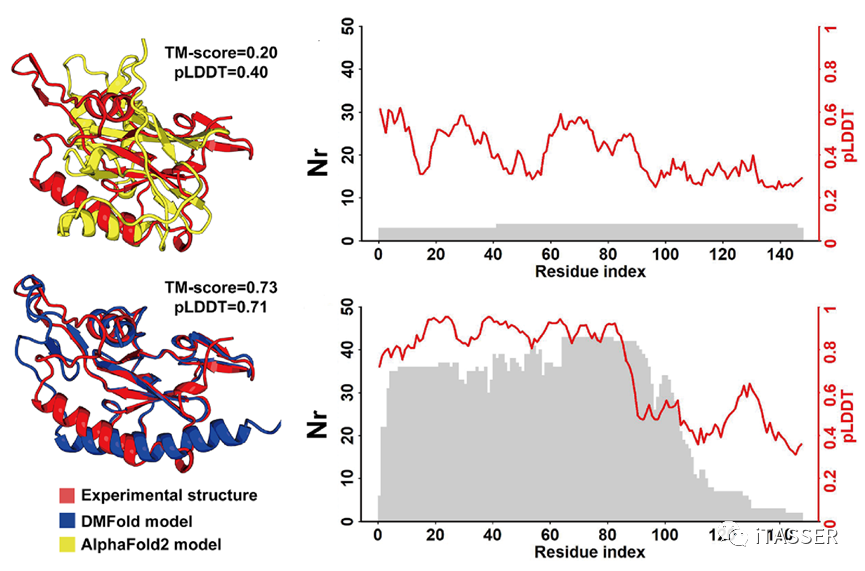
图6. DNA依赖性RNA聚合酶上AlphaFold2预测的结构(黄色)与DMFold预测结构(蓝色)精度的比较,Nr为每个氨基酸位点上MSA中包含的比对上的同源氨基酸数量
(2) 提升人类蛋白质组单体结构预测精度
为了验证该算法对于大规模结构建模的实用性,论文作者进一步将DMFold应用于人类蛋白质组的结构预测。作者选取了AlphaFold2 DB数据库中5042个AlphaFold2预测失败(pLDDT<0.7)的人类蛋白。根据DMFold和AlphaFold2 DB数据库对这5042个蛋白质的pLDDT分数直方图分布(图7A)显示,DMFold能够成功预测其中1934个人类蛋白的结构。对这1934个人类蛋白的残基水平pLDDT分数的进一步比较显示(图7B),对于93%残基,DMFold预测的氨基酸水平置信系数比相应的AlphaFold2的结果更高。
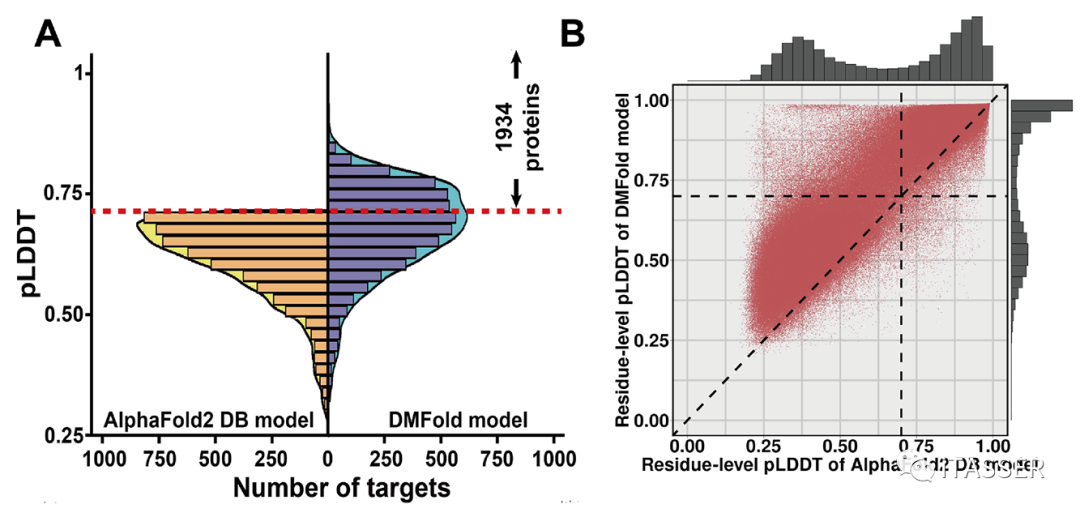
图7. DMFold预测的5042个人类蛋白质结构精度与AlphaFold2的比较
作者认为,DMFold预测的人类蛋白组结构精度较AlphaFold2能够显著提升的原因主要来自于两方面(图8):一是DMFold能够显著的提升蛋白质的整体拓扑结构。例如,在Q6ZQT0这个蛋白上,AlphaFold2形成了一个桶装卷曲雏形,而DMFold预测的结构形成了明显的β桶装结构,观测其pLDDT分布,可以看出DMFold在每个氨基酸上的预测精度都有大幅提升。二是DMFold能够优化蛋白质局部的二级结构。例如在Q6IED9这个蛋白上,AlphaFold2预测的结构与DMFold预测的模型具有相同的拓扑结构。但是,如果观察两个模型的差异,会发现DMFold在红色高亮的局部片段区域形成了明显的β片层,并且相应区域的pLDDT打分也有明显提升。
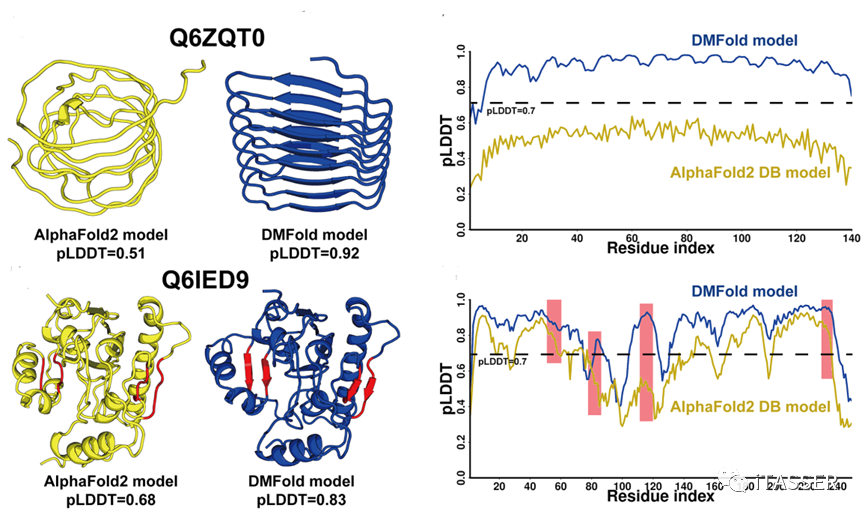
图8. 在两个人类蛋白组蛋白上AlphaFold2预测的结构(黄色)与DMFold预测结构(蓝色)精度的比较。
(3) 提升蛋白质-蛋白质复合物结构预测精度
为了测试DMFold对蛋白质互作的结构预测能力(即DMFold-Multimer),该研究从早期CASP比赛(即CASP13和CASP14)中,收集了54个蛋白质复合物。这些复合物由2到8条单链蛋白组成,其中有14个是异源复合物,40个是同源复合物。图9比较了DMFold-Multimer与AlphaFold2-Multimer对这54个蛋白复合物的结构预测精度。平均来讲,DMFold-Multimer预测的模型的TM-score为0.83,而AlphaFold2-Multimer的精度为0.74。对于特别困难的蛋白质复合物(如图9所示的红色框内),DMFold-Multimer的精度(0.72)较AlphaFold2-Multimer的预测精度(0.54)有着33%的显著提升。
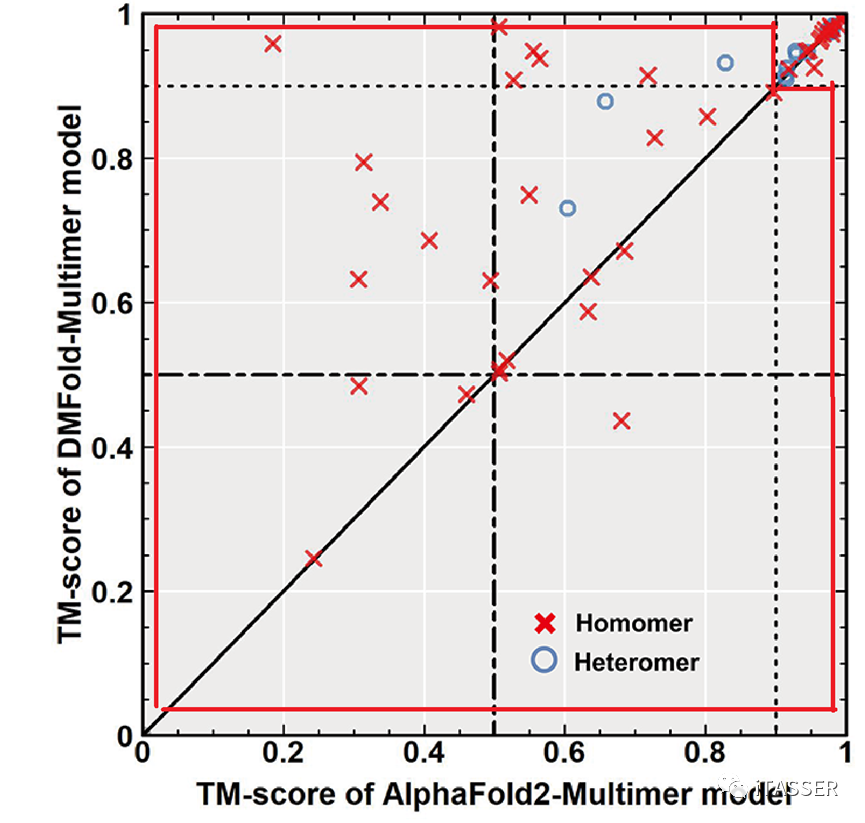
图9. DMFold-Multimer算法(Y轴)与AlphaFold2-Multimer算法(X轴)对蛋白质复合物结构预测的结果比较
作者分析指出,DMFold-Multimer之所以能够产生高质量互作模型的原因主要在于两方面:一是DeepMSA2-Multimer使用了新颖的MSA构建、排序、配对和选择机制。如图10所示,即使DMFold-Multimer与AlphaFold2-Multimer使用相同的数据库,其对复合物的预测精度(绿色柱)也是优于AlphaFold2-Multimer的(紫色柱),说明DeepMSA2-Multimer的MSA构造机制优于AlphaFold2-Multimer;二是DeepMSA2-Multimer使用了作者创建的一个包含约400亿个蛋白质序列的宏基因组数据库。当使用该数据库后(黄色柱),DMFold-Multimer的预测精度相较于只使用与AlphaFold2-Multimer相同的数据库的版本,有了更进一步的提升。说明更大的宏基因组数据库对蛋白质复合物结构预测是有明显影响的。
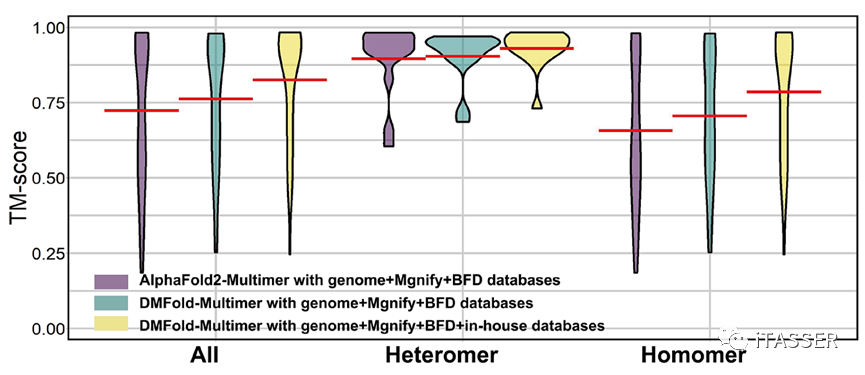
图10. DMFold-Multimer算法使用不同数据库与AlphaFold2-Multimer算法对蛋白质复合物结构预测的结果比较
进一步地,图11通过两个代表性例子表明了DMFold-Multimer主要能够在两个方面提升蛋白质复合物的精度:一是提升组成蛋白质复合物的单体蛋白的预测精度(图11,T0988o);二是提升单体蛋白间的相对扭转位置(既蛋白质接触面)的预测精度(图11,T1038o)。这些结果表明,互作MSA的正确构建对于蛋白质复合物结构预测至关重要。
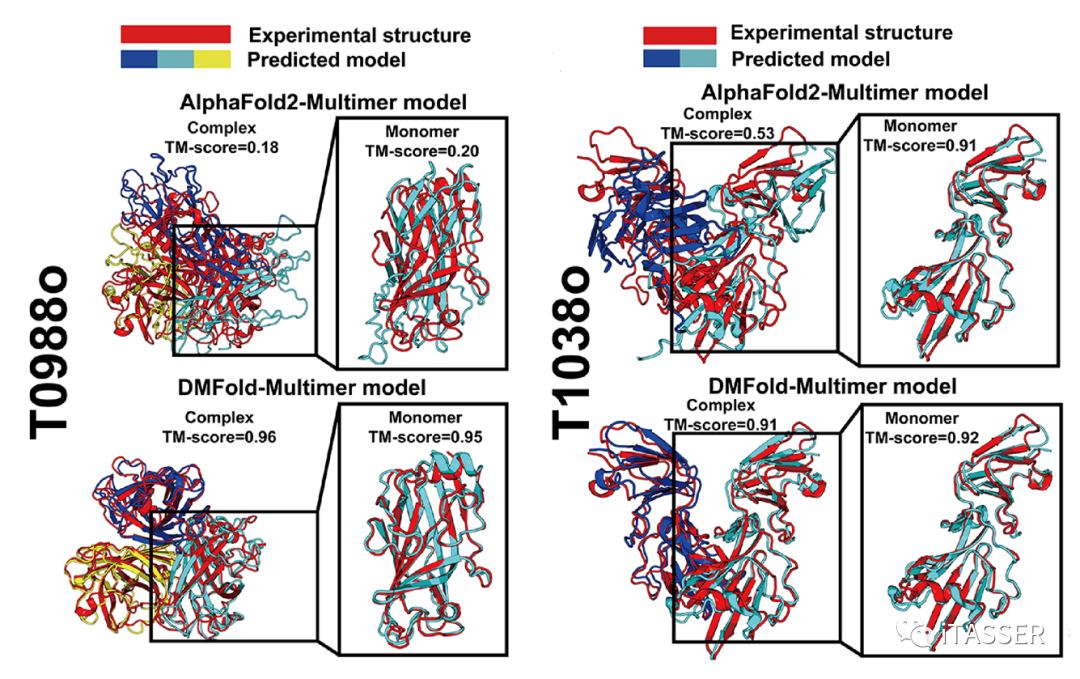
图11. DMFold-Multimer算法与AlphaFold2-Multimer算法在T0988o及T1038o两种蛋白质复合物上结构预测结果的比较。
(4) CASP大赛盲测结果
DMFold-Multimer算法(参赛名:Zheng)参加了2022年举行的最近一届蛋白质结构预测大赛(CASP15),并在蛋白质-蛋白质复合物预测比赛中排名第一。CASP是世界范围的蛋白质结构预测比赛,每两年举行一次,旨在对领域内的蛋白质结构预测技术做出客观的测试和评价。CASP采用严格的双盲预测机制,被誉为评估蛋白质结构预测技术的金标准,也被业界视为“蛋白质结构预测的奥林匹克”。张阳团队在过去16年举办的八届CASP大赛中(CASP7-14),均取得了优异的成绩,连续八次取得自动组蛋白质结构预测冠军。
蛋白质-蛋白质相互作用结构预测是CASP近年来增加的新赛道。按照CASP15组委会对蛋白质互作结构预测给出的官方Z-score打分结果,DMFold-Multimer整体打分(35.4)比标准版的AlphaFold2-Multimer(12.3)高出2.9倍;同时也比所以参赛团队的第二名(29.9)和第三名(28.4)分别高出18%和25%(图12)。
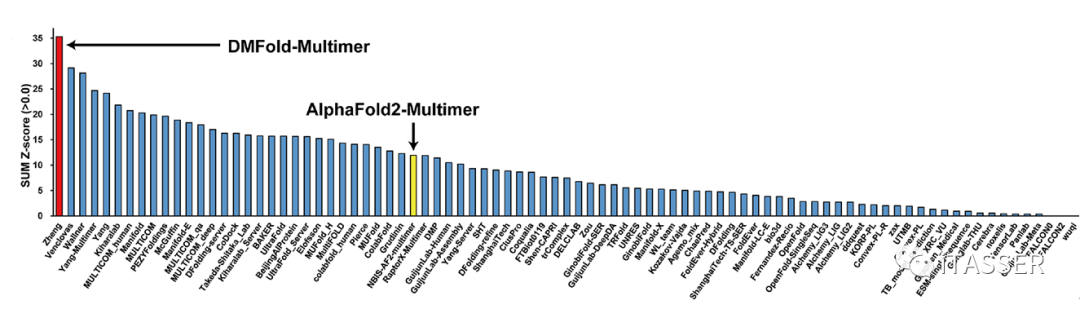
图12. CASP15世界蛋白质结构预测大赛蛋白质-蛋白质复合物预测比赛排名
图13进一步展示了DMFold-Multimer算法在27个蛋白质复合物上双盲预测的结构模型(彩色)与实验结构(黑色)的结构重叠比对。DMFold-Multimer构建的结构模型的TM-score均大于0.8。这些成功的例子中包括7个超大尺寸的复合物(H1111、H1114、H1137、T1170o、H1171、H1172和T1181o),它们分别包含8460、7988、4592、1908、1956、2004和2064个氨基酸残基,而DMFold-Multimer预测模型的TM-score分别为0.98、0.91、0.94、0.93、0.93、0.91和0.85。其中,最为复杂的复合物是H1137,其化学计量式为“A1B1C1D1E1F1G2H1I1”,包含9种不同的蛋白质的10条子链,而DMFold-Multimer对该互作结构预测的TM-score为0.94。长期以来,对大尺寸蛋白质复合物的结构预测被认为是分子生物学的一个重大挑战。这些结果表明,DMFold-Multimer算法能够对许多超大尺寸蛋白质-蛋白质复合物结构进行精准预测;这一成功标志着该领域朝向最终解决蛋白质互作结构模建问题迈出了重要的一步。
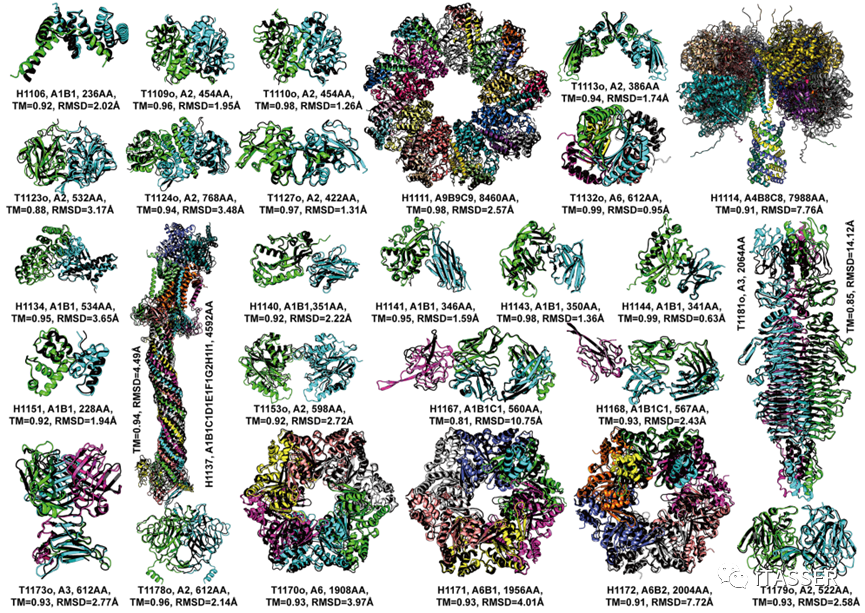
图13. DMFold-Multimer算法在27个蛋白质互作复合物上预测的结构模型(彩色)与实验结构(黑色)的结构比较
抗体-抗原的相互作用对人体抵御外界病原体的入侵起着至关重要的作用。一般的普通抗体包括两个重链和两个轻链,形成了复杂的Y型结构。而纳米抗体是一种只含有一个可变结构域的特殊抗体,它通过与抗原相互作用启动关键的免疫反应。图14展示了DMFold-Multimer和AlphaFold2-Multimer对CASP15中三个纳米抗体-抗原复合物(H1140、H1141和H1144)的结构预测结果。其中抗原均为小鼠的CNPase,而抗体则是三种不同的纳米抗体。AlphaFold2-Multimer算法预测的复合物模型的TM-score相对较低,均低于0.7。相比之下,DMFold-Multimer表现出了卓越的纳米抗体-抗原复合物预测能力,其所构建的三个结构模型的TM-score分别为0.92、0.95和0.99,几乎可以媲美实验解析的结构精度。
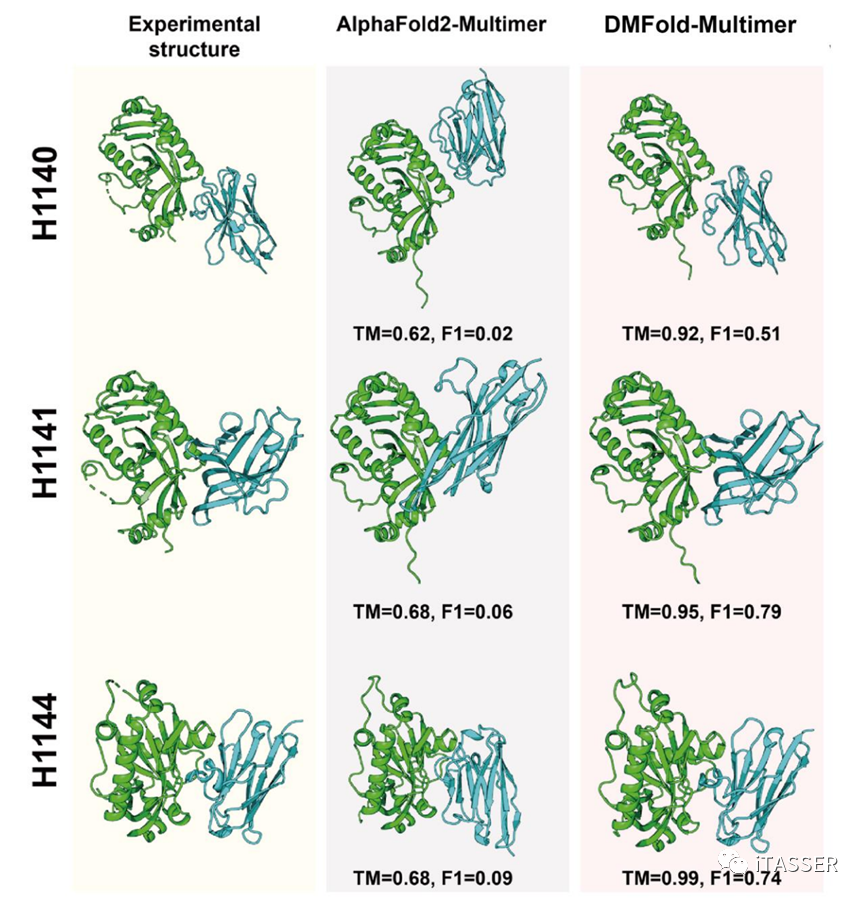
图14. CASP15比赛中纳米抗体-抗原复合物的案例分析
四、结论与展望
本研究开发了两种基于AI的蛋白质互作结构预测软件。其中DeepMSA2可用于从宏基因序列数据库中搜索提取多序列比对和蛋白质互作共进化信息,而DMFold则用于从MSA和共进化数据中构建蛋白质复合物三维结构模型。实验表明,DeepMSA2算法可以显著提高蛋白质复合物结构预测的准确性。与现有的MSA构建方法相比,DeepMSA2的一大优势在于迭代搜索和基于深度学习模型的预选策略,这种快速迭代搜索策略使得该算法可以检索海量的宏基因组序列数据库,有助于增加所得MSA的多样性和覆盖范围。另外,DeepMSA2的多MSA的组合策略也极大地丰富了蛋白质复合物中联合MSA所包含的共进化信息,进而提升蛋白质复合物结构预测的精度。正是因为DeepMSA2对共进化信息的有效提取,DMFold表现出了卓越的蛋白互作结构的预测能力。对某些特殊蛋白质复合物,包括纳米抗体-抗原相互作用,DMFold构建的结构模型几乎可以和实验解析的结构相媲美。
虽然DeepMSA2/DMFold的最初目标是针对蛋白质-蛋白质复合物四级结构的构建,但是它们也可以同时用于对蛋白质单体三级结构的预测。大规模的基准和双盲测试都表明,DeepMSA2/DMFold对蛋白质单体和复合物的结构预测都可以产生远超目前标准版本的AlphaFold2的预测精度。
尽管如此,作者认为DeepMSA2/DMFold仍然存在一些挑战。例如,DeepMSA2的复合体MSA是从单体MSA联合构建而来的。因此亟需解决的一个基本问题是如何有效连接不同组分MSA的序列以形成最佳复合物MSA。由于目前基于物种注释的序列连接机制仅适用于基因组序列,所以现有算法无法充分利用宏基因组数据库中信息丰富的同源序列来指导复合体结构组装。如何优化基于宏基因组的MSA配对与构建,对于进一步改进蛋白质互作精度以及相关研究(例如预测任意两个蛋白质是否为相互作用),都具有很大价值。
一直以来,蛋白质结构预测(包括蛋白互作结构预测)领域的研究重心主要是在开发新的深度学习算法和GPU资源来构造和训练新的AI模型。DeepMSA2和DMFold的成功也显示出,对于序列比对和共进化等输入特性的提取和优化,可能与AI算法和模型训练本身一样重要(如果不是更重要的话),应该引起业界足够的重视和进一步地研究。
五、相关结构预测在线服务器
1. Homologous Sequence Searching (同源序列检索):
https://zhanggroup.org/DeepMSA
2. Protein Complex Structure Prediction (蛋白质复合物结构预测):
https://zhanggroup.org/DMFold
3. Protein Structure Prediction (蛋白质单体结构和功能预测):
https://zhanggroup.org/D-I-TASSER
文章原文地址:
https://www.nature.com/articles/s41592-023-02130-4
高颜值免费 SCI 在线绘图(点击图片直达)
往期精品(点击图片直达文字对应教程)