- 1sha256加密_【总结】基于 FPGA 的 SHA256 高效数字加密研究(五)
- 2OpenCV直方图均衡化
- 3智公网:公务员行测基础考点
- 4Day17_集合与数据结构(链表,栈和队列,Map,Collections工具类,二叉树,哈希表)
- 5CVPR2022论文列表(中英对照)
- 6【unity3D】Transform组件(如何访问和获取Transform组件)_unity 获得transform
- 7用HTML5和JavaScript实现黑客帝国风格的字符雨效果
- 8Pytorch 训练与测试时爆显存(out of memory)的一个解决方案_pytorch 缓存
- 9MySQL5.7精简操作_mysql share目录精简
- 10Linux内核链表之list_head_linux list_head
【Mo 人工智能技术博客】特征选择和数据可视化_#数据可视化选择要分析的特征列
赞
踩
作者:支广达
数据对于我们大多数人来说,都是抽象无序的,今天就让我们来试一试,如何用python将抽象数据可视化为清晰明了的图表吧!
对从事算法研究或者数据分析的人来说,数据可视化可能并不是很受欢迎,毕竟数据可视化并不能给研究的内容带来直接的回报,而且制作过程可能比较枯燥,可以说是有点吃力不讨好。但是其实数据可视化可以潜在的让你更加了解你的数据,一个好的数据可视化思路,可以让你在着手自己研究的内容之前,指明方向从而少走弯路。
下面我们结合一些例子,来教大家如何巧妙的运用可视化工具对你的数据进行特征选择的分析。
0.导入数据
首先我们导入一些必要的数据处理工具包,并用pandas加载我们的数据,在这个例子中我们使用的数据是一个乳腺癌的诊断数据集,对于这种数据集,我们虽然没有医学方面的知识,但是可以通过数据分析得出一些结论
import numpy as np # linear algebra
import pandas as pd # data processing, CSV file I/O (e.g. pd.read_csv)
import seaborn as sns # data visualization library
import matplotlib.pyplot as plt
import time
from subprocess import check_output
data=pd.read_csv('data.csv')
1.数据分析
在特征选择和提取之前,我们先对数据进行基础的分析,先来看看我们的数据有哪些特征
data.head()

首先通过查看数据的特征,我们要注意到四个点:1. id 不是能够用于分类的数据 2.诊断结果(diagnosis)应该作为我们的label 3. 最后一列 Unamed 有NAN数据,所以这一列舍弃 4. 我们不知道其他特征都代表了什么,但是这并不影响我们对数据的分析。
下面我们将label与需要的特征分开:
#col存放特征的名字
col=data.columns
# y 存放标签
y=data.diagnosis
list=['Unnamed: 32','id','diagnosis']
x=data.drop(list,axis=1)
x.head()

我们用seaborn库画出对label内容的统计图
ax = sns.countplot(y,label="Count")

接下来我们来看特征,我们并不需要理解这些特征是什么意思,但是我们可以知道数据的方差(variance)和偏差(bias)以及数据中最大值和最小值是多少。这些类型的信息有助于了解数据的状态,好进行下一步的工作。 例如,area_mean特征的最大值为2500,smoothness_mean特征的最大值为0.16340。 因此,在可视化,特征选择,特征提取或分类之前,我们需要将这些数据进行标准化。
下面调用pandas中的describe函数计算出上述的基本信
x.describe()

2.数据可视化
为了使数据可视化,我们使用seaborn库,因为这个库里面有很多种图表类型,可以多方面的对数据进行分析
下面我们介绍一种小提琴图(violin plot),小提琴图可以很好的展现出在不同特征下标签的分布结果
#First ten features
data_dia = y
data = x
#将数据标准化
data_n_2 = (data - data.mean()) / (data.std())
#选取前十个数据
data = pd.concat([y,data_n_2.iloc[:,0:10]],axis=1)
data = pd.melt(data,id_vars="diagnosis",
var_name="features",
value_name='value')
plt.figure(figsize=(10,10))
sns.violinplot(x="features", y="value", hue="diagnosis", data=data,split=True, inner="quart")
plt.xticks(rotation=90)

通过上面的小提琴图,我们可以看出texture_mean 特征对应的标签分布良性和恶性分布的中心是区分开来的,而相比之下fractal_dimension_mean特征对应的分布则并不能很好的分开,所以texture_mean特征相对于fractal_dimension_mean更适合于用于标签的区分
下面是第二组特征的小提琴图
# Second ten features
data = pd.concat([y,data_n_2.iloc[:,10:20]],axis=1)
data = pd.melt(data,id_vars="diagnosis",
var_name="features",
value_name='value')
plt.figure(figsize=(10,10))
sns.violinplot(x="features", y="value", hue="diagnosis", data=data,split=True, inner="quart")
plt.xticks(rotation=90)

作为小提琴图的替代品,箱型图也可以得到同样的效果。箱型图还可以用来查看是否有离群的数据
plt.figure(figsize=(10,10))
sns.boxplot(x="features", y="value", hue="diagnosis", data=data)
plt.xticks(rotation=90)

箱型图的结构如下图所示
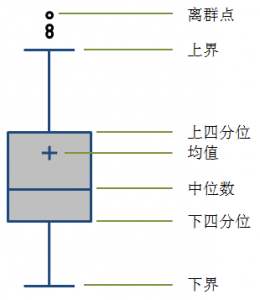
从上面的图可以得出一点,concavity_worst和 concave point_worst特征对应的标签分布看起来特别像,可以推断出他们很有可能是相关的,在大部分情况下,相关的特征可以舍弃掉其中一个。
为了更加深入的对比这两个变量,我们下面用一种叫joint plot的图。
sns.jointplot(x.loc[:,'concavity_worst'], x.loc[:,'concave points_worst'], kind="regg", color="#ce1414")

如上图所示,x轴和y轴分别是两个特征,joint plot画出所有数据点对应的点,从图中可以看出两个变量的pearsonr值(度量线性关系的指标)是很高的。
那么怎么对比多个变量的关系呢,我们可以使用 pair grid plot
sns.set(style="white")
df = x.loc[:,['radius_worst','perimeter_worst','area_worst']]
g = sns.PairGrid(df, diag_sharey=False)
g.map_lower(sns.kdeplot, cmap="Blues_d")
g.map_upper(plt.scatter)
g.map_diag(sns.kdeplot, lw=3)

Swarm plot 也是一种非常实用的数据分析的图表,下面分三张swarm plot来展示我们所有的数据
sns.set(style="whitegrid", palette="muted")
data_dia = y
data = x
data_n_2 = (data - data.mean()) / (data.std()) # standardization
data = pd.concat([y,data_n_2.iloc[:,0:10]],axis=1)
data = pd.melt(data,id_vars="diagnosis",
var_name="features",
value_name='value')
plt.figure(figsize=(10,10))
tic = time.time()
sns.swarmplot(x="features", y="value", hue="diagnosis", data=data)
plt.xticks(rotation=90)

data = pd.concat([y,data_n_2.iloc[:,10:20]],axis=1)
data = pd.melt(data,id_vars="diagnosis",
var_name="features",
value_name='value')
plt.figure(figsize=(10,10))
sns.swarmplot(x="features", y="value", hue="diagnosis", data=data)
plt.xticks(rotation=90)

data = pd.concat([y,data_n_2.iloc[:,20:31]],axis=1)
data = pd.melt(data,id_vars="diagnosis",
var_name="features",
value_name='value')
plt.figure(figsize=(10,10))
sns.swarmplot(x="features", y="value", hue="diagnosis", data=data)
toc = time.time()
plt.xticks(rotation=90)
print("swarm plot time: ", toc-tic ," s")

通过这上面三张图,我们可以将方差(variance)看的更加清楚,我们可以很直观的看出在第三张图中的perimeter_worst和area_worst两个特征在良性和恶性的分类上表现的最明显,而第二张图中的smoothness_se则非常不明显。
如果我们想看,各个特征之间的关系,我们可以使用热力图。
#correlation map
f,ax = plt.subplots(figsize=(18, 18))
sns.heatmap(x.corr(), annot=True, linewidths=.5, fmt= '.1f',ax=ax)

3.特征的选择和用随机森林预测结果
通过上面的图表,我们可以通过相关性选择我们预测所使用的特征了。通过上面的热力图,我们可以看出 特征radius_mean,perimeter_mean 和 area_mean 是相关的(图中相关性为1.0的两个变量),所以我们可以只选择area_mean这个变量,之所以选择area_mean特征,是因为在swarm plot中area_mean的数据在良性和恶性分类上更加清晰可辨。同样的我们选择出其余的相关性不大的特征,得到如下我们可以丢弃的特征,并丢弃,得到剩余我们用于预测的特征
drop_list1 = ['perimeter_mean','radius_mean','compactness_mean','concave points_mean','radius_se','perimeter_se','radius_worst','perimeter_worst','compactness_worst','concave points_worst','compactness_se','concave points_se','texture_worst','area_worst']
x_1 = x.drop(drop_list1,axis = 1 ) # do not modify x, we will use it later
x_1.head()

下面我们用这些筛选出来的特征用随机森林模型来测试下预测准确率
from sklearn.model_selection import train_test_split
from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import f1_score,confusion_matrix
from sklearn.metrics import accuracy_score
# split data train 70 % and test 30 %
x_train, x_test, y_train, y_test = train_test_split(x_1, y, test_size=0.3, random_state=42)
#random forest classifier with n_estimators=10 (default)
clf_rf = RandomForestClassifier(random_state=43)
clr_rf = clf_rf.fit(x_train,y_train)
ac = accuracy_score(y_test,clf_rf.predict(x_test))
print('Accuracy is: ',ac)
cm = confusion_matrix(y_test,clf_rf.predict(x_test))
sns.heatmap(cm,annot=True,fmt="d")

预测准确率超过了百分之96
4.总结
本文主要通过预测肿瘤是良性还是恶性的例子,介绍了小提琴图(violin plot),箱型图(box plot),joint plot,swarm plot和热力图(heatmap)的画法,并通过这些图进行特征的分析和筛选,最后进行预测的测试。筛选特征的方法有很多,而且用一些算法可能进行特征筛选可能更加高效,但是这些数据可视化的图标可以更好的用于展示和给人更加直观的理解
实现项目地址:Mo-人工智能开发教程,AI人工智能编程培训,培训平台/机构/课程,在线学习AI编程,一蓦官网.
关于我们
Mo(网址:https://momodel.cn)是一个支持 Python 的人工智能在线建模平台,能帮助你快速开发、训练并部署模型。
Mo 人工智能俱乐部 是由人工智能在线建模平台(网址:https://momodel.cn)的研发与产品团队发起、致力于降低人工智能开发与使用门槛的俱乐部。团队具备大数据处理分析、可视化与数据建模经验,已承担多领域智能项目,具备从底层到前端的全线设计开发能力。主要研究方向为大数据管理分析与人工智能技术,并以此来促进数据驱动的科学研究。
近期 Mo 也在持续进行机器学习相关的入门课程和论文分享活动,欢迎大家关注我们的公众号:MomodelAI获取新资讯!


