- 1运算放大器(运放)选型、参数分析以及应用OPA2350
- 2java 服务证书问题-生成jssecacerts CA证书
- 3相对全面的四足机器人驱动规划MATLAB和Simulink实现方式(足端摆线规划,Hopf-CPG,Kimura-CPG)_如何在simulink中获取足端轨迹
- 4Java -枚举的使用_java枚举的使用
- 5推荐系统笔记(一)_推荐系统技术架构
- 6使用XGBoost进行时间序列预测流程代码_xgboost 时间序列
- 7AWS-EKS 给其他IAM赋予集群管理权限
- 8Flink_CDC搭建及简单使用_flink cdc部署
- 9建议收藏!30套经典互联网思维研究模型_优化引导模型
- 10大数据分析-第八章 推荐系统_spricoder的博客
光谱预处理最简单实现方法--基于OpenSA光谱分析库
赞
踩
系列文章目录
“光晰本质,谱见不同”,光谱作为物质的指纹,被广泛应用于成分分析中。伴随微型光谱仪/光谱成像仪的发展与普及,基于光谱的分析技术将不只停留于工业和实验室,即将走入生活,实现万物感知,见微知著。本系列文章致力于光谱分析技术的科普和应用。
前言
典型的光谱分析模型(以近红外光谱作为示意,可见光、中远红外、荧光、拉曼、高光谱等分析流程亦相似)建立流程如下所示,在建立过程中,需要使用算法对训练样本进行选择,然后使用预处理算法对光谱进行预处理,或对光谱的特征进行提取,再构建校正模型实现定量分析,最后针对不同测量仪器或环境,进行模型转移或传递。因此训练样本的选择、光谱的预处理、波长筛选、校正模型、模型传递以及上述算法的参数都影响着模型的应用效果。
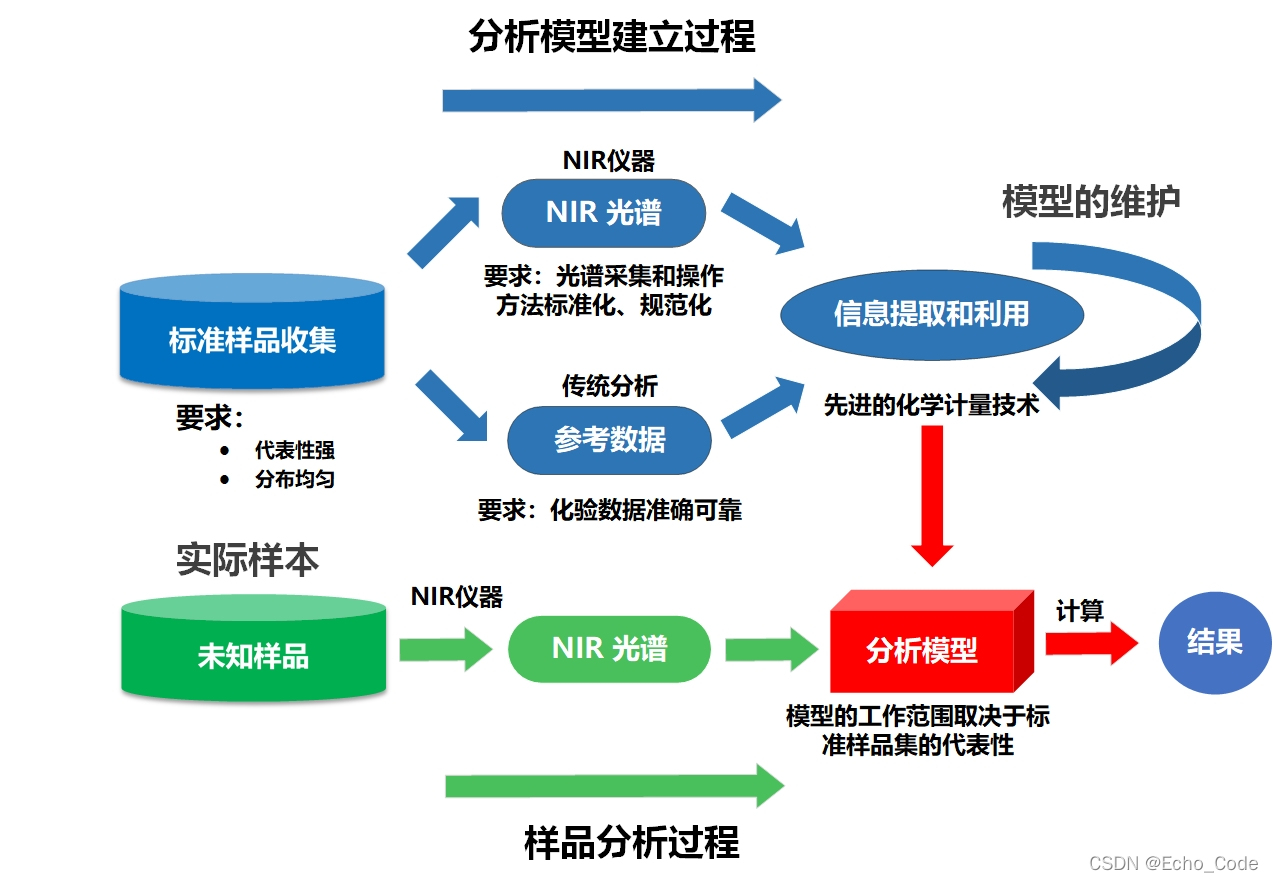
针对光谱分析流程所涉及的常见的训练样本的划分、光谱的预处理、波长筛选、校正模型算法建立了完整的算法库,名为OpenSA(OpenSpectrumAnalysis)。整套算法库的架构如下所示。
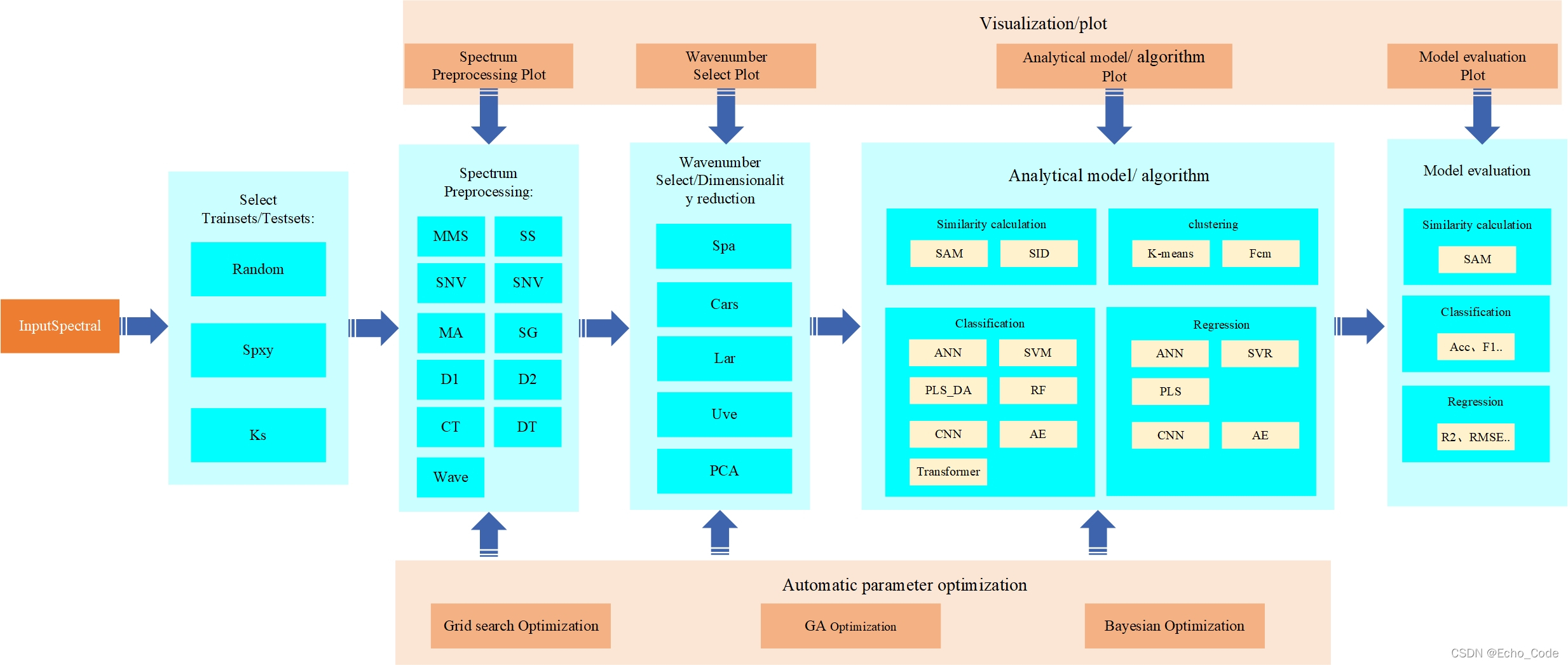
样本划分模块提供随机划分、SPXY划分、KS划分三种数据集划分方法,光谱预处理模块提供常见光谱预处理,波长筛选模块提供Spa、Cars、Lars、Uve、Pca等特征降维方法,分析模块由光谱相似度计算、聚类、分类(定性分析)、回归(定量分析)构建,光谱相似度子模块计算提供SAM、SID、MSSIM、MPSNR等相似计算方法,聚类子模块提供KMeans、FCM等聚类方法,分类子模块提供ANN、SVM、PLS_DA、RF等经典化学计量学方法,亦提供CNN、AE、Transformer等前沿深度学习方法,回归子模块提供ANN、SVR、PLS等经典化学计量学定量分析方法,亦提供CNN、AE、Transformer等前沿深度学习定量分析方法。模型评估模块提供常见的评价指标,用于模型评估。自动参数优化模块用于自动进行最佳的模型设置参数寻找,提供网格搜索、遗传算法、贝叶斯概率三种最优参数寻找方法。可视化模块提供全程的分析可视化,可为科研绘图,模型选择提供视觉信息。可通过几行代码快速实现完整的光谱分析及应用(注: 自动参数优化模块和可视化模块暂不开源,等毕业后再说)
本篇针对OpenSA的光谱预处理模块进行代码开源和使用示意。
一、光谱数据读入
提供两个开源数据作为实列,一个为公开定量分析数据集,一个为公开定性分析数据集,本章仅以公开定量分析数据集作为演示。
1.1 光谱数据读入
# 分别使用一个回归、一个分类的公开数据集做为example def LoadNirtest(type): if type == "Rgs": CDataPath1 = './/Data//Rgs//Cdata1.csv' VDataPath1 = './/Data//Rgs//Vdata1.csv' TDataPath1 = './/Data//Rgs//Tdata1.csv' Cdata1 = np.loadtxt(open(CDataPath1, 'rb'), dtype=np.float64, delimiter=',', skiprows=0) Vdata1 = np.loadtxt(open(VDataPath1, 'rb'), dtype=np.float64, delimiter=',', skiprows=0) Tdata1 = np.loadtxt(open(TDataPath1, 'rb'), dtype=np.float64, delimiter=',', skiprows=0) Nirdata1 = np.concatenate((Cdata1, Vdata1)) Nirdata = np.concatenate((Nirdata1, Tdata1)) data = Nirdata[:, :-4] label = Nirdata[:, -1] elif type == "Cls": path = './/Data//Cls//table.csv' Nirdata = np.loadtxt(open(path, 'rb'), dtype=np.float64, delimiter=',', skiprows=0) data = Nirdata[:, :-1] label = Nirdata[:, -1] return data, label
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
1.2 光谱可视化
#载入原始数据并可视化
data, label = LoadNirtest('Rgs')
plotspc(data, "raw specturm")
- 1
- 2
- 3
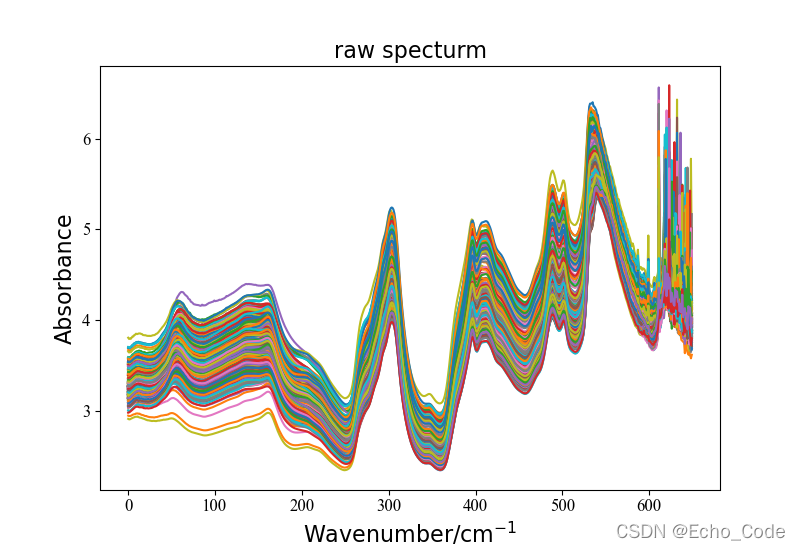
采用的开源光谱如图所示:
二、光谱预处理
2.1 光谱预处理模块
将常见的光谱进行了封装,使用者仅需要改变名字,即可选择对应的光谱分析,下面是光谱预处理模块的核心代码
""" -*- coding: utf-8 -*- @Time :2022/04/12 17:10 @Author : Pengyou FU @blogs : https://blog.csdn.net/Echo_Code?spm=1000.2115.3001.5343 @github : @WeChat : Fu_siry @License: """ import numpy as np from scipy import signal from sklearn.linear_model import LinearRegression from sklearn.preprocessing import MinMaxScaler, StandardScaler from copy import deepcopy import pandas as pd import pywt # 最大最小值归一化 def MMS(data): """ :param data: raw spectrum data, shape (n_samples, n_features) :return: data after MinMaxScaler :(n_samples, n_features) """ return MinMaxScaler().fit_transform(data) # 标准化 def SS(data): """ :param data: raw spectrum data, shape (n_samples, n_features) :return: data after StandScaler :(n_samples, n_features) """ return StandardScaler().fit_transform(data) # 均值中心化 def CT(data): """ :param data: raw spectrum data, shape (n_samples, n_features) :return: data after MeanScaler :(n_samples, n_features) """ for i in range(data.shape[0]): MEAN = np.mean(data[i]) data[i] = data[i] - MEAN return data # 标准正态变换 def SNV(data): """ :param data: raw spectrum data, shape (n_samples, n_features) :return: data after SNV :(n_samples, n_features) """ m = data.shape[0] n = data.shape[1] print(m, n) # # 求标准差 data_std = np.std(data, axis=1) # 每条光谱的标准差 # 求平均值 data_average = np.mean(data, axis=1) # 每条光谱的平均值 # SNV计算 data_snv = [[((data[i][j] - data_average[i]) / data_std[i]) for j in range(n)] for i in range(m)] return data_snv # 移动平均平滑 def MA(data, WSZ=11): """ :param data: raw spectrum data, shape (n_samples, n_features) :param WSZ: int :return: data after MA :(n_samples, n_features) """ for i in range(data.shape[0]): out0 = np.convolve(data[i], np.ones(WSZ, dtype=int), 'valid') / WSZ # WSZ是窗口宽度,是奇数 r = np.arange(1, WSZ - 1, 2) start = np.cumsum(data[i, :WSZ - 1])[::2] / r stop = (np.cumsum(data[i, :-WSZ:-1])[::2] / r)[::-1] data[i] = np.concatenate((start, out0, stop)) return data # Savitzky-Golay平滑滤波 def SG(data, w=11, p=2): """ :param data: raw spectrum data, shape (n_samples, n_features) :param w: int :param p: int :return: data after SG :(n_samples, n_features) """ return signal.savgol_filter(data, w, p) # 一阶导数 def D1(data): """ :param data: raw spectrum data, shape (n_samples, n_features) :return: data after First derivative :(n_samples, n_features) """ n, p = data.shape Di = np.ones((n, p - 1)) for i in range(n): Di[i] = np.diff(data[i]) return Di # 二阶导数 def D2(data): """ :param data: raw spectrum data, shape (n_samples, n_features) :return: data after second derivative :(n_samples, n_features) """ data = deepcopy(data) if isinstance(data, pd.DataFrame): data = data.values temp2 = (pd.DataFrame(data)).diff(axis=1) temp3 = np.delete(temp2.values, 0, axis=1) temp4 = (pd.DataFrame(temp3)).diff(axis=1) spec_D2 = np.delete(temp4.values, 0, axis=1) return spec_D2 # 趋势校正(DT) def DT(data): """ :param data: raw spectrum data, shape (n_samples, n_features) :return: data after DT :(n_samples, n_features) """ lenth = data.shape[1] x = np.asarray(range(lenth), dtype=np.float32) out = np.array(data) l = LinearRegression() for i in range(out.shape[0]): l.fit(x.reshape(-1, 1), out[i].reshape(-1, 1)) k = l.coef_ b = l.intercept_ for j in range(out.shape[1]): out[i][j] = out[i][j] - (j * k + b) return out # 多元散射校正 def MSC(data): """ :param data: raw spectrum data, shape (n_samples, n_features) :return: data after MSC :(n_samples, n_features) """ n, p = data.shape msc = np.ones((n, p)) for j in range(n): mean = np.mean(data, axis=0) # 线性拟合 for i in range(n): y = data[i, :] l = LinearRegression() l.fit(mean.reshape(-1, 1), y.reshape(-1, 1)) k = l.coef_ b = l.intercept_ msc[i, :] = (y - b) / k return msc # 小波变换 def wave(data): """ :param data: raw spectrum data, shape (n_samples, n_features) :return: data after wave :(n_samples, n_features) """ data = deepcopy(data) if isinstance(data, pd.DataFrame): data = data.values def wave_(data): w = pywt.Wavelet('db8') # 选用Daubechies8小波 maxlev = pywt.dwt_max_level(len(data), w.dec_len) coeffs = pywt.wavedec(data, 'db8', level=maxlev) threshold = 0.04 for i in range(1, len(coeffs)): coeffs[i] = pywt.threshold(coeffs[i], threshold * max(coeffs[i])) datarec = pywt.waverec(coeffs, 'db8') return datarec tmp = None for i in range(data.shape[0]): if (i == 0): tmp = wave_(data[i]) else: tmp = np.vstack((tmp, wave_(data[i]))) return tmp def Preprocessing(method, data): if method == "None": data = data elif method == 'MMS': data = MMS(data) elif method == 'SS': data = SS(data) elif method == 'CT': data = CT(data) elif method == 'SNV': data = SNV(data) elif method == 'MA': data = MA(data) elif method == 'SG': data = SG(data) elif method == 'MSC': data = MSC(data) elif method == 'D1': data = D1(data) elif method == 'D2': data = D2(data) elif method == 'DT': data = DT(data) elif method == 'WVAE': data = wave(data) else: print("no this method of preprocessing!") return data
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
- 54
- 55
- 56
- 57
- 58
- 59
- 60
- 61
- 62
- 63
- 64
- 65
- 66
- 67
- 68
- 69
- 70
- 71
- 72
- 73
- 74
- 75
- 76
- 77
- 78
- 79
- 80
- 81
- 82
- 83
- 84
- 85
- 86
- 87
- 88
- 89
- 90
- 91
- 92
- 93
- 94
- 95
- 96
- 97
- 98
- 99
- 100
- 101
- 102
- 103
- 104
- 105
- 106
- 107
- 108
- 109
- 110
- 111
- 112
- 113
- 114
- 115
- 116
- 117
- 118
- 119
- 120
- 121
- 122
- 123
- 124
- 125
- 126
- 127
- 128
- 129
- 130
- 131
- 132
- 133
- 134
- 135
- 136
- 137
- 138
- 139
- 140
- 141
- 142
- 143
- 144
- 145
- 146
- 147
- 148
- 149
- 150
- 151
- 152
- 153
- 154
- 155
- 156
- 157
- 158
- 159
- 160
- 161
- 162
- 163
- 164
- 165
- 166
- 167
- 168
- 169
- 170
- 171
- 172
- 173
- 174
- 175
- 176
- 177
- 178
- 179
- 180
- 181
- 182
- 183
- 184
- 185
- 186
- 187
- 188
- 189
- 190
- 191
- 192
- 193
- 194
- 195
- 196
- 197
- 198
- 199
- 200
- 201
- 202
- 203
- 204
- 205
- 206
- 207
- 208
- 209
- 210
- 211
- 212
- 213
- 214
- 215
- 216
- 217
- 218
- 219
- 220
- 221
- 222
- 223
- 224
- 225
- 226
- 227
2 .2 光谱预处理的使用
在example.py文件中,提供了光谱预处理模块的使用方法,具体如下,仅需要两行代码即可实现所有常见的光谱预处理。
示意1:利用OpenSA实现MSC多元散射校正
#载入原始数据并可视化
data, label = LoadNirtest('Rgs')
plotspc(data, "raw specturm")
#光谱预处理并可视化
method = "MSC"
Preprocessingdata = Preprocessing(method, data)
plotspc(Preprocessingdata, method)
- 1
- 2
- 3
- 4
- 5
- 6
- 7
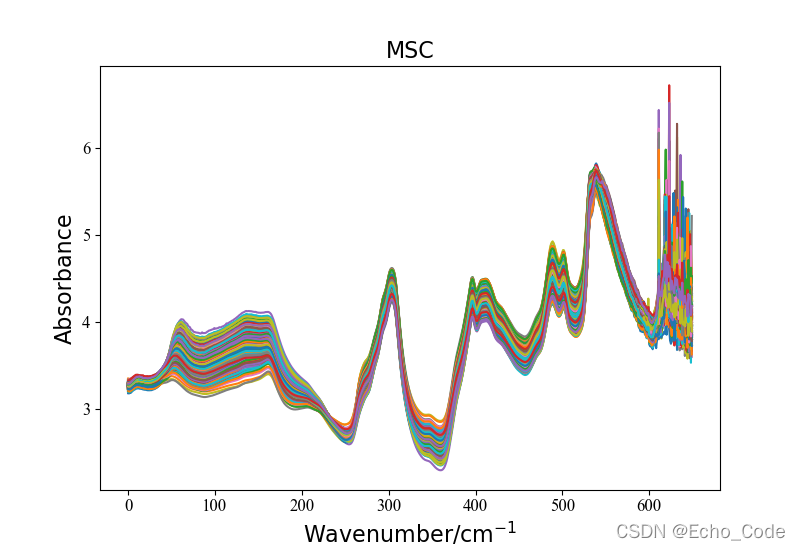
预处理后的光谱数据如图所示:
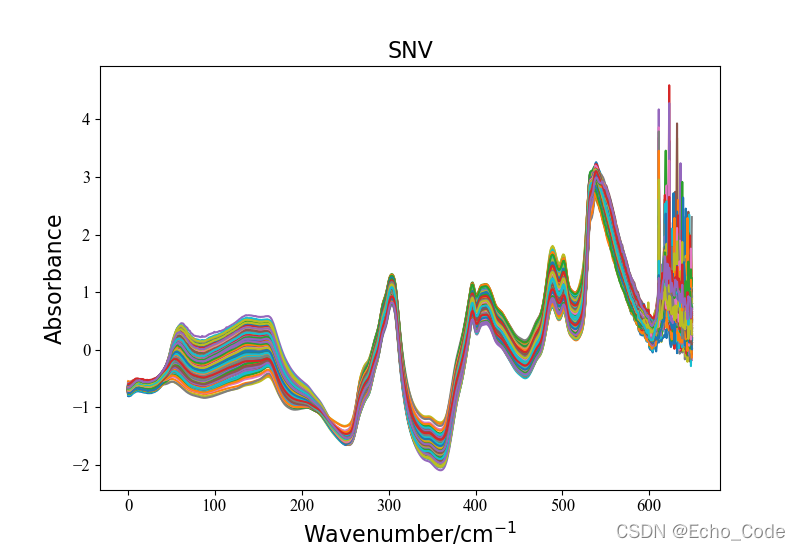
示意2:利用OpenSA实现SNV预处理
#载入原始数据并可视化
data, label = LoadNirtest('Rgs')
plotspc(data, "raw specturm")
#光谱预处理并可视化
method = "SNV"
Preprocessingdata = Preprocessing(method, data)
plotspc(Preprocessingdata, method)
- 1
- 2
- 3
- 4
- 5
- 6
- 7
预处理后的光谱数据如图所示:
总结
利用OpenSA可以非常简单的实现对光谱的预处理,完整代码可从获得GitHub仓库 如果对您有用,请点赞!
代码现仅供学术使用,若对您的学术研究有帮助,请引用本人的论文,同时,未经许可不得用于商业化应用,欢迎大家继续补充OpenSA中所涉及到的算法,如有问题



