- 1大数据之DataX介绍、主要特点与工作流程_datax任务
- 2VM虚拟机的安装与配置及操作系统的安装
- 3【远程管理】Mac下的putty远程管理软件_苹果笔记本怎么用putty
- 4git clone下载文件到指定目录_git clone 如何将repo直接下载到某个文件夹
- 5云账户实际业务SQL对比测试ClickHouse、TiDB和StarRocks_starrocks tidb
- 6Spark(42) -- SparkStreaming -- reduceByKeyAndWindow 函数详解_spark streaming reducebykeyandwindow计算top10
- 7python开发api_Python-接口开发入门
- 8百度大模型安全荣获2024世界智能产业博览会“Find智能科技创新应用典型案例”
- 9深度学习目标检测算法之RetinaNet算法
- 10yolov9 瑞芯微 rknn 部署 C++代码_yolov9 转rknn
无监督学习:从理论到实践的全面指南
赞
踩
本文深入讲解了无监督学习中的K-means、层次聚类、密度聚类、PCA、t-SNE和自编码器算法,涵盖其原理、数学基础、实现步骤及应用实例,并提供了详细的代码示例。
关注作者,复旦AI博士,分享AI领域全维度知识与研究。拥有10+年AI领域研究经验、复旦机器人智能实验室成员,国家级大学生赛事评审专家,发表多篇SCI核心期刊学术论文,上亿营收AI产品研发负责人。
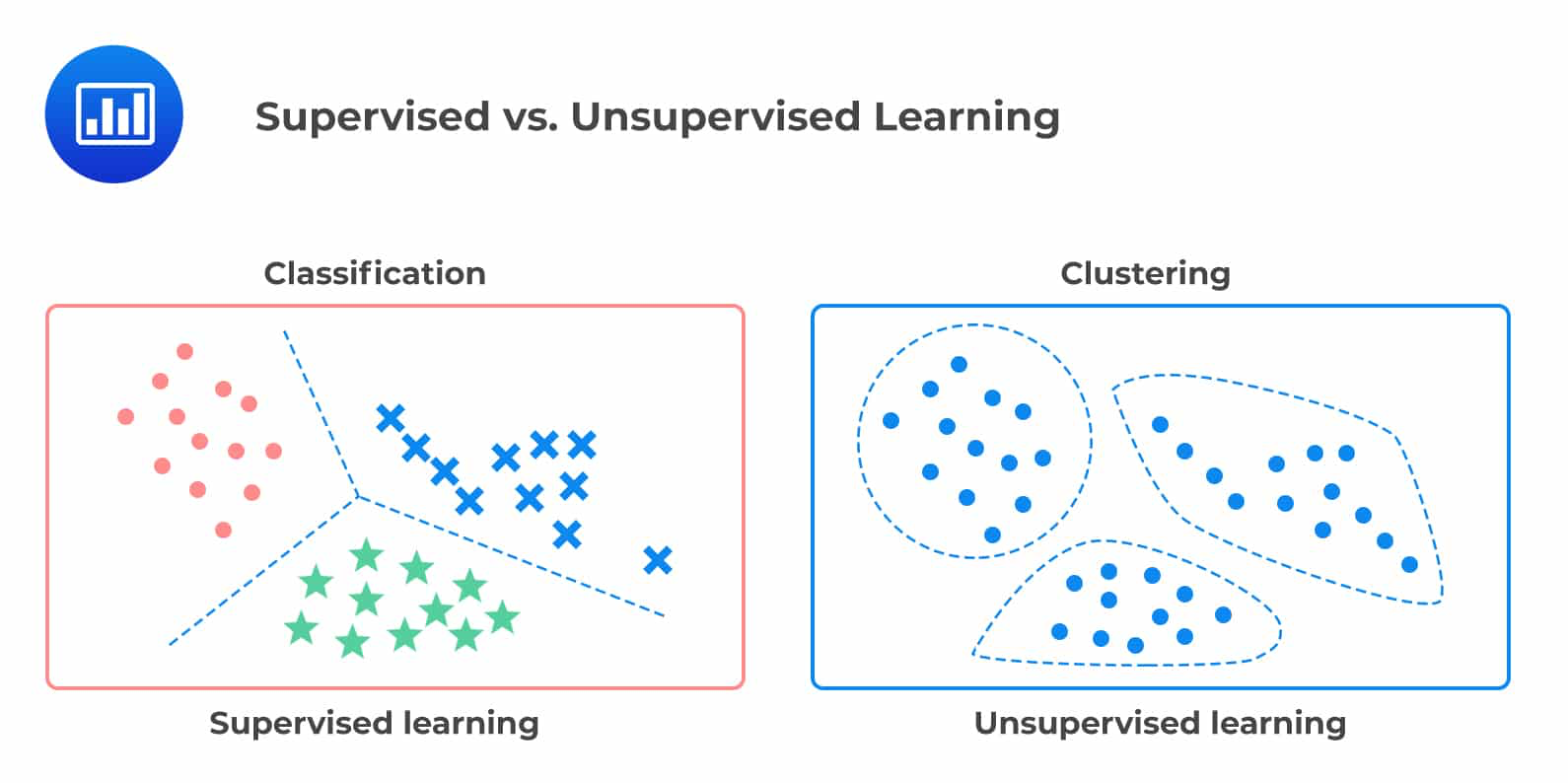
一、无监督学习概述
1.1 无监督学习的定义与背景
无监督学习(Unsupervised Learning)是一类机器学习任务,其中算法在没有标签的情况下,从未标记的数据中学习模式和结构。与有监督学习不同,无监督学习不依赖于预定义的输出,而是从数据本身提取信息,用于发现数据的内在规律和特征。
背景
无监督学习的应用背景非常广泛,尤其是在数据标注成本高昂或数据标签缺失的领域中。例如,生物信息学中的基因表达分析、天文学中的星系分类、社交网络分析中的社区检测等。随着数据规模的不断增长,无监督学习的重要性也日益凸显。
1.2 无监督学习的应用场景
无监督学习在许多领域中都有广泛的应用。以下是一些典型的应用场景:
数据聚类
数据聚类是无监督学习的一种主要任务,旨在将相似的数据点分组。例如,市场营销中的客户细分、图像处理中的图像分割、文本分析中的文档聚类等。
降维
降维技术用于减少数据的维度,以便更好地可视化和分析数据。例如,主成分分析(PCA)和t-SNE常用于高维数据的降维和可视化,帮助研究人员发现数据中的潜在结构和模式。
异常检测
无监督学习还用于检测数据中的异常点或异常模式。例如,在网络安全中检测异常流量,在金融行业中检测异常交易行为等。
1.3 与有监督学习的区别
数据依赖性
有监督学习依赖于大量标记数据进行训练,模型通过已知的输入-输出对进行学习。无监督学习则不需要标记数据,完全依赖数据的内在结构进行学习。
目标导向
有监督学习的目标是预测或分类,例如图像分类、语音识别等。无监督学习的目标是发现数据的模式和结构,例如聚类、降维等。
复杂性与挑战
无监督学习的挑战在于其不确定性。由于缺乏标签,评估无监督学习模型的效果往往更加复杂,需要依赖于外部指标或人为判断。
1.4 主要技术方法
聚类算法
聚类算法是无监督学习中最常见的技术之一。其目的是将相似的数据点分组,使同一组内的数据点尽可能相似,而不同组之间的数据点尽可能不同。常见的聚类算法包括K-means、层次聚类和DBSCAN。
降维技术
降维技术用于减少数据的维度,同时保留尽可能多的有用信息。这对于高维数据的处理和可视化尤为重要。主成分分析(PCA)和t-SNE是两种常见的降维技术。
生成模型
生成模型如生成对抗网络(GAN)和变分自编码器(VAE)近年来在无监督学习中取得了显著的进展。这些模型通过学习数据的分布来生成与原始数据相似的新数据,广泛应用于图像生成、数据增强等领域。
二、算法精讲
2.1 K-means算法精讲
K-means算法是一种经典且广泛应用的聚类算法,旨在将数据集分割成K个簇,使得同一簇内的数据点尽可能相似,而不同簇之间的数据点差异尽可能大。本文将详细介绍K-means算法的原理、数学基础、优化方法,并通过代码示例展示其具体实现。
2.1.1 算法原理
K-means算法通过迭代优化以下两个步骤实现数据的聚类:
- 初始化:随机选择K个数据点作为初始的簇中心(centroids)。
- 迭代优化:
- 分配步骤(Assignment Step):将每个数据点分配到距离其最近的簇中心所属的簇。
- 更新步骤(Update Step):重新计算每个簇的中心,即每个簇内所有数据点的平均值,作为新的簇中心。
该过程不断重复,直到簇中心不再发生显著变化或达到预设的迭代次数。
2.1.2 数学基础

2.1.3 优化方法
尽管K-means算法简单且高效,但其结果依赖于初始簇中心的选择,容易陷入局部最优解。以下是几种常见的优化方法:
- 多次运行K-means:通过多次运行K-means算法,每次随机初始化簇中心,然后选择最优的结果。
- K-means++初始化:一种改进的初始化方法,选择初始簇中心时更加注重分布,能够显著提升算法的聚类效果。
- Mini-batch K-means:在大数据集上使用小批量数据进行更新,以提高计算效率。
2.1.4 代码示例
以下是使用Python和SciPy库实现K-means算法的示例代码:
import numpy as np import matplotlib.pyplot as plt from sklearn.datasets import make_blobs from scipy.spatial.distance import cdist def initialize_centroids(X, k): """随机初始化k个簇中心""" indices = np.random.choice(X.shape[0], k, replace=False) return X[indices] def assign_clusters(X, centroids): """分配数据点到最近的簇中心""" distances = cdist(X, centroids, 'euclidean') return np.argmin(distances, axis=1) def update_centroids(X, labels, k): """更新簇中心为簇内所有数据点的平均值""" new_centroids = np.zeros((k, X.shape[1])) for i in range(k): points = X[labels == i] new_centroids[i] = points.mean(axis=0) return new_centroids def kmeans(X, k, max_iters=100, tol=1e-4): """K-means算法实现""" centroids = initialize_centroids(X, k) for _ in range(max_iters): labels = assign_clusters(X, centroids) new_centroids = update_centroids(X, labels, k) if np.all(np.abs(new_centroids - centroids) < tol): break centroids = new_centroids return labels, centroids # 生成样本数据 X, y = make_blobs(n_samples=300, centers=4, random_state=42) # 执行K-means算法 k = 4 labels, centroids = kmeans(X, k) # 可视化聚类结果 plt.scatter(X[:, 0], X[:, 1], c=labels, cmap='viridis', marker='o') plt.scatter(centroids[:, 0], centroids[:, 1], s=300, c='red', marker='x') plt.title("K-means Clustering") plt.xlabel("Feature 1") plt.ylabel("Feature 2") plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
代码解析
- 初始化簇中心:
initialize_centroids函数通过随机选择数据点作为初始簇中心。 - 分配数据点:
assign_clusters函数计算每个数据点到所有簇中心的欧几里得距离,并将数据点分配到最近的簇。 - 更新簇中心:
update_centroids函数计算每个簇的新中心,即簇内所有数据点的平均值。 - K-means主函数:
kmeans函数在给定的迭代次数和收敛阈值下执行K-means算法,返回最终的簇标签和簇中心。
2.2 层次聚类算法精讲
层次聚类(Hierarchical Clustering)是一种无监督学习方法,通过建立层次结构将数据集进行聚类。与K-means等平面聚类方法不同,层次聚类创建一个树状结构(或称为树状图),能够展示数据点之间的嵌套关系。本文将详细介绍层次聚类的基本原理、类型、计算方法及其应用,并通过代码示例展示具体实现。
2.2.1 算法原理
层次聚类分为两种主要方法:凝聚(自下而上)和分裂(自上而下)。
凝聚层次聚类(Agglomerative Clustering)
- 初始化:将每个数据点视为一个独立的簇。
- 迭代合并:在每一步中,找到距离最近的两个簇并将其合并,重复这一过程直到所有数据点被合并到一个簇中或达到预设的簇数。
分裂层次聚类(Divisive Clustering)
- 初始化:将所有数据点视为一个单一的簇。
- 迭代分裂:在每一步中,选择一个簇并将其拆分为两个子簇,重复这一过程直到每个数据点成为一个独立的簇或达到预设的簇数。
2.2.2 距离度量
层次聚类中,定义簇之间的距离是关键步骤。常用的距离度量方法包括:
- 最短距离法(Single Linkage):两个簇中最近点之间的距离。
- 最长距离法(Complete Linkage):两个簇中最远点之间的距离。
- 平均距离法(Average Linkage):两个簇中所有点对之间的平均距离。
- 质心法(Centroid Linkage):两个簇的质心之间的距离。
2.2.3 数学基础
层次聚类算法的核心在于不断计算和更新簇间距离,具体步骤如下:
- 距离矩阵初始化:计算所有数据点对之间的距离,形成距离矩阵。
- 簇合并:根据选定的距离度量方法,找到距离最近的两个簇并合并。
- 距离矩阵更新:合并后重新计算新的簇与其他簇之间的距离,更新距离矩阵。
2.2.4 代码示例
以下是使用Python和SciPy库实现凝聚层次聚类的示例代码:
import numpy as np import matplotlib.pyplot as plt from scipy.cluster.hierarchy import dendrogram, linkage, fcluster from sklearn.datasets import make_blobs # 生成样本数据 X, y = make_blobs(n_samples=300, centers=4, random_state=42) # 计算层次聚类的链接矩阵 Z = linkage(X, method='ward') # 绘制树状图 plt.figure(figsize=(10, 7)) dendrogram(Z) plt.title('Hierarchical Clustering Dendrogram') plt.xlabel('Sample index') plt.ylabel('Distance') plt.show() # 根据距离阈值提取簇 max_d = 50 # 距离阈值 clusters = fcluster(Z, max_d, criterion='distance') # 可视化聚类结果 plt.scatter(X[:, 0], X[:, 1], c=clusters, cmap='viridis', marker='o') plt.title('Agglomerative Clustering') plt.xlabel('Feature 1') plt.ylabel('Feature 2') plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
代码解析
- 生成样本数据:使用
make_blobs生成一个包含四个簇的样本数据集。 - 计算链接矩阵:使用
linkage函数计算层次聚类的链接矩阵,方法选择ward,即最小方差法。 - 绘制树状图:使用
dendrogram函数绘制层次聚类的树状图,展示聚类的层次结构。 - 提取簇:使用
fcluster函数根据距离阈值提取簇,max_d为距离阈值。 - 可视化聚类结果:根据提取的簇标签,绘制聚类结果的散点图。
2.2.5 层次聚类的优缺点
优点
- 无需预定义簇数:层次聚类不需要预先指定簇的数量,这对于数据的探索性分析非常有用。
- 层次结构:层次聚类可以生成树状图,展示数据点之间的层次关系,有助于理解数据的内在结构。
缺点
- 计算复杂度高:层次聚类的计算复杂度较高,特别是对于大规模数据集,计算和内存开销都非常大。
- 敏感性强:层次聚类对噪声和离群点非常敏感,这可能导致不准确的聚类结果。
2.2.6 应用实例
层次聚类广泛应用于各种领域,例如:
- 生物信息学:基因表达数据的聚类分析,构建基因共表达网络。
- 市场营销:客户细分,根据购买行为和偏好进行市场细分。
- 文档聚类:基于内容的文档聚类,组织和分类大量文本数据。
2.3 密度聚类算法精讲
密度聚类算法是一类基于数据点密度的无监督学习方法,能够有效处理具有复杂形状和噪声的数据集。最著名的密度聚类算法是DBSCAN(Density-Based Spatial Clustering of Applications with Noise)。本文将详细介绍DBSCAN算法的原理、数学基础、参数选择、优缺点,并通过代码示例展示其具体实现。
2.3.1 算法原理
DBSCAN算法通过以下步骤实现聚类:
- 核心点识别:对每个数据点,计算其ε-邻域内的数据点数目。如果数据点的邻域内数据点数目超过最小点数(MinPts)阈值,则该数据点为核心点。
- 簇形成:从核心点出发,将其邻域内的所有点(包括其他核心点和边界点)加入同一簇。迭代进行,直到所有核心点都被处理。
- 噪声识别:未能被任何簇包含的点被标记为噪声。
2.3.2 数学基础
DBSCAN算法依赖于两个重要参数:
- ε(Epsilon):定义数据点的邻域半径。
- MinPts:定义一个点成为核心点所需的最小邻域点数。
核心点、边界点和噪声点
- 核心点:邻域内数据点数目大于等于MinPts。
- 边界点:邻域内数据点数目小于MinPts,但在核心点的邻域内。
- 噪声点:既不是核心点也不是边界点的点。
2.3.3 算法步骤
- 初始化:遍历所有数据点。
- 扩展簇:
- 如果数据点为核心点,创建新簇,并将其邻域内的所有点添加到簇中。
- 对于每个邻域内的核心点,继续扩展簇,直到无法再扩展。
- 处理完所有点:继续处理其他点,直到所有点都被访问。
2.3.4 参数选择
选择合适的ε和MinPts参数对DBSCAN的效果至关重要:
- ε的选择:通过k-距离图选择ε值。绘制数据集中每个点到其k-最近邻的距离,寻找“肘部”点对应的距离作为ε值。
- MinPts的选择:通常选择至少为数据集维度的2倍,即MinPts ≥ 2 * Dim。
2.3.5 优缺点
优点
- 识别任意形状簇:DBSCAN能够发现任意形状的簇,不受簇形状的限制。
- 处理噪声:DBSCAN可以有效地识别和处理噪声数据点。
- 无需预定义簇数:DBSCAN不需要预定义簇的数量,这对于未知簇数的数据集非常有用。
缺点
- 参数敏感性:DBSCAN对参数ε和MinPts较为敏感,选择不当会影响聚类效果。
- 高维数据性能差:在高维数据中,DBSCAN的效果可能较差,需要进行降维处理。
2.3.6 代码示例
以下是使用Python和Scikit-learn库实现DBSCAN算法的示例代码:
import numpy as np import matplotlib.pyplot as plt from sklearn.datasets import make_blobs from sklearn.cluster import DBSCAN from sklearn.preprocessing import StandardScaler # 生成样本数据 X, y = make_blobs(n_samples=300, centers=4, random_state=42) # 数据标准化处理 X = StandardScaler().fit_transform(X) # DBSCAN聚类 db = DBSCAN(eps=0.3, min_samples=10).fit(X) labels = db.labels_ # 获取核心点和噪声点 core_samples_mask = np.zeros_like(labels, dtype=bool) core_samples_mask[db.core_sample_indices_] = True n_clusters_ = len(set(labels)) - (1 if -1 in labels else 0) n_noise_ = list(labels).count(-1) # 可视化聚类结果 unique_labels = set(labels) colors = [plt.cm.Spectral(each) for each in np.linspace(0, 1, len(unique_labels))] plt.figure(figsize=(10, 7)) for k, col in zip(unique_labels, colors): if k == -1: # 黑色用于噪声点 col = [0, 0, 0, 1] class_member_mask = (labels == k) xy = X[class_member_mask & core_samples_mask] plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=tuple(col), markeredgecolor='k', markersize=14) xy = X[class_member_mask & ~core_samples_mask] plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=tuple(col), markeredgecolor='k', markersize=6) plt.title(f'Estimated number of clusters: {n_clusters_}\n' f'Number of noise points: {n_noise_}') plt.xlabel('Feature 1') plt.ylabel('Feature 2') plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
代码解析
- 生成样本数据:使用
make_blobs生成一个包含四个簇的样本数据集。 - 数据标准化:使用
StandardScaler对数据进行标准化处理,确保不同特征在相同尺度上。 - DBSCAN聚类:使用
DBSCAN函数进行聚类,设置参数eps和min_samples。 - 获取核心点和噪声点:通过
core_sample_indices_获取核心点,并统计簇的数量和噪声点的数量。 - 可视化聚类结果:根据簇标签绘制不同颜色的散点图,噪声点用黑色表示。
2.3.7 应用实例
DBSCAN广泛应用于各种领域,例如:
- 地理信息系统:地理空间数据的聚类分析,识别城市区域或地貌特征。
- 图像处理:图像中的目标检测和分割。
- 金融分析:异常交易行为检测,识别潜在的欺诈行为。
2.4 主成分分析算法精讲
主成分分析(Principal Component Analysis,PCA)是一种经典的降维技术,通过线性变换将高维数据映射到低维空间,同时尽可能保留原始数据中的主要信息。PCA在数据预处理、特征提取、模式识别和数据可视化等领域具有广泛应用。本文将详细介绍PCA的原理、数学基础、实现步骤,并通过代码示例展示其具体实现。
2.4.1 算法原理
PCA通过寻找数据的主成分,将数据投影到这些主成分构成的子空间中。主成分是数据在变换后的坐标系中的新基向量,这些基向量是按数据方差大小排序的。具体步骤如下:
- 数据标准化:将数据中心化,使其均值为零。
- 协方差矩阵计算:计算数据的协方差矩阵。
- 特征值分解:对协方差矩阵进行特征值分解,得到特征值和特征向量。
- 选择主成分:选择前k个特征值对应的特征向量作为主成分。
- 数据变换:将原始数据投影到选定的主成分上,得到降维后的数据。
2.4.2 数学基础

2.4.3 实现步骤
以下是PCA算法的具体实现步骤:
- 数据准备:加载并标准化数据。
- 计算协方差矩阵:根据标准化数据计算协方差矩阵。
- 特征值分解:对协方差矩阵进行特征值分解。
- 选择主成分:选择前k个特征值对应的特征向量。
- 数据变换:将原始数据投影到选定的主成分上。
2.4.4 代码示例
以下是使用Python和Scikit-learn库实现PCA的示例代码:
import numpy as np import matplotlib.pyplot as plt from sklearn.decomposition import PCA from sklearn.preprocessing import StandardScaler from sklearn.datasets import load_iris # 加载示例数据集 data = load_iris() X = data.data y = data.target # 数据标准化 scaler = StandardScaler() X_std = scaler.fit_transform(X) # PCA降维,选择前2个主成分 pca = PCA(n_components=2) X_pca = pca.fit_transform(X_std) # 可视化降维后的数据 plt.figure(figsize=(10, 7)) plt.scatter(X_pca[:, 0], X_pca[:, 1], c=y, cmap='viridis', edgecolor='k', s=150) plt.title('PCA of Iris Dataset') plt.xlabel('Principal Component 1') plt.ylabel('Principal Component 2') plt.colorbar() plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
代码解析
- 加载数据:使用
load_iris函数加载鸢尾花数据集。 - 数据标准化:使用
StandardScaler对数据进行标准化处理,确保每个特征具有零均值和单位方差。 - PCA降维:使用
PCA类对标准化后的数据进行降维,选择前两个主成分。 - 可视化:绘制降维后的数据散点图,展示不同类别的数据点在主成分空间的分布。
2.4.5 优缺点
优点
- 降维效果显著:PCA能够有效减少数据的维度,保留主要信息,便于后续分析和处理。
- 计算高效:PCA的计算复杂度较低,适用于大规模数据集。
- 无参数要求:PCA不需要设置超参数,使用方便。
缺点
- 线性假设:PCA假设数据与主成分之间的关系是线性的,对于非线性数据表现不佳。
- 解释性差:主成分是线性组合,难以直接解释每个主成分的具体含义。
- 对噪声敏感:PCA对噪声数据较为敏感,可能受到噪声的干扰。
2.4.6 应用实例
PCA广泛应用于各种领域,例如:
- 图像处理:图像压缩与特征提取。
- 金融分析:股票收益率数据的降维与风险管理。
- 生物信息学:基因表达数据的降维与聚类分析。
2.5 t-SNE算法精讲
t-SNE(t-Distributed Stochastic Neighbor Embedding)是一种非线性降维技术,尤其擅长在低维空间中保持高维数据的局部结构。t-SNE广泛应用于数据可视化,特别是在处理高维数据时,能够揭示数据的内在结构和模式。本文将详细介绍t-SNE的原理、数学基础、实现步骤,并通过代码示例展示其具体实现。
2.5.1 算法原理
t-SNE通过构建高维数据点之间的相似度,然后将这些相似度映射到低维空间中,使得相似的数据点在低维空间中尽可能靠近。其核心思想包括以下几个步骤:
- 高维空间中的相似度计算:在高维空间中,t-SNE使用高斯分布计算数据点之间的相似度。
- 低维空间中的相似度计算:在低维空间中,t-SNE使用t分布计算数据点之间的相似度。
- 相似度匹配:通过最小化两种空间中相似度分布之间的Kullback-Leibler散度(KL散度),将高维数据映射到低维空间。
2.5.2 数学基础

2.5.3 实现步骤
- 数据准备:加载并标准化数据。
- 相似度计算:在高维空间中计算数据点之间的相似度。
- 初始嵌入:在低维空间中初始化数据点的位置。
- 优化:通过梯度下降法最小化KL散度,更新低维嵌入。
- 可视化:展示降维后的数据。
2.5.4 代码示例
以下是使用Python和Scikit-learn库实现t-SNE的示例代码:
import numpy as np import matplotlib.pyplot as plt from sklearn.manifold import TSNE from sklearn.preprocessing import StandardScaler from sklearn.datasets import load_digits # 加载示例数据集 data = load_digits() X = data.data y = data.target # 数据标准化 scaler = StandardScaler() X_std = scaler.fit_transform(X) # t-SNE降维 tsne = TSNE(n_components=2, perplexity=30, n_iter=300) X_tsne = tsne.fit_transform(X_std) # 可视化降维后的数据 plt.figure(figsize=(10, 7)) scatter = plt.scatter(X_tsne[:, 0], X_tsne[:, 1], c=y, cmap='viridis', edgecolor='k', s=100) plt.title('t-SNE of Digits Dataset') plt.xlabel('t-SNE Component 1') plt.ylabel('t-SNE Component 2') plt.colorbar(scatter) plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
代码解析
- 加载数据:使用
load_digits函数加载手写数字数据集。 - 数据标准化:使用
StandardScaler对数据进行标准化处理,确保每个特征具有零均值和单位方差。 - t-SNE降维:使用
t-SNE类对标准化后的数据进行降维,设置参数n_components为2(即二维空间),perplexity为30,n_iter为300。 - 可视化:绘制降维后的数据散点图,展示不同类别的数据点在t-SNE空间的分布。
2.5.5 优缺点
优点
- 保持局部结构:t-SNE能够有效保持高维数据的局部结构,使得相似的数据点在低维空间中靠近。
- 适合高维数据:t-SNE在处理高维数据时表现出色,能够揭示数据的内在模式和结构。
缺点
- 计算复杂度高:t-SNE的计算复杂度较高,特别是在大规模数据集上,计算时间较长。
- 参数敏感性:t-SNE对参数(如perplexity)较为敏感,需要仔细调整以获得最佳效果。
- 难以解释:t-SNE的结果难以解释,不适用于所有降维任务。
2.5.6 应用实例
t-SNE广泛应用于各种领域,例如:
- 图像处理:高维图像特征的可视化分析。
- 自然语言处理:词向量和句向量的可视化。
- 生物信息学:基因表达数据的可视化。
2.6 自编码器算法精讲
自编码器(Autoencoder)是一类用于无监督学习的神经网络,主要用于降维、特征提取和数据生成。自编码器通过将输入数据编码为低维表示,然后再解码为原始数据,来学习数据的有效表示。本文将详细介绍自编码器的原理、数学基础、常见变种及其实现步骤,并通过代码示例展示其具体实现。
2.6.1 算法原理

2.6.2 数学基础

2.6.3 常见变种
去噪自编码器(Denoising Autoencoder, DAE)
在输入数据上添加噪声,通过去噪训练自编码器,使其更具鲁棒性。
稀疏自编码器(Sparse Autoencoder, SAE)
在编码器中添加稀疏性约束,鼓励模型学习稀疏表示。
变分自编码器(Variational Autoencoder, VAE)
基于概率模型的自编码器,通过学习数据的潜在分布生成新数据。
2.6.4 实现步骤
- 数据准备:加载并标准化数据。
- 模型构建:定义编码器和解码器的结构。
- 模型训练:使用反向传播算法最小化重建误差,训练自编码器。
- 数据重建:使用训练好的自编码器对数据进行重建和降维。
2.6.5 代码示例
以下是使用Python和PyTorch实现一个简单自编码器的示例代码:
import torch import torch.nn as nn import torch.optim as optim import torchvision import torchvision.transforms as transforms from torch.utils.data import DataLoader # 定义自编码器模型 class Autoencoder(nn.Module): def __init__(self): super(Autoencoder, self).__init__() self.encoder = nn.Sequential( nn.Linear(28 * 28, 128), nn.ReLU(True), nn.Linear(128, 64), nn.ReLU(True), nn.Linear(64, 12), nn.ReLU(True), nn.Linear(12, 3) ) self.decoder = nn.Sequential( nn.Linear(3, 12), nn.ReLU(True), nn.Linear(12, 64), nn.ReLU(True), nn.Linear(64, 128), nn.ReLU(True), nn.Linear(128, 28 * 28), nn.Tanh() ) def forward(self, x): x = self.encoder(x) x = self.decoder(x) return x # 数据加载和预处理 transform = transforms.Compose([transforms.ToTensor(), transforms.Normalize((0.5,), (0.5,))]) train_dataset = torchvision.datasets.MNIST(root='./data', train=True, transform=transform, download=True) train_loader = DataLoader(dataset=train_dataset, batch_size=128, shuffle=True) # 初始化模型、损失函数和优化器 model = Autoencoder() criterion = nn.MSELoss() optimizer = optim.Adam(model.parameters(), lr=1e-3) # 模型训练 num_epochs = 20 for epoch in range(num_epochs): for data in train_loader: img, _ = data img = img.view(img.size(0), -1) output = model(img) loss = criterion(output, img) optimizer.zero_grad() loss.backward() optimizer.step() print(f'Epoch [{epoch+1}/{num_epochs}], Loss: {loss.item():.4f}') # 可视化重建结果 import matplotlib.pyplot as plt # 获取一个批次的测试数据 test_dataset = torchvision.datasets.MNIST(root='./data', train=False, transform=transform, download=True) test_loader = DataLoader(dataset=test_dataset, batch_size=10, shuffle=True) dataiter = iter(test_loader) images, _ = dataiter.next() images = images.view(images.size(0), -1) # 重建图像 outputs = model(images) # 展示原始图像和重建图像 fig, axes = plt.subplots(2, 10, figsize=(10, 2)) for i in range(10): axes[0, i].imshow(images[i].view(28, 28).detach().numpy(), cmap='gray') axes[1, i].imshow(outputs[i].view(28, 28).detach().numpy(), cmap='gray') plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
- 54
- 55
- 56
- 57
- 58
- 59
- 60
- 61
- 62
- 63
- 64
- 65
- 66
- 67
- 68
- 69
- 70
- 71
- 72
- 73
- 74
- 75
- 76
- 77
- 78
代码解析
- 定义模型:使用PyTorch定义一个简单的自编码器模型,包括编码器和解码器。编码器将输入数据降维,解码器将低维表示还原为原始数据。
- 数据加载和预处理:使用
torchvision加载MNIST数据集,并对数据进行标准化处理。 - 初始化模型、损失函数和优化器:使用均方误差(MSE)作为损失函数,使用Adam优化器进行模型训练。
- 模型训练:通过反向传播算法最小化重建误差,迭代训练自编码器。
- 可视化重建结果:对测试数据进行重建,并展示原始图像和重建图像的对比。
2.6.6 优缺点
优点
- 特征提取:自编码器能够自动学习数据的低维表示,有助于特征提取和数据降维。
- 数据生成:通过变种自编码器(如VAE),可以生成新数据,具有广泛应用。
- 无监督学习:无需标签数据,自编码器能够在无监督学习任务中发挥重要作用。
缺点
- 过拟合风险:自编码器可能在训练数据上过拟合,导致泛化能力差。
- 高计算成本:训练深度自编码器需要大量计算资源,特别是在大规模数据集上。
- 对噪声敏感:自编码器对噪声数据较为敏感,可能需要去噪自编码器(DAE)来提高鲁棒性。
2.6.7 应用实例
自编码器在多个领域具有广泛应用,例如:
- 图像处理:图像降噪、图像压缩与重建。
- 自然语言处理:文本表示学习、语言模型预训练。
- 生物信息学:基因表达数据降维与聚类分析。



