- 12024年最全一文带你了解——安服岗和渗透岗的区别_安服转渗透容易吗(3)
- 2【Home Assistant】HACS 安装_安装hacs后在集成中找不到
- 3实战Java高并发程序设计 PDF
- 4css设置滚动条透明和自定义_::-webkit-scrollbar{ width: 0; height: 0; color: t
- 5Stable Diffusion 3 如何下载安装使用及性能优化
- 6机器学习Notes之One-Hot编码_dataframe one-hot编码
- 7Spark系列二:SparkCore的RDD算子Transformation和Action详解_rdd上支持的transformation和action算子
- 8新书速览|PyTorch 2.0深度学习从零开始学_pytorch2.0深度学习从零开始学 pdf
- 9华为OD机试C卷-- 二叉树的广度优先遍历(Java & JS & Python & C)_二叉树的广度优先遍历 华为od
- 10GPT实战系列-LangChain构建自定义Agent_langchain 自定义agent
EM算法(期望最大化算法)理论概述_maximum likelihood from incomplete data via the em
赞
踩
1.EM算法
1.1概述
EM(Expectation-Maximum)算法也称期望最大化算法,曾入选“数据挖掘十大算法”中,可见EM算法在机器学习、数据挖掘中的影响力。EM算法是最常见的隐变量估计方法,在机器学习中有极为广泛的用途,例如常被用来学习高斯混合模型(Gaussian mixture model,简称GMM)的参数;隐式马尔科夫算法(HMM)、LDA主题模型的变分推断等等。
EM算法是一种迭代优化策略,由于它的计算方法中每一次迭代都分两步,其中一个为期望步(E步),另一个为极大步(M步),所以算法被称为EM算法(Expectation-Maximization Algorithm)。EM算法受到缺失思想影响,最初是为了解决数据缺失情况下的参数估计问题,其算法基础和收敛有效性等问题在Dempster、Laird和Rubin三人于1977年所做的文章《Maximum likelihood from incomplete data via the EM algorithm》中给出了详细的阐述。其基本思想是:首先根据己经给出的观测数据,估计出模型参数的值;然后再依据上一步估计出的参数值估计缺失数据的值,再根据估计出的缺失数据加上之前己经观测到的数据重新再对参数值进行估计,然后反复迭代,直至最后收敛,迭代结束。EM算法的核心就是最大似然估计
1.2 预备知识
想清晰的了解EM算法推导过程和其原理,我们需要知道两个基础知识:“极大似然估计”和“Jensen不等式”。
1.3 极大似然估计
EM算法的核心:极大似然估计


1.4 Jensen不等式(EM算法推导)
问题:样本集{x(1), x(2),...x(m)}有m个独立样本,其中每个样本i对应的类别z(i)都是未知的,所以很难用最大似然估计求解
 所以只能选用迭代的方式进行求解。
所以只能选用迭代的方式进行求解。
 对于m一个样本,先选取某一个样本进行考虑
对于m一个样本,先选取某一个样本进行考虑






1.5 EM算法流程

1.6 GMM高斯混合模型

2.代码实现
2.1 GMM实现
2.1.1 数据集地址
https://download.csdn.net/download/bigData1994pb/19189840?spm=1001.2014.3001.5501
2.1.2 代码详解
项目分析如下所:
- import pandas as pd
- from matplotlib import pyplot as plt
- from sklearn.decomposition import PCA
- from sklearn.mixture import GaussianMixture
- from sklearn.datasets.samples_generator import make_blobs
- data = pd.read_csv(r'C:\Users\Desktop\Fremont.csv', index_col='Date', parse_dates=True)
- print(data.head())
- data = data.drop(columns=['Fremont Bridge Total'], axis=1)
- plt.plot(data) # 这是以每个小时的用户量所画的图,不容易观察趋势,对于这种是时间序列的而且又是密集的,我们可以对数据在时间上进行重采样
- plt.show()
- # 数据重采样,按周进行计算
- plt.plot(data.resample('w').sum())
- plt.show()
- # 对数据做一个滑动窗口进行统计(每一个点都表示前365天)
- data_stas = data.resample('d').sum().rolling(365).sum()
- plt.plot(data_stas)
- plt.show()
- data.columns = ['West', 'East']
- # print(data.head())
- data['Total'] = data['West']+data['East']
- # 按照时间的维度画图示表
- pivoted = data.pivot_table('Total', index=data.index.time, columns=data.index.date)
- print(pivoted.iloc[:5, :5])
- # plt.plot(pivoted,alpha=0.01)
- # # pivoted.plot(alpha=0.01)
- # plt.xticks(rotation=45)
- # plt.show()
- print(pivoted.shape)
- # 首先进行缺失值填充。再者进行转置,因为上面是(24,1763)样本数太少,而特征太多,因此进行转置处理
- X = pivoted.fillna(0).T.values # 变为(1763, 24)
- print(X)
-
- # 原来是24维的降维到2维的
- X2 = PCA(2).fit_transform(X)
- print(X2.shape)
-
- # 画出散点图
- plt.scatter(X2[:, 0], X2[:, 1])
- plt.show()
- gmm = GaussianMixture(2)
- gmm.fit(X)
- # 预测每个样本属于两个分类的概率
- labels = gmm.predict_proba(X) # 得到一个预测概率值
- print(labels)
- labels = gmm.predict(X) # 得到一个预测标签值
- print(labels)
- # 对0和1分别画不同的颜色
- plt.scatter(X2[:, 0], X2[:, 1], c=labels, cmap='rainbow')
- plt.show()
- # 由于前面分析的是经过降维后的数据,因此若想看降维钱的数据分布则需要做一下处理
- fig, ax = plt.subplots(1, 2, figsize=(14, 6))
- pivoted.T[labels==0].T.plot(legend=False, alpha=0.1, ax=ax[0])
- pivoted.T[labels==1].T.plot(legend=False, alpha=0.1, ax=ax[1])
- ax[0].set_title('Purple Cluster')
- ax[1].set_title('Red Cluster')
- # 可以通过绘图发现紫色的分布和红色的分布是完全不一样的,因此利用高斯分布可以进行很好的聚类
- plt.show()

2.2 GMM与K-mens的区别
在这里咱们用一个小案例进行解释:
首先随机生成一组数据:
- X, y_true =make_blobs(n_samples=800, centers=4, random_state =11)# 有四个中心说明有4个堆
- plt.scatter(X[:, 0], X[:, 1])
- plt.show()

利用生成的一组数据验证K-means与GMM的区别:
1.k-means聚类结果
用不同的颜色对不同的标签数据染色
- kmeans = KMeans(n_clusters=4)
- kmeans.fit(X)
- y_kmeans = kmeans.predict(X)
- plt.scatter(X[:, 0], X[:, 1], c=y_kmeans, s=50, cmap='viridis') # 给不同的标签值赋予不同的颜色
- # kmeans.cluster_centers_
- plt.show()
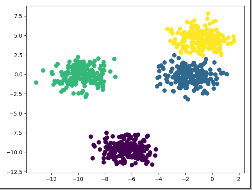
2.GMM聚类
- # 利用GMM聚类
- gmm = GaussianMixture(n_components=4).fit(X)
- labels = gmm.predict(X)
- plt.scatter(X[:, 0], X[:, 1], c=labels, cmap='viridis')
- plt.show()

通过以上数据可以看出利用K-means与GMM算法差距不大,那么我们换一组数据试试。
再生成一组随机数如下做k-means和GMm处理如下所示:
- rng = np.random.RandomState(13) # 产生一个随机数,随机数种子13或者其他数只要相同生成的随机数就都是一样的
- x_stretched = np.dot(X, rng.randn(2, 2))
- kmeans = KMeans(n_clusters=4, random_state=1)
- kmeans.fit(x_stretched)
- y_kmeans = kmeans.predict(x_stretched)
- plt.scatter(x_stretched[:, 0], x_stretched[:, 1], c=y_kmeans, s=50, cmap='viridis')
- plt.show()

可以看出利用k-means不容易区分,我的想法是要上面一类下面一类,但是在左上角好像没有分的开。由于k-means是基于距离的聚类,而不是基于数据内部服从某种分布的聚类
- gmm = GaussianMixture(n_components=4).fit(x_stretched)
- labels = gmm.predict(x_stretched)
- plt.scatter(x_stretched[:, 0], x_stretched[:, 1], c=labels, cmap='viridis')
- plt.show()

可以看出利用GMM可以利用数据之间服从不同的分布而进行分类


