- 1如何使用git上传本地代码到码云上(超详细的呦)_本地的代码仓库如何跟码云同步
- 2经典算法题汇总
- 3机器学习(五)Python+OpenCV+dlib实现人脸识别_python人脸识别门禁系统 opencv+dlib
- 4理论+实验:MySQL数据库基本概念和介绍_mysql的理论和实验的联系
- 5LeetCode70爬楼梯_line 8: char 16: runtime error: index -2 out of bo
- 6在图像中隐藏数据:用 Python 来实现图像隐写术_hugo隐写算法python实现
- 7leetocde662. 二叉树最大宽度,面试必刷题,思路清晰,分点解析,附代码详解带你完全弄懂
- 8Vue 实现电子签名_vue实现电子签名
- 9了解和使用 Docker 镜像仓库_docker 镜像库
- 10关系型数据库学习手记——初见倾心PostgreSQL、MySQL、SQLite、MongoDB_在 postgresql (pg) 中实现动态哈希分表
机器学习笔记(十)聚类算法DBSCAN原理和实践_dbscan -1
赞
踩
在前面的文章中,我们分别介绍了《K-means原理和实践》和《Birch和层次聚类》两种聚类算法,本文我们继续介绍另一种常用的聚类算法DBSCAN。相对于前两种算法,DBSCAN的原理要简单的多,但是这并不意味着它的效果就会差,在很多算法表现不好的非凸数据集上(凸数据集可以简单理解为数据集中的任意两点连线上的点都在数据集内),DBSCAN往往能取得较好的效果,见下图,这也是DBSCAN最大的优势,而且DBSCAN还可以作为异常检测算法,发现噪声点(离群点)。另外,本文讲解原理篇多引用了文献[1]的内容,有时间的话,建议也看看原文。

一、原理
DBSCAN(Density-Based Spatial Clustering of Applications with Noise,具有噪声的基于密度的聚类方法)是一种基于密度的空间聚类算法。 该算法将具有足够密度的区域划分为簇,并在具有噪声的空间数据库中发现任意形状的簇,它将簇定义为密度相连的点的最大集合。关于什么是密度相连我们下面解释。
1.1 密度定义
DBSCAN是基于一组邻域来描述样本集的紧密程度的,参数(eps, min_samples)用来描述邻域的样本分布紧密程度。其中,eps描述了某一样本的邻域距离阈值,min_samples描述了某一样本的距离为eps的邻域中样本个数的阈值[1]。
假设已知样本集
- eps-邻域:对于
xj∈D ,其eps-邻域包含样本集D中与xj 的距离不大于eps的子样本集,这个子样本集的个数记为|N∈(xj)| ,这里需要注意距离并不一定是欧氏距离,具体在API章节会再详细介绍。 - 核心对象:核心对象,又叫核心点。对于任一样本
xj∈D ,如果其eps-邻域对应的|N∈(xj)| 不小于 min_samples,即如果|N∈(xj)| ≥ min_samples,则xj 是核心对象。 - 密度直达:如果
xi 位于xj 的eps-邻域中,且xj 是核心对象,则称xi 由xj 密度直达。注意反之不一定成立,即此时不能说xj 由xi 密度直达, 除非且xi 也是核心对象。 - 密度可达:对于
xi 和xj ,如果存在样本序列p1,p2,p3,...,pT , 满足p1=xi,pT=xj ,且pt+1 由pt 密度直达,则称xj 由xi 密度可达。也就是说,密度可达满足传递性。此时序列中的传递样本p1,p2,...,pT−1 均为核心对象,因为只有核心对象才能使其他样本密度直达。注意密度可达也不满足对称性,这个可以由密度直达的不对称性得出。 - 密度相连:对于
xi 和xj ,如果存在核心对象样本xk ,使xi 和xj 均由xk 密度可达,则称xi 和xj 密度相连。注意密度相连关系是满足对称性的。这一点由密度可达的定义可以很容易得出。
上面的定义理解起来会有点抽象,看下面这个图就比较容易理解了,图中min_samples=5,红色点都是核心对象。位于每一个核心对象圆圈内(eps-邻域)的点都可以由对应的核心对象密度直达,图中绿色箭头连接起来的核心对象都是密度可达的样本序列,在这些密度可达的样本序列的eps-邻域(黑色圆圈内的点)内所有的样本相互都是密度相连的。

1.2 聚类原理
有了密度定义,DBSCAN的聚类原理就很简单了,由密度可达关系导出的最大密度相连的样本集合,即为我们最终聚类的一个类别,或者说一个簇。所以任意一个DBSACN簇都至少有一个核心对象,如果有多于一个核心对象,则这些核心对象必然可以组成密度可达序列。
那么怎么才能找到这样的簇样本集合呢?DBSCAN使用的方法很简单,它任意选择一个没有类别的核心对象作为种子,然后找到所有这个核心对象能够密度可达的样本集合,即为一个聚类簇。接着继续选择另一个没有类别的核心对象去寻找密度可达的样本集合,这样就得到另一个聚类簇。一直运行到所有核心对象都有类别为止。参考上图的两个绿色箭头路径,聚类的两个簇。
聚类完成后,样本集中的点一般可以分为三类:核心点,边界点和离群点(异常点)。边界点是簇中的非核心点,离群点就是样本集中不属于任意簇的样本点。此外,在簇中还有一类特殊的样本点,该类样本点可以由两个核心对象密度直达,但是这两个核心对象又不能密度可达(属于两个簇),该类样本的最终划分簇一般由算法执行顺序决定,即先被划分到哪个簇就属于哪个簇,也就是说DBSCAN的算法不是完全稳定的算法。这也意味着DBSCAN是并行算法,对于两个并行运算结果簇,如果两个簇中存在两个核心对象密度可达,则两个簇聚为一个簇。
根据上面介绍的原理可知,DBSCAN的主要计算量仍然是在距离上,这里同样可以借鉴 elkan K-means 和 KNN KD树/球树的原理进行优化,减少计算量。
1.3 调参
DBSCAN有两个关键参数eps,和min_samples,一般可通过网格搜索加无监督聚类指标的方法调参(具体指标可参考kmeans篇),也可以结合特征的方差、均值分布来调参。对于特定场景的业务,如果已知某些先验参数,例如簇数,则可以通过该指标对网格参数结果进行筛选,然后再从剩下的聚类结果中选择合适的结果。如果是有监督聚类建模,还可以在聚类之前通过 NCA 等方法对特征进行变换,一般对于 DBSCAN 这种基于密度聚类的算法可以得到一个更好的结果。
1.4 优缺点
(1)优点
- 不需要预先指定聚类数量,可以对任意形状的稠密数据集进行聚类,相对的,K-Means之类的聚类算法一般只适用于凸数据集。
- 可以在聚类的同时发现异常点,对数据集中的异常点不敏感。
- 聚类结果没有偏倚,相对的,K-Means之类的聚类算法初始值对聚类结果有很大影响。
(2)缺点
- 如果样本集的密度不均匀、聚类间距差相差很大时,聚类质量较差,这时用DBSCAN聚类一般不适合。
- 如果样本集较大时,聚类收敛时间较长,此时可以对搜索最近邻时建立的KD树或者球树进行规模限制来改进。
- 调参相对于传统的K-Means之类的聚类算法稍复杂,主要需要对距离阈值eps,邻域样本数阈值min_samples联合调参,不同的参数组合对最后的聚类效果有较大影响。eps越大,得到的簇数也就越少,得到的簇就越大,离群点就越少;min_samples越大,离群点就越多,得到的簇越小。
最后再给一个聚类算法的可视化测试网址,有兴趣的可以去玩玩,对于理解聚类的过程非常友好:
https://www.naftaliharris.com/blog/visualizing-dbscan-clustering/
二、实践
本节我们同样通过 sklearn 来学习 DBSCAN的使用。
2.1 API
- sklearn.cluster.DBSCAN(
- eps=0.5,
- *,
- min_samples=5, ## min_samples参数计算的时候注意是包括样本点自身的
- metric='euclidean', ## 距离度量方式,默认是欧氏距离,具体选择可参考KNN篇,选择precomputed表示自己计算距离,fit时X传入距离矩阵即可
- metric_params=None, ## metric的辅助参数,根据metric的值决定
- algorithm='auto', ## 近邻点的查找方法,可选‘auto’, ‘ball_tree’, ‘kd_tree’, ‘brute’。默认auto会根据样本数量自动选择,具体可参考前面KNN的文章
- leaf_size=30, ## BallTree or cKDTree参数
- p=None, ## 闵可夫斯基距离的p,默认None就是2,表示欧式距离
- n_jobs=None)
labels_ 属性可以返回聚类结果,-1表示是离群点。
另外,提一下关于 DBSCAN 预测的问题,可以发现在 sklearn 中 DBSCAN 是没有 predict 方法的,因为很多聚类算法和 kmeans 原理不同,不能根据训练样本的簇中心点来预测新的样本所属簇。不过我们可以自己根据原理来实现预测过程,第一种就是像 kmeans 一样,得到簇结果以后,计算出簇的中心点,根据新样本距离所有簇中心的远近判断所属的簇,但是这种方法对于 DBSCAN 不适合。第二种是基于聚类结果,再运行 KNN 算法,找到距离新样本点最近的一个或者多个样本点所属的簇就是新样本点所属的簇。
2.2 实践
使用sklearn先对特征进行归一化,再聚类。
- import numpy as np
-
- from sklearn.cluster import DBSCAN
- from sklearn import metrics
- from sklearn.datasets import make_blobs
- from sklearn.preprocessing import StandardScaler
-
-
- # #############################################################################
- # Generate sample data
- centers = [[1, 1], [-1, -1], [1, -1]]
- X, labels_true = make_blobs(n_samples=750, centers=centers, cluster_std=0.4,
- random_state=0)
-
- X = StandardScaler().fit_transform(X)
-
- # #############################################################################
- # Compute DBSCAN
- db = DBSCAN(eps=0.3, min_samples=10).fit(X)
- core_samples_mask = np.zeros_like(db.labels_, dtype=bool)
- core_samples_mask[db.core_sample_indices_] = True
- labels = db.labels_
-
- # Number of clusters in labels, ignoring noise if present.
- n_clusters_ = len(set(labels)) - (1 if -1 in labels else 0)
- n_noise_ = list(labels).count(-1)
-
- print('Estimated number of clusters: %d' % n_clusters_)
- print('Estimated number of noise points: %d' % n_noise_)
- print("Homogeneity: %0.3f" % metrics.homogeneity_score(labels_true, labels))
- print("Completeness: %0.3f" % metrics.completeness_score(labels_true, labels))
- print("V-measure: %0.3f" % metrics.v_measure_score(labels_true, labels))
- print("Adjusted Rand Index: %0.3f"
- % metrics.adjusted_rand_score(labels_true, labels))
- print("Adjusted Mutual Information: %0.3f"
- % metrics.adjusted_mutual_info_score(labels_true, labels))
- print("Silhouette Coefficient: %0.3f"
- % metrics.silhouette_score(X, labels))
-
- # #############################################################################
- # Plot result
- import matplotlib.pyplot as plt
-
- # Black removed and is used for noise instead.
- unique_labels = set(labels)
- colors = [plt.cm.Spectral(each)
- for each in np.linspace(0, 1, len(unique_labels))]
- for k, col in zip(unique_labels, colors):
- if k == -1:
- # Black used for noise.
- col = [0, 0, 0, 1]
-
- class_member_mask = (labels == k)
-
- xy = X[class_member_mask & core_samples_mask]
- plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=tuple(col),
- markeredgecolor='k', markersize=14)
-
- xy = X[class_member_mask & ~core_samples_mask]
- plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=tuple(col),
- markeredgecolor='k', markersize=6)
-
- plt.title('Estimated number of clusters: %d' % n_clusters_)
- plt.show()
-
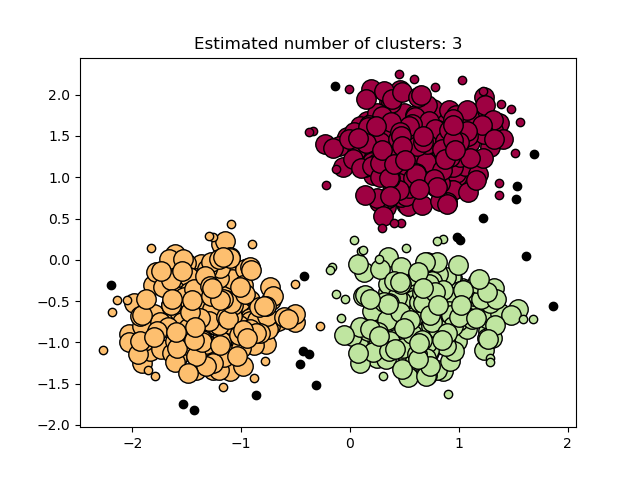
- #######################运行结果################################
-
- Estimated number of clusters: 3
- Estimated number of noise points: 18
- Homogeneity: 0.953
- Completeness: 0.883
- V-measure: 0.917
- Adjusted Rand Index: 0.952
- Adjusted Mutual Information: 0.916
- Silhouette Coefficient: 0.626

参考文献
[1] https://www.cnblogs.com/pinard/p/6208966.html
[2] https://blog.csdn.net/u012848304/article/details/108710864


