- 1udhcpd简单配置_udhcpd 配置
- 2堆的数据结构以及堆的相应操作
- 3无线通信基本原理_无线通信原理
- 4访问CentOS7光盘_centos7怎么打开光盘命令
- 5《UE5_C++多人TPS完整教程》学习笔记2 ——《P3 多人游戏概念(Multiplayer Concept)》
- 6chatgpt赋能python:用Python进行股票分析_用chatgpt写股票分析报告
- 7机器学习笔记 - 深度学习在网球运动分析上的应用_ibm slamtracker
- 8vue3 中Element Plus的el-select 样式(包括更换小图标样式)_vue
- 9使用python实现跨年烟花代码_python跨年代码
- 10大语言模型介绍(一)
Freesurfer学习笔记——Introduction to Freesurfer Output(输出简介)_freesurfer 输出文件大小
赞
踩
先看看通过freesurfer我们得到的输出是什么样子的,做到胸中插竹子。
Preparations(准备工作):下载数据集,设置环境
- export TUTORIAL_DATA=<path_to_your_tutorial_data> //导入环境,设置你的数据集解压后的文件夹路径:TUTORIAL_DATA
- export SUBJECTS_DIR=$TUTORIAL_DATA/buckner_data/tutorial_subjs //设置工作目录
- cd $SUBJECTS_DIR //进入工作目录
Viewing Volumes with Freeview(用Freeview看看我们的脑子长什么样)
- $> export FREESURFER_HOME=/Applications/freesurfer
- $> source $FREESURFER_HOME/SetUpFreeSurfer.sh //启动freesurfer应用程序
- freeview -v \ //反斜杠允许我们多行同时操作(直接复制这一整段命令上去就行)
- good_output/mri/T1.mgz \
- good_output/mri/wm.mgz \
- good_output/mri/brainmask.mgz \
- good_output/mri/aseg.mgz:colormap=lut:opacity=0.2 \
- -f good_output/surf/lh.white:edgecolor=blue \
- good_output/surf/lh.pial:edgecolor=red \
- good_output/surf/rh.white:edgecolor=blue \
- good_output/surf/rh.pial:edgecolor=red //启动freeview,看看T1/wm/brainmask/aseg,并且把左边/右边white表面用蓝色标示
命令-v :打开常用的volumes
brainmask.mgz:主要用于故障排除的头盖骨剥离体积
wm.mgz:白质遮罩也用于故障排除
aseg.mgz :皮层下分割,用相应的颜色表加载,透明度低。有关皮层下分割的详细信息,请参见此处。
命令-f :用于打开surfaces
每个半球都有白色和圆形表面,颜色由“edgecolor”表示

Freeview顶部工具栏中下图三个按钮用来更改主查看窗口中显示的正交视图。矢状视图(sagittal)|冠状视图(coronal)|水平视图(Axial)。

Freeview顶部工具栏中下图四个按钮用来更改查看窗格的组织。

要查看大脑组织切片进行微调时用键盘中的上下键,mac用户使用fn+上下键。
尽管Freeview可以一次加载多个卷,但您不一定能同时看到所有卷。您可以在左侧菜单中看到列表顶部的卷。但wm.mgz和aseg.mz这样的卷是一个例外,它们可以被设置为半透明的,这样您就可以同时查看它们包含的信息和列表中它正下方的卷。例如,您当前可以看到来自aseg(标记结构)和brainmask(体素强度)的信息。
通过取消选中图层名称旁边的复选框,可以隐藏或关闭图层。您还可以使用上下箭头(位于左侧菜单下方)将列表上的aseg向下移动到脑罩下方(试试!)。现在让我们把wm卷移到列表的顶部,但是不要使用箭头,试试这个快捷方式:双击显示wm的地方。它应该会自动移动到顶部。
键盘快捷键:Alt+c(mac:Fn+Option+c)将允许您快速循环浏览所有图层。每次点击时,列表顶部的volumes都会移到列表底部。Mac用户,所有Freeview键盘快捷键都位于此处。
Freeview顶部工具栏中下图按钮选中后,按住鼠标中键(滑轮)可以在查看窗口移动图像,也可以使用中键滚动来进行缩放。左键单击图像,出现光标(小红十字线),当您更改方向(轴向或矢状)时,您将看到与光标位置相交的切片。

注意:在查看窗口中执行的任何操作(即擦除、更改亮度等)都将在左菜单中当前突出显示的volumes上执行,而不管哪个文件位于列表顶部。
------------------------------------------------------
双击左侧菜单中的“brainmask”,将其置于volumes列表的顶部。白质表面(蓝线)用于计算总白质体积,并应准确遵循白质和灰质之间的边界。软脑膜表面(红线)用于计算皮质灰质体积,并应准确遵循灰质和脑脊液之间的边界。
键盘快捷键:Alt+f(mac:Command+f)将打开和关闭菜单窗口中突出显示的任何曲面surface。
左侧菜单中的brainmask高亮显示时,通过按住“Shift”键的同时左键单击图像并拖动鼠标,可以调整亮度和对比度从而识别灰度和白质之间强度的变化。或者通过左侧菜单下方的“窗口”和“级别”滑块来执行此操作。
切片号显示在查看窗口的右上角。
----------------------------------------------
Subcortical Segmentation(皮质下分割)
取消选中所有surfaces,双击aseg把他调整到顶部,brainmask的上面,将显示皮质下结构的完整分割。

每个结构都用唯一的颜色/数字区分标记。如果单击体素,则结构的名称和编号标签将显示在“aseg”一词旁边的查看窗口下的“Cursor”部分。如果将鼠标悬停在光标不在的体素上,该体素的值将显示在“鼠标”部分下。
键盘快捷键:Alt+v(mac:Option+v)将打开和关闭当前高亮显示的图层。
确保“aseg”在左侧菜单中突出显示,然后按Alt+v将其关闭和打开。进行此操作时,请确保aseg精确地遵循每个结构的基本强度边界。你也可以调整“不透明度Opacity”滑块,以更好地看到潜在的brainmask。
键盘快捷键:Alt+a(mac:Command+a,Toggle aseg.mgz visibility in layer list)(mac:Command+w,Toggle wm.mgz visibility in layer list)和Alt+s将更改当前高亮显示的图层的不透明度。
--------------------------------------------------------------------------------------
Skull Strip
关闭aseg,图像中只去掉了头骨。将T1.mgz调到volumes列表的顶部,并在它和brainmask.mgz (Alt+v)之间切换,可以看到去掉头骨前后的变化。
Intensity Normalization
在brainmask图像中滚动鼠标,强度不变,同时白质或灰质中无任何非常亮或非常暗的斑点。 点击白质中的一个体素,可以看到它已经被标准化为110的强度(或接近110的强度)(在光标部分下面,它旁边写着“brainmask”)。当wm体素远离110的值时,它们可能被错误地排除在白质表面之外。我们所说的“接近”,是指wm体素的强度值在100到110之间。我们所说的“远”是指wm体素的值在85到100之间。
WM Volume
双击wm Volume将其置于列表顶部,他是FreeSurfer对白质的初始分割(以灰色显示),并添加了自动拓扑修复程序(以白色显示)。这个“遮罩mask”是white surface的起点,white surface从这里生长,并以brainmask.mgz体积中的强度梯度作为指导,停在一个更精确的位置。wm.mgz可以用来添加丢失的wm体素或删除非白质但包含在曲面中的体素。将wm mask更改为热覆盖有利于编辑。“heat”设置用于编辑,因为它允许我们看到底层解剖结构,同时仍然能够在wm.mgz“mask”中定位不准确的地方。也可以使用“Jet”覆盖。在左侧菜单中突出显示wm时,查看颜色映射(color map)的选项,然后选择“Heat”或“Jet”。然后调整不透明度,这样你也可以看到下面的brainmask(下降到大约0.25)。

第一次加载wm.mgz时,可以通过运行下面这行命令,实现heat overlay:
freeview -v wm.mgz:colormap=heat:opacity=0.25 brainmask.mgz
通过点击Freeview显示窗口上的X或Ctrl-q可以关闭Freeview。
---------------------------------------------------------------------------------
Viewing Surfaces in 3D using Freeview
1.pial, white and inflated surface
2.sulcal and curvature maps
3.thickness maps
4.cortical parcellation
通过下面命令可以加载上面这些surfaces,并且显示为3D:
- freeview -f good_output/surf/lh.pial:annot=aparc.annot:name=pial_aparc:visible=0 \
- good_output/surf/lh.pial:annot=aparc.a2009s.annot:name=pial_aparc_des:visible=0 \
- good_output/surf/lh.inflated:overlay=lh.thickness:overlay_threshold=0.1,3::name=inflated_thickness:visible=0 \
- good_output/surf/lh.inflated:visible=0 \
- good_output/surf/lh.white:visible=0 \
- good_output/surf/lh.pial \
- --viewport 3d//
Pial Surface
Pial surface 是 brainmask volume 的2D切片上红色surface的完整3D表示。绿色区域是脑回,红色区域是脑沟。脑沟大部分被隐藏了。左键单击并拖动鼠标,即可随意移动膨胀的大脑。 如果您想使大脑恢复到原始状态,请转到“视图”>“重置视图”,按Ctrl + r(mac:command+r),或单击“重置”按钮:![]()

要消除显示在曲面上的黄线(对应于3个切片视图),请右键单击曲面并选中“隐藏所有切片”,或单击Crtl + Shift + s(mac:Shift+Command+s)。
White Surface
按Alt + c(mac:Fn+Option+c)循环到white surface。

white surface显示了白质和灰质之间的边界。它是brainmask的2D切片上的蓝色surface的3D表示。
Inflated Surface

随着表面的膨胀,可以完全看到脑沟。 选择左侧菜单中的“曲率Curvature”中的binary(二进制)可以查看灰度的曲率,选择off则可以查看光滑的充气表面。
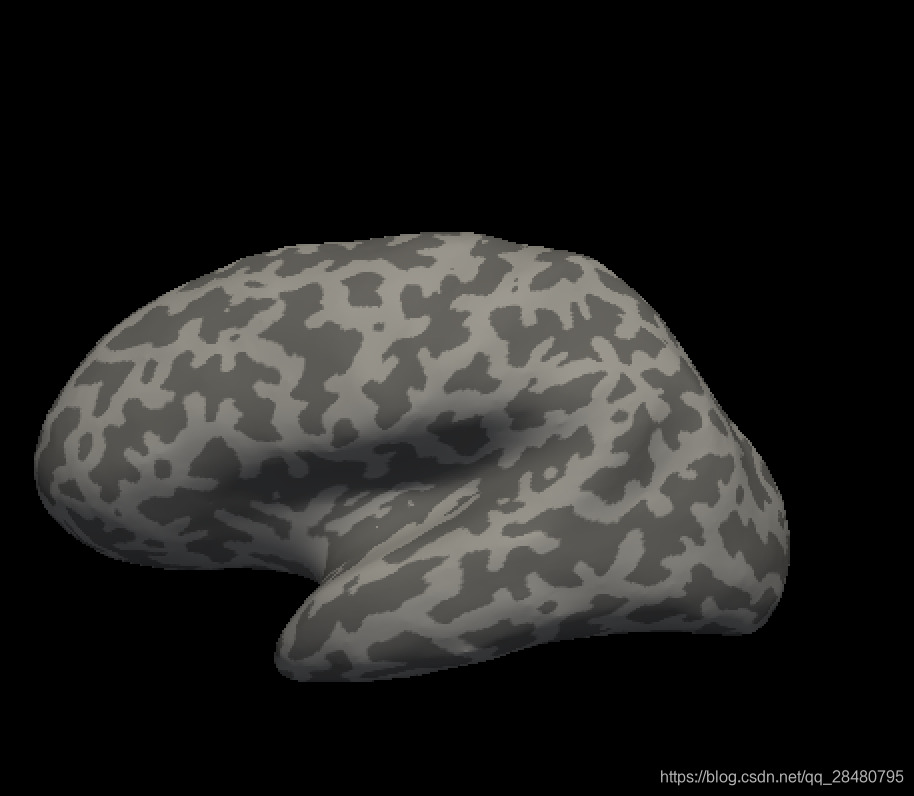

充气后的表面有助于发现可能需要纠正的凸块,孔洞或其他缺陷。 如果单击充气后的曲面,将在“光标Cursor”窗口中看到所单击的顶点的坐标。
Thickness Map(厚度图)

Cortical Parcellation(皮质分割)
左侧(pial_aparc)是Desikan-Killiany组合,右侧(pial_aparc_des)是Destrieux组合。
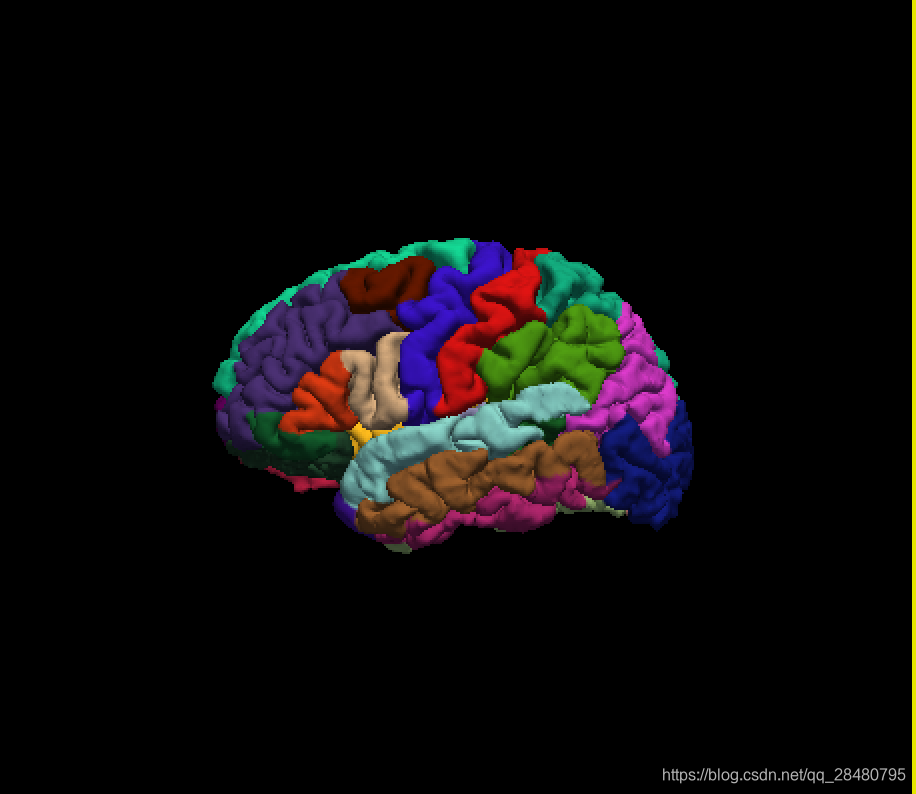

单击一种颜色,然后在“光标Cursor”或“鼠标Mouse”窗口中显示“ aparc.annot”的位置查看皮质区域的名称。 旋转大脑,以便观察内侧墙。 注意,所有皮层下灰质都不是surface labels的一部分(再次,因为这些区域不会计入皮层表面度量)。
默认情况下,运行recon-all时会进行两个分割。 这两个部分是使用Desikan-Killiany图集创建的aparc.annot 和使用Destrieux图集创建的aparc.a2009s.annot 。 区别在于所标记区域的数量和名称。 可以通过单击左侧面板“Annotation注释”旁边的下拉框,选择“Load from file.... 从文件加载...”来加载Destrieux parcellation。浏览至lh.aparc.a2009s.annot并单击“打开”。
海马和杏仁核周围的区域并非准确无误,由于皮层下区域被排除在皮层测量范围之外,因此这些不正确之处可以忽略不计。
Surfaces can be viewed in 3D such as the pial, white, and inflated surfaces, sulcul and cortical maps, thickness maps, and cortical parcellation.
FreeviewGuide 中可以查看全部的按钮或者菜单选项的信息。
//


