- 1微信小程序获取用户定位(经纬度)信息_微信小程序获取位置信息
- 2[异常处理]class kafka.common.UnknownTopicOrPartitionException (kafka.server.ReplicaFetcherThread)_unknown topic or partition error in listoffset
- 3centos 杀死进程命令kill
- 4微信小程序页面开发-本地生活_为想见你小程序开发本地生活
- 5Git——多人协作/版本控制,在一个gitee仓库下开发(Gitee版教程)手把手教学,包好用的!_gitee多人协作
- 6FPGA 20个例程篇:20.USB2.0/RS232/LAN控制并行DAC输出任意频率正弦波、梯形波、三角波、方波(三)_fpga怎么输出任意频率的方波
- 7华为超聚变2288H V6服务器配置RAID及安装ESXI7.0-闭坑,亲测_华为超聚变服务器 怎么配置raid
- 8如何快速解决验证码图像问题 | 最佳图像(OCR)验证码解决工具
- 9ios开发错误积累_ios 17.4 simulator
- 10京东面经分享
(python实现)一篇文章教会你k-means聚类算法(包括最优聚类数目k的确定)_kmeans聚类算法确定聚类数
赞
踩
摘要
Kmeans算法中,K值所决定的是在该聚类算法中,所要分配聚类的簇的多少。Kmeans算法对初始值是⽐较敏感的,对于同样的k值,选取的点不同,会影响算法的聚类效果和迭代的次数。本文通过计算原始数据中的:手肘法、轮廓系数、CH值和DB值,四种指标来衡量K-means的最佳聚类数目,并使用K-means进行聚类,最后可视化聚类的结果。
一篇文章教会你如何使用matlab进行K-means聚类,以及如何确定最优k值,不要再去看那些付费文章了。
1. K-means算法
1.1 聚类算法简介
对于"监督学习"(supervised learning),其训练样本是带有标记信息的,并且监督学习的目的是:对带有标记的数据集进行模型学习,从而便于对新的样本进行分类。而在“无监督学习”(unsupervised learning)中,训练样本的标记信息是未知的,目标是通过对无标记训练样本的学习来揭示数据的内在性质及规律,为进一步的数据分析提供基础。对于无监督学习,应用最广的便是"聚类"(clustering)。
"聚类算法"试图将数据集中的样本划分为若干个通常是不相交的子集,每个子集称为一个“簇”(cluster),通过这样的划分,每个簇可能对应于一些潜在的概念或类别。
1.2 K-means聚类算法
kmeans算法又名k均值算法,K-means算法中的k表示的是聚类为k个簇,means代表取每一个聚类中数据值的均值作为该簇的中心,或者称为质心,即用每一个的类的质心对该簇进行描述。
其算法思想大致为:先从样本集中随机选取 k个样本作为簇中心,并计算所有样本与这 k个“簇中心”的距离,对于每一个样本,将其划分到与其距离最近的“簇中心”所在的簇中,对于新的簇计算各个簇的新的“簇中心”。
根据以上描述,我们大致可以猜测到实现kmeans算法的主要四点:
(1)簇个数 k 的选择
(2)各个样本点到“簇中心”的距离
(3)根据新划分的簇,更新“簇中心”
(4)重复上述2、3过程,直至"簇中心"没有移动。
1.3 代码实现
注意:需要先通过下文2部分确定k值
import random import pandas as pd import numpy as np import matplotlib.pyplot as plt # 计算欧式距离 def calcDis(dataSet, centroids, k): clalist=[] for data in dataSet: diff = np.tile(data, (k, 1)) - centroids #相减 (np.tile(a,(2,1))就是把a先沿x轴复制1倍,即没有复制,仍然是 [0,1,2]。 再把结果沿y方向复制2倍得到array([[0,1,2],[0,1,2]])) squaredDiff = diff ** 2 #平方 squaredDist = np.sum(squaredDiff, axis=1) #和 (axis=1表示行) distance = squaredDist ** 0.5 #开根号 clalist.append(distance) clalist = np.array(clalist) #返回一个每个点到质点的距离len(dateSet)*k的数组 return clalist # 计算质心 def classify(dataSet, centroids, k): # 计算样本到质心的距离 clalist = calcDis(dataSet, centroids, k) # 分组并计算新的质心 minDistIndices = np.argmin(clalist, axis=1) #axis=1 表示求出每行的最小值的下标 newCentroids = pd.DataFrame(dataSet).groupby(minDistIndices).mean() #DataFramte(dataSet)对DataSet分组,groupby(min)按照min进行统计分类,mean()对分类结果求均值 newCentroids = newCentroids.values # 计算变化量 changed = newCentroids - centroids return changed, newCentroids # 使用k-means分类 def kmeans(dataSet, k): # 随机取质心 centroids = random.sample(dataSet, k) # 更新质心 直到变化量全为0 changed, newCentroids = classify(dataSet, centroids, k) while np.any(changed != 0): changed, newCentroids = classify(dataSet, newCentroids, k) centroids = sorted(newCentroids.tolist()) #tolist()将矩阵转换成列表 sorted()排序 # 根据质心计算每个集群 cluster = [] clalist = calcDis(dataSet, centroids, k) #调用欧拉距离 minDistIndices = np.argmin(clalist, axis=1) for i in range(k): cluster.append([]) for i, j in enumerate(minDistIndices): #enymerate()可同时遍历索引和遍历元素 cluster[j].append(dataSet[i]) return centroids, cluster # 创建数据集 def createDataSet(): return [[1, 1], [1, 2], [2, 1], [6, 4], [6, 3], [5, 4]] if __name__=='__main__': dataset = createDataSet() centroids, cluster = kmeans(dataset, 2) print('质心为:%s' % centroids) print('集群为:%s' % cluster) for i in range(len(dataset)): plt.scatter(dataset[i][0],dataset[i][1], marker = 'o',color = 'green', s = 40 ,label = '原始点') # 记号形状 颜色 点的大小 设置标签 for j in range(len(centroids)): plt.scatter(centroids[j][0],centroids[j][1],marker='x',color='red',s=50,label='质心') plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
- 54
- 55
- 56
- 57
- 58
- 59
- 60
- 61
- 62
- 63
- 64
- 65
- 66
- 67
- 68
- 69
- 70
2. 最优聚类数目K的确定
2.1 手肘法–Elbow(经验方法)
我们知道k-means是以最小化样本与质点平方误差作为目标函数,将每个簇的质点与簇内样本点的平方距离误差和称为畸变程度(distortions),那么,对于一个簇,它的畸变程度越低,代表簇内成员越紧密,畸变程度越高,代表簇内结构越松散。 畸变程度会随着类别的增加而降低,但对于有一定区分度的数据,在达到某个临界点时畸变程度会得到极大改善,之后缓慢下降,这个临界点就可以考虑为聚类性能较好的点。
# 导包 import numpy as np import pandas as pd import matplotlib.pyplot as plt from scipy.spatial.distance import cdist from sklearn.cluster import KMeans # 1.利用pandas读入数据 data = pd.read_csv(data_path, header=None) # data_path换成你的需要聚类的数据所在路径,我的数据没有表头,所以设置header=None x = data[0: 150] # 设置需要聚类的数据列数,我的是150维 # 2.绘制手肘图 dispersions = [] for k in range(1, 20): # k:需要聚几类,按需修改,推荐至少10类 kmeans = KMeans(n_clusters=k, random_state=9) y_pred = kmeans.fit_predict(x) dispersions.append(sum(np.min(cdist(x, kmeans.cluster_centers_, 'euclidean'), axis=1))/x.shape[0]) print(dispersions) plt.plot(range(1, 20), dispersions, 'bx-') plt.xlabel('Number of Clusters (k)') plt.ylabel('Average Dispersion') plt.title('the Elbow Method') plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
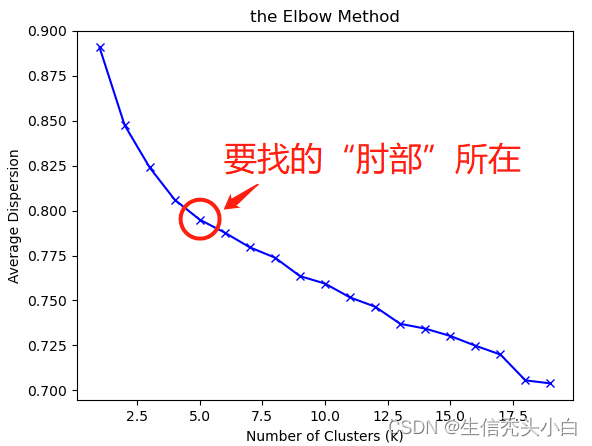
我的数据比较差,暂且认为在聚5类之前走势变换程度较大,聚5类以后走势变得平缓,所以如果按照手肘法,我的k值确定为5。
注意:手肘法通过人眼观察肘部图的走向来确定肘部位置进而来确定k值,主观因素很大,不推荐在写论文时使用,解释不清,以免审稿人刁难。
2.2 Silhouette Coefficient(轮廓系数,理论方法)
对于一个聚类任务,我们希望得到的类别簇中,簇内尽量紧密,簇间尽量远离,轮廓系数便是类的密集与分散程度的评价指标,公式表达为s=(b−a)/max(a,b),其中a簇样本到彼此间距离的均值,b代表样本到除自身所在簇外的最近簇的样本的均值,s取值在[-1, 1]之间,越接近1表示聚类效果越好
import pandas as pd import matplotlib.pyplot as plt from sklearn.cluster import KMeans from sklearn.metrics import silhouette_score data = pd.read_csv(data_path, header=None) x = data[0: 150] silhouette_scores = [] for k in range(2, 10): y_pred = KMeans(n_clusters=k, random_state=9).fit_predict(x) silhouette_scores.append(silhouette_score(x, y_pred)) print(silhouette_scores) plt.plot(range(2, 10), silhouette_scores, 'bx-') plt.xlabel('Number of Clusters (k)') plt.ylabel('silhouette') plt.title('the Silhouette Method') plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
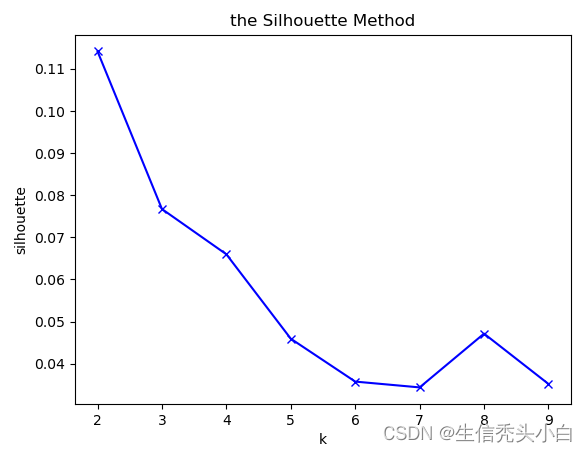
2.3 Calinski-Harabasz Criterion(卡林斯基-哈拉巴斯指标,CH值,理论方法)
定义就不多说了,直接上代码
import pandas as pd import matplotlib.pyplot as plt from sklearn import metrics from sklearn.cluster import KMeans data = pd.read_csv(data_path, header=None) x = data[0: 520] scores = [] for k in range(2, 20): y_pred = KMeans(n_clusters=k, random_state=9).fit_predict(x) score = metrics.calinski_harabasz_score(x, y_pred) scores.append(score) print(scores) plt.plot(range(2, 20), scores, 'bx-') plt.xlabel('k') plt.ylabel('the Calinski Carabasz Score') plt.title('the CH Method') plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
2.4 Davies-Bouldin Criterion(戴维斯-博尔丁指标,DB值,理论方法)
import pandas as pd import matplotlib.pyplot as plt from sklearn import metrics from sklearn.cluster import KMeans data = pd.read_csv(data_path, header=None) x = data[0: 520] scores = [] for k in range(2, 20): y_pred = KMeans(n_clusters=k, random_state=9).fit_predict(x) score = metrics.davies_bouldin_score(x, y_pred) scores.append(score) print(scores) plt.plot(range(2, 20), scores, 'bx-') plt.xlabel('k') plt.ylabel('the Davies Bouldin Score') plt.title('the DB Method') plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21




