- 1用Python的tkinter库做一个简单的打包作业的工具_tkinter打包成apk
- 2AI服务器的互连芯片技术创新和突破研究
- 3leetcode-227. 基本计算器 II_基本计算器 leetcode
- 4# Redis 入门到精通(四)-- linux 环境安装 redis
- 5网易笔试题_如果一根针从一个随机且相当高的高度掉到我们的游戏板上,这是一个画有平行线的表
- 6计算机毕业设计springboot的工程机械设备租赁系统wbv109【附源码+数据库+部署+LW】
- 7ofbiz安装
- 8树状数组(Binary Indexed Tree),看这一篇就够了
- 9LKA相关知识_lka 对eps的要求
- 10五种常用的异常值检测方法(均方差、箱形图、DBScan 聚类、孤立森林、Robust Random Cut Forest_使用均值和方差的异常检测算法
Science:纽约西奈山医学院房刚组定量分析真核生物DNA 6mA解析细菌污染的影响...
赞
踩

https://www.science.org/doi/10.1126/science.abe7489
DNA甲基化对于人类发育和多种疾病有着至关重要的影响。过去几十年的研究主要侧重于5mC(5-甲基胞嘧啶)以及其去甲基化的产物。2015年Cell 发表3篇研究,报道藻类【1】、果蝇【2】和线虫【3】的基因组中具有6mA修饰。2016年Nature发表研究报道6mA修饰在小鼠胚胎干细胞中的功能【4】。随后一系列研究报道6mA在多种植物和动物中广泛存在,并在多种人类疾病中起到作用。这些研究虽然对于表观遗传学和表观组学的研究带来了新的机遇,但同时也提出了新的挑战。过去的几年里,多项研究报道了真核生物6mA研究中存在的一些质疑,比如说6mA抗体的非特异性【5】,细菌污染【5, 6】, RNA对于抗体的干扰【7】,DNA中整合的RNA m6A修饰残基【8,9】等等。这些质疑对6mA在真核生物中的存在和功能引发了一系列的讨论与思考(详细内容请参考:『珍藏版』综述 | “路漫漫其修远兮,吾将上下而求索”,DNA 6mA修饰研究全纪录)。
2022年,2月4日纽约西奈山医学院房刚团队在Science发表长文: Critical assessment of DNA adenine methylation in eukaryotes using quantitative deconvolution【10】,论文对DNA 6mA这一重要生物学问题持续探究,报道了使用“宏表观组”新方法,定量分析真核生物DNA 6mA并评估细菌污染的影响。作者认为目前真核生物DNA 6mA研究的最大挑战是缺少一个既能准确定量又能精确测序的分析技术。已有的方法中,质谱的定量分析非常灵敏,但是它无法有效分析细菌污染的影响。基于6mA抗体的共免疫沉淀的测序方法(DIP-seq)虽然可以有针对性的分析真核生物基因组,但是其无法进行6mA定量分析(同时,抗体的非特异性也为DIP-seq的分析带来了困扰【5】)。基于限制性内切酶的测序方法的只适用于特定的DNA基序(motif),因此无法提供全面的定量分析。三代测序虽然可以检测DNA中的 6mA修饰,但是现有的方法主要适用于细菌和少量6mA含量高的单细胞真核生物【5, 11-13】,使用这些方法在大部分真核生物中检测DNA修饰需要非常谨慎。这篇Science文章中有详细讨论相关的技术细节。

为了有效处理上述挑战,文章利用宏基因组分析的策略开发了一个“宏表观组”方法6mASCOPE。其基本原理有三个方面:1)开发不依赖参考基因组的6mA分析方法,对样本中所有测序得到的DNA分子(200-400bp)逐一进行6mA分析,而不是只针对于感兴趣的真核生物基因组,从而包括了可能的细菌污染;2)获取三代测序底层数据并深入分析单个DNA分子信号以尽量减少6mA分析的背景噪音;3)利用机器学习算法对样本中不同物种、可能的细菌污染以及不同基因组区域进行6mA的分组定量。(图1)
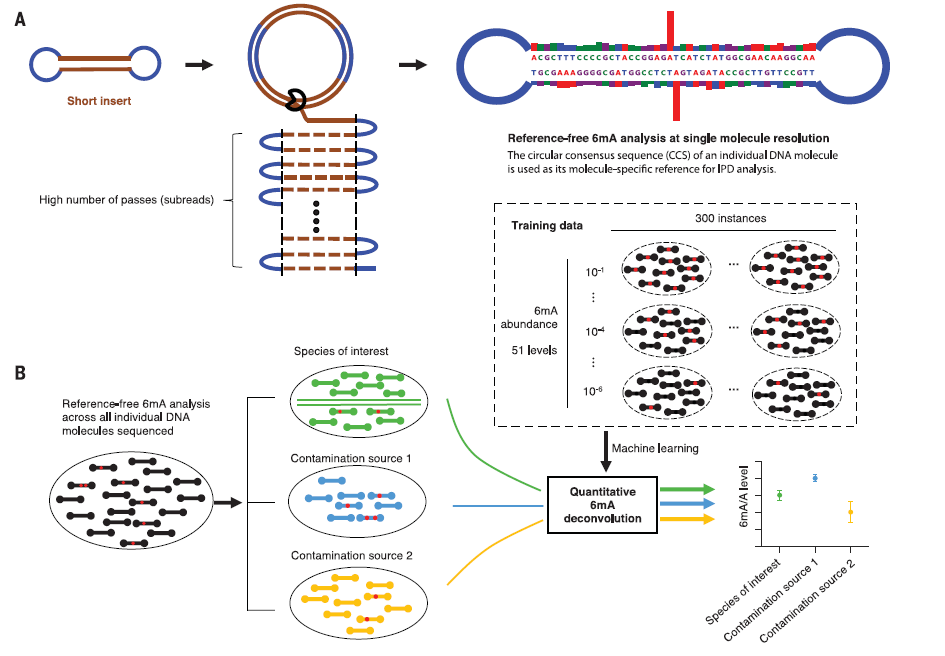
图1:6mASCOPE“宏表观组”方法设计的基本原理
文章首先对6mASCOPE的方法进行了全面评测。通过实验混合多物种DNA,模拟获取大跨度的6mA/A水平的样本(丰度10-2至10-6) 。与独立的质谱数据对比,确认了6mASCOPE可以进行可靠的6mA定量分析。然后,作者利用6mASCOPE分析了两个单细胞真核生物:莱茵衣藻 (Chlamydomonas reinhardtii) 和四膜虫 (Tetrahymena thermophila)。已有的研究发现6mA在这两个物种中的含量非常高(~10-2),并富集在转录起始位点附近的几个核小体间区【2】,但是对于6mA的精确定位尚不明确。文章作者利用6mASCOPE发现C. reinhardtii 中的6mA主要富集在核小体的边缘,而T. thermophila中的6mA更多的在两个核小体间区的中间(图2)。
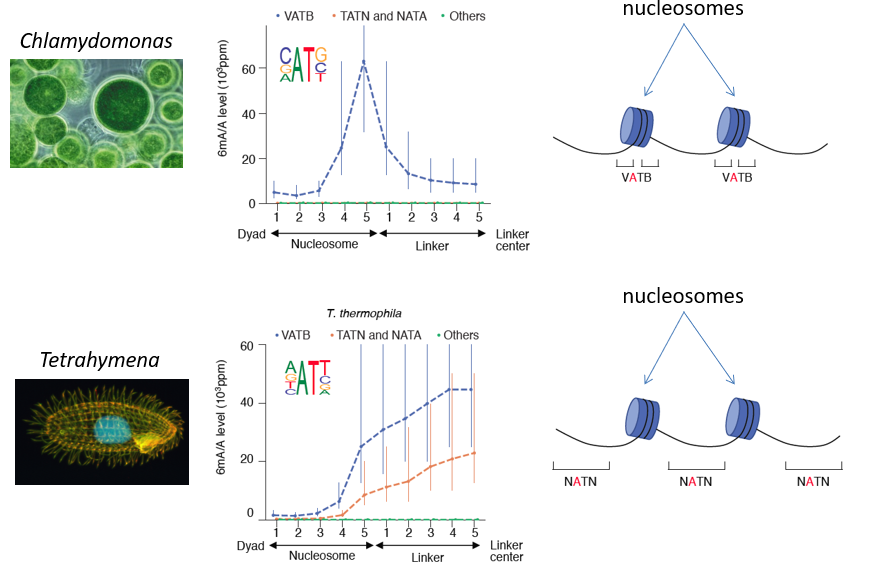
图2:6mASCOPE定量分析发现6mA在两个单细胞真核生物中定位的共同性和区别。
接下来,作者对果蝇和拟南芥的DNA样本进行分析。之前的研究报道果蝇早期胚胎基因组中具有很高的6mA【2】。利用6mASCOPE,作者发现果蝇早期胚胎样本有少量微生物DNA,包括果蝇的食物(S. cerevisiae)以及肠道菌群(Acetobacter、Lactobacillus等)。说明即使是非常谨慎的实验操作,胚胎样本DNA的提取过程中仍会带入果蝇的肠道菌群。对这些物种进行分组的6mA定量分析,作者发现虽然整体样品中6mA含量很高,来自于果蝇基因组的6mA含量却非常低。相比之下,果蝇肠道细菌的6mA含量极高(此点与预期相符,绝大多数细菌中存在高丰度的6mA)。结合样品中不同组分的含量以及单独的6mA定量,文章发现来自果蝇基因组的6mA只能解释样品中1.44%的6mA。该分析说明即使是非常少的细菌污染,仍然可能对真核生物中6mA的研究产生巨大的影响。
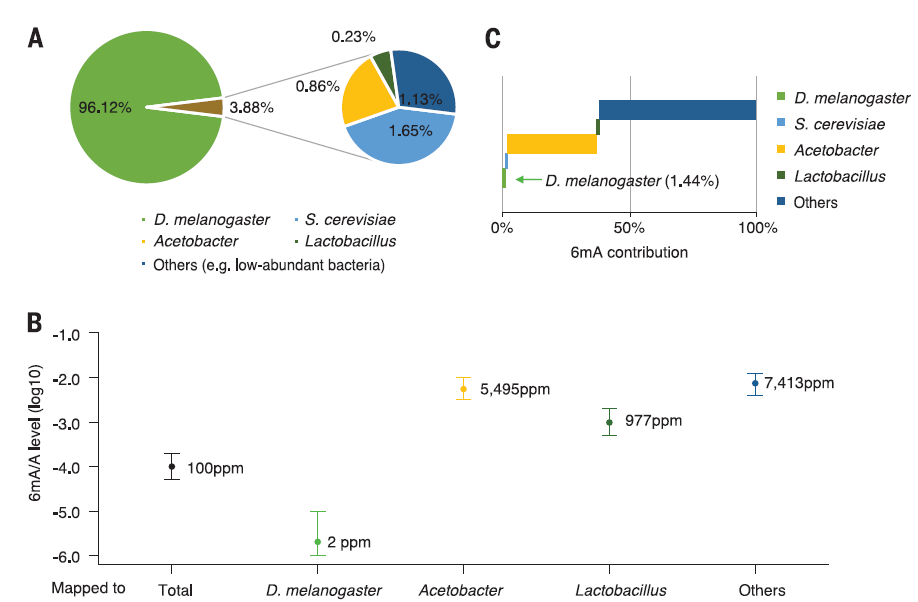
图3:6mASCOPE定量分析发现果蝇肠道菌群解释样品中绝大部分6mA。
同样地,作者用6mASCOPE在拟南芥的根部提取的基因组DNA发现了少量的土壤微生物。这些土壤微生物同样具有很高的6mA水平,而拟南芥本身基因组的6mA含量却非常低,只解释了样本中~4%的6mA。
最后,参考之前的研究报道的6mA含量较高的人类细胞或组织类型,文章选择了人类血液细胞和脑部胶质瘤样本进行6mASCOPE分析。虽然作者并未发现类似于果蝇或拟南芥样本的显著细菌污染,但是这些人类细胞中的6mA含量却远低于先前的研究【14,15】。说明6mA的丰度可能远低于之前估计的的丰度、或者可能在人的不同临床样本、疾病亚型、细胞亚型和组织部位存在差异。同时,文章利用实验证明遗传操作中经常使用的质粒也会携带大量的6mA,因为这些质粒是通过大肠杆菌培养得到的。文章特别指出,即使敲除了大肠杆菌中的6mA 甲基转移酶Dam,基因组中仍会有很高的6mA残留,这是因为Dam并不是大肠杆菌中唯一的6mA 甲基转移酶(Nat Rev Genet发表房刚组细菌表观组综述论文, Nat Methods | 房刚团队开发新方法检测多种细菌DNA甲基化,助力细菌表观遗传学研究和菌群分析【12】和2021年Nature Methods纳米孔技术方法【13】)。
作者在文章中强调了以下几点。第一,文章的分析结果仅针对该研究中使用到的样本,并不排除在该项研究外存在高丰度6mA的多细胞真核生物的可能性。但是,作者建议利用6mASCOPE对高丰度6mA的真核样本进行细菌污染的定量分析, 从而排除其影响,最终帮助更可靠的研究真核生物中的6mA (图四)。 第二,6mASCOPE侧重于对于单个物种基因组以及特定基因组区间的6mA整体定量,而非单个位点的6mA检测。对于单个位点的6mA检测,尤其是在6mA含量较低的样本中,研究人员需特别谨慎分析中可能存在的假阳性。虽然该项研究基于PacBio单分子测序技术, 但是纳米孔测序及二代测序分析6mA的方法同样面临假阳性的挑战,也需谨慎对待。第三,作者强调三代测序对6mA检测的特异性 (specificity) 需要谨慎考量,建议结合质谱数据和6mASCOPE分析进行真核生物中6mA的定量。第四,虽然文章侧重于6mA的分析,另外一种DNA甲基化,4mC的分析同样需要引起注意,因为很多细菌中也包含高丰度的4mC。更全面的分析与讨论请见文章原文。
图4: 示例6mASCOPE 通过定量分析去除细菌污染,准确解析真核生物中的6mA。
孔艺萌为该文章第一作者。另外,曹蕾、Gintaras Deikus、范宇和Edward Mead为文章并列第二作者。6mA质谱分析由中科院汪海林课题组完成。罗格斯大学医学院张雪松助力细菌和拟南芥的分析。文章致谢Emory大学医学院姚冰课题组提供果蝇样本。
最后,房刚课题组 (http://fanglab.bio) 正在招聘博士后参与第三代测序在病原菌、宏基因组、人类表观基因组和疾病研究中的应用。
原文链接:
http://doi.org/10.1126/science.abe7489
参考文献
[1] Fu Y, Luo G-Z, Chen K, Deng X, Yu M, Han D, Hao Z, Liu J, Lu X, Doré LC. 2015. N6 Methyldeoxyadenosine marks active transcription start sites in Chlamydomonas. Cell 161: 879–892.
[2] Zhang G, Huang H, Liu D, Cheng Y, Liu X, ZhangW, Yin R, Zhang D, Zhang P, Liu J. 2015. N6-Methyladenine DNA modification in Drosophila. Cell 161: 893–906.
[3] Greer EL, Blanco MA, Gu L, Sendinc E, Liu J, Aristizábal-Corrales D, Hsu C-H, Aravind L, He C, Shi Y. 2015. DNA methylation on N6-adenine in C. elegans. Cell 161: 868–878.
[4] T. P. Wu, T. Wang, M. G. Seetin, Y. Lai, S. Zhu, K. Lin, Y. Liu, S. D. Byrum, S. G. Mackintosh, M. Zhong, A. Tackett, G. Wang, L. S. Hon, G. Fang, J. A. Swenberg, A. Z. Xiao, DNA methylation on N6-adenine in mammalian embryonic stem cells. Nature. 532, 329–333 (2016)..
[5] A. Lentini, C. Lagerwall, S. Vikingsson, H. K. Mjoseng, K. Douvlataniotis, H. Vogt, H. Green, R. R. Meehan, M. Benson, C. E. Nestor, A reassessment of DNA-immunoprecipitation-based genomic profiling. Nat. Methods. 15, 499–504 (2018).
[6] Z. K. O’Brown, K. Boulias, J. Wang, S. Y. Wang, N. M. O’Brown, Z. Hao, H. Shibuya, P. E. Fady, Y. Shi, C. He, S. G. Megason, T. Liu, E. L. Greer, Sources of artifact in measurements of 6mA and 4mC abundance in eukaryotic genomic DNA. BMC Genomics. 20, 1–15 (2019).
[7] K. Douvlataniotis, M. Bensberg, A. Lentini, B. Gylemo, C. E. Nestor, No evidence for DNA N6-methyladenine in mammals. Sci. Adv. 6, 1–10 (2020).
[8] M. U. Musheev, A. Baumgärtner, L. Krebs, C. Niehrs, The origin of genomic N 6-methyl-deoxyadenosine in mammalian cells. Nat. Chem. Biol. 16, 630–634 (2020).
[9] X. Liu, W. Lai, Y. Li, S. Chen, B. Liu, N. Zhang, J. Mo, C. Lyu, J. Zheng, Y. R. Du, G. Jiang, G. L. Xu, H. Wang, N6-methyladenine is incorporated into mammalian genome by DNA polymerase. Cell Res., 3–6 (2020).
[10] Y. Kong, L. Cao, G. Deikus, Y. Fan, E. A. Mead, W. Lai, Y. Zhang, R. Yong, R. Sebra, H. Wang, X-S. Zhang, G. Fang, Criticalassessment of DNA adenine methylation across eukaryotes usingquantitative deconvolution, Science, 2022 10.1126/science.abe7489
[11] S. Zhu, J. Beaulaurier, G. Deikus, T. P. Wu, M. Strahl, Z. Hao, G. Luo, J. A. Gregory, A. Chess, C. He, A. Xiao, R. Sebra, E. E. Schadt, G. Fang, Mapping and characterizing N6-methyladenine in eukaryotic genomes using single-molecule real-time sequencing. Genome Res. 28, 1067–1078 (2018).
[12] J. Beaulaurier, E. E. Schadt, G. Fang, Deciphering bacterial epigenomes using modern sequencing technologies. Nat. Rev. Genet. 20, 157–172 (2019).
[13] A. Tourancheau, E. A. Mead, X. S. Zhang, G. Fang, Discovering multiple types of DNA methylation from bacteria and microbiome using nanopore sequencing. Nat. Methods. 18, 491–498 (2021).
[14] Q. Xie, T. P. Wu, R. C. Gimple, Z. Li, B. C. Prager, Q. Wu, Y. Yu, P. Wang, Y. Wang, D. U. Gorkin, C. Zhang, A. V. Dowiak, K. Lin, C. Zeng, Y. Sui, L. J. Y. Kim, T. E. Miller, L. Jiang, C. H. Lee, Z. Huang, X. Fang, K. Zhai, S. C. Mack, M. Sander, S. Bao, A. E. Kerstetter-Fogle, A. E. Sloan, A. Z. Xiao, J. N. Rich, N6-methyladenine DNA Modification in Glioblastoma. Cell. 175, 1228-1243.e20 (2018).
[15] C. Le Xiao, S. Zhu, M. He, D. Chen, Q. Zhang, Y. Chen, G. Yu, J. Liu, S. Q. Xie, F. Luo, Z. Liang, D. P. Wang, X. C. Bo, X. F. Gu, K. Wang, G. R. Yan, N 6 -Methyladenine DNA Modification in the Human Genome. Mol. Cell. 71, 306-318.e7 (2018).
(可上下滑动阅览)
猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
文献阅读 热心肠 SemanticScholar Geenmedical
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。PI请明示身份,另有海内外微生物相关PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。

学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
点击阅读原文,跳转最新文章目录阅读


