- 1ElasticSearch 高级查询语法Query DSL实战_{ "query": { "match_all": {} } }
- 2logback日志回滚原理
- 3C语言解析json--超详细,接口、参数全覆盖,一看就会_json c语言
- 4C#系列-Dapper.Contrib.Extensions应用实例(41)_dapperextensions 示例
- 5Uniapp-小程序简单的时间选择组件-年月日时分
- 6Fetch、Axios、Ajax 的理解_fetch、ajax、axios
- 7Java基础学习总结:IO之(四)缓冲流、内存流、标准输入输出流_java 文件流 缓冲区 buf 多大合适
- 8基于SqlSugar的开发框架循序渐进介绍(31)-- 在查询接口中实现多表联合和单表对象的统一处理_sqlsugar mergetable
- 9提升科研效率的关键:掌握3D科研绘图技能_科研绘图 柔性
- 10【深度学习】RuntimeError: Unable to find a valid cuDNN algorithm to run convolution
rpnnp的matlab,数学建模及机器学习算法(一):聚类-kmeans(Python及MATLAB实现,包括k值选取与聚类效果评估)...
赞
踩
一、聚类的概念
聚类分析是在数据中发现数据对象之间的关系,将数据进行分组,组内的相似性越大,组间的差别越大,则聚类效果越好。我们事先并不知道数据的正确结果(类标),通过聚类算法来发现和挖掘数据本身的结构信息,对数据进行分簇(分类)。聚类算法的目标是,簇内相似度高,簇间相似度低
二、基本的聚类分析算法
1. K均值(K-Means):
基于原型的、划分的距离技术,它试图发现用户指定个数(K)的簇。
2. 凝聚的层次距离:
思想是开始时,每个点都作为一个单点簇,然后,重复的合并两个最靠近的簇,直到尝试单个、包含所有点的簇。
3. DBSCAN:
一种基于密度的划分距离的算法,簇的个数有算法自动的确定,低密度中的点被视为噪声而忽略,因此其不产生完全聚类。
三、距离量度
不同的距离量度会对距离的结果产生影响,常见的距离量度如下所示:


四、K-Means
在聚类算法中K-Means算法是一种最流行的、使用最广泛的一种聚类算法,因为它的易于实现且计算效率也高。聚类算法的应用领域也是非常广泛的,包括不同类型的文档分类、音乐、电影、基于用户购买行为的分类、基于用户兴趣爱好来构建推荐系统等。
优点:易于实现
缺点:可能收敛于局部最小值,在大规模数据收敛慢
算法思想:
1 选择K个点作为初始质心2 repeat3 将每个点指派到最近的质心,形成K个簇4 重新计算每个簇的质心5 until 簇不发生变化或达到最大迭代次数
这里的重新计算每个簇的质心,更新过程是:首先找到与每个点距离最近的中心点,构成每个中心点划分的k个点集,然后对于每个点集,计算点的均值代替中心点。
如何计算是根据目标函数得来的,因此在开始时我们要考虑距离度量和目标函数。
考虑欧几里得距离的数据,使用误差平方和(Sum of the Squared Error,SSE)作为聚类的目标函数,两次运行K均值产生的两个不同的簇集,我们更喜欢SSE最小的那个:
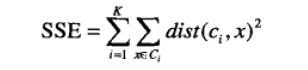
k表示k个聚类中心,ci表示第几个中心,dist表示的是欧几里得距离。
前面说的我们更新质心是让所有的点的平均值,这里就是SSE所决定的:

因此K-Means算法的实现步骤,主要分为四个步骤:
1、从样本集合中随机抽取k个样本点作为初始簇的中心。
2、将每个样本点划分到距离它最近的中心点所代表的簇中。
3、用各个簇中所有样本点的中心点代表簇的中心点。
4、重复2和3,直到簇的中心点不变或达到设定的迭代次数或达到设定的容错范围。
五、k-means代码实现
本文采用sklearn来实现一个k-means算法的应用,细节的底层实现可见文末第一个链接。
1.首先使用sklearn的数据集,数据集中包含150个随机生成的点,样本点分为三个不同的簇:
1 from sklearn.datasets importmake_blobs2 importmatplotlib.pyplot as plt3
4
5 if __name__ == "__main__":6 '''
7 n_samples:代表样本点的个数8 n_features:表示每个样本由两个特征组成9 center:表示样本点中心的个数(簇)10 cluster_std:表示每个样本簇方差的大小11 '''
12 x,y = make_blobs(n_samples=150,n_features=2,centers=3,13 cluster_std=0.5,shuffle=True,random_state=0)14 #绘点
15 plt.scatter(x[:,0],x[:,1],marker="o",color="blue")16 #以表格的形式显示
17 plt.grid()18 plt.show()
效果如下:
2.下面使用sklearn内置的KMeans算法来实现对上面样本点的聚类分析:
1 from sklearn.cluster importKMeans2 '''
3 n_clusters:设置簇的个数4 init:random表示使用Kmeans算法,默认是k-means++5 n_init:设置初始样本中心的个数6 max_iter:设置最大迭代次数7 tol:设置算法的容错范围SSE(簇内误平方差)8 '''
9 kmeans = KMeans(n_clusters=3,init="random",n_init=10,max_iter=300,10 tol=1e-04,random_state=0)11 y_km = kmeans.fit_predict(x)
六、k-means缺陷
k均值算法非常简单且使用广泛,但有一些缺点:
1. K值需要预先给定,属于预先知识,很多情况下K值的估计是非常困难的,因此会有后面的k值确定。
2. K-Means算法对初始选取的聚类中心点是敏感的,不同的随机种子点得到的聚类结果完全不同 。可能只能得到局部的最优解,而无法得到全局的最优解。
3. K-Means算法并不是很所有的数据类型。它不能处理非球形簇、不同尺寸和不同密度的簇。
4. 对离群点的数据进行聚类时,K-Means也有问题。
七、K-Means++
K-Means算法需要随机选择初始化的中心点,如果中心点选择不合适,可能会导致簇的效果不好或产生收敛速度慢等问题。解决这个问题一个比较合适的方法就是,在数据集上多次运行K-Means算法,根据簇内误差平方和(SSE)来选择性能最好的模型。除此之外,还可以通过K-Means++算法,让初始的中心点彼此的距离尽可能的远,相比K-Means算法,它能够产生更好的模型。
K-Means++有下面几个步骤组成:
1、初始化一个空的集合M,用于存储选定的k个中心点
2、从输入的样本中随机选择第一个中心点μ,并将其加入到集合M中
3、对于集合M之外的任意样本点x,通过计算找到与其距离最小的样本d(x,M)
4、使用加权概率分布来随机来随机选择下一个中心点μ
5、重复步骤2和3,直到选定k个中心点
6、基于选定的中心点执行k-means算法
使用sklearn来实现K-Means++,只需要将init参数设置为"k-means++",默认设置是"k-means++"。
1 km = KMeans(n_clusters=3,init="k-means++",n_init=10,max_iter=300,2 tol=1e-04,random_state=0)3 #y_km中保存了聚类的结果
4 y_km =km.fit_predict(x)5 #绘制不同簇的点
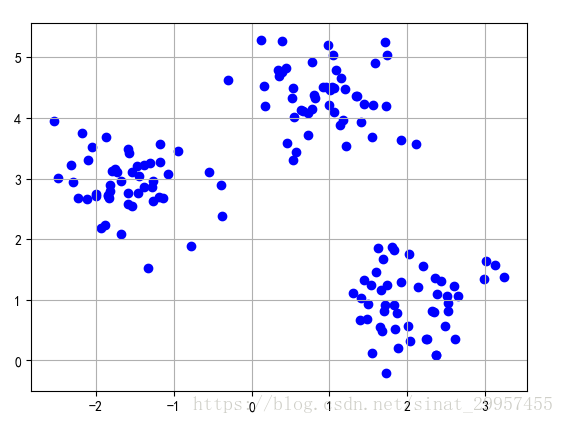
6 plt.scatter(x[y_km==0,0],x[y_km==0,1],s=50,c="orange",marker="o",label="cluster 1")7 plt.scatter(x[y_km==1,0],x[y_km==1,1],s=50,c="green",marker="s",label="cluster 2")8 plt.scatter(x[y_km==2,0],x[y_km==2,1],s=50,c="blue",marker="^",label="cluster 3")9 #绘制簇的中心点
10 plt.scatter(km.cluster_centers_[:,0],km.cluster_centers_[:,1],s=250,marker="*",c="red"
11 ,label="cluster center")12 plt.legend()13 plt.grid()14 plt.show()
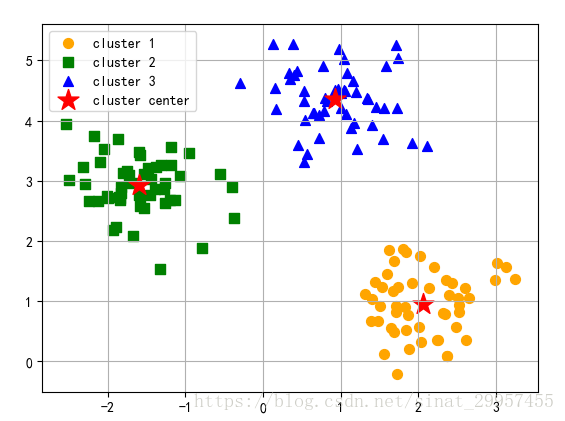
通过上面图可以发现k-means++的聚类效果还不错,簇的中心点,基本位于球心。
使用技巧:
1.在实际情况中使用k-means++算法可能会遇到,由于样本的维度太高无法可视化,从而无法设定样本的簇数。可视化问题可在聚类后显示前用pca对数据降维显示。
2.由于k-means算法是基于欧式距离来计算的,所以k-means算法对于数据的范围比较敏感,所以在使用k-means算法之前,需要先对数据进行标准化,保证k-means算法不受特征量纲的影响。
八、Mini Batch k-Means
在原始的K-means算法中,每一次的划分所有的样本都要参与运算,如果数据量非常大的话,这个时间是非常高的,因此有了一种分批处理的改进算法。
使用Mini Batch(分批处理)的方法对数据点之间的距离进行计算。
Mini Batch的好处:不必使用所有的数据样本,而是从不同类别的样本中抽取一部分样本来代表各自类型进行计算。n 由于计算样本量少,所以会相应的减少运行时间n 但另一方面抽样也必然会带来准确度的下降。
(其他关于k-means的优化还有很多,可参考链接或自行总结)
九、K值的确定
1.根据实际需要
2.肘部法则(Elbow Method)-见后文
3.轮廓系数(Silhouette Coefficient)-见后文
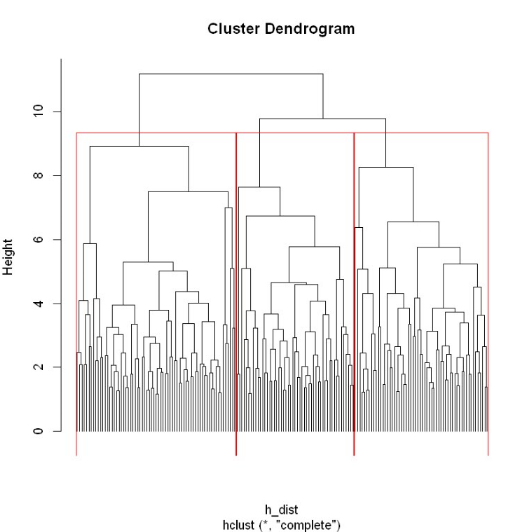
4.层次聚类
层次聚类是通过可视化然后人为去判断大致聚为几类,很明显在共同父节点的一颗子树可以被聚类为一个类
5.Canopy算法
肘部法则(Elbow Method)和轮廓系数(Silhouette Coefficient)来对k值进行最终的确定,但是这些方法都是属于“事后”判断的,而Canopy算法的作用就在于它是通过事先粗聚类的方式,为k-means算法确定初始聚类中心个数和聚类中心点。
与传统的聚类算法(比如K-Means)不同,Canopy聚类最大的特点是不需要事先指定k值(即clustering的个数),因此具有很大的实际应用价值。与其他聚类算法相比,Canopy聚类虽然精度较低,但其在速度上有很大优势,因此可以使用Canopy聚类先对数据进行“粗”聚类,得到k值,以及大致的k个中心点,再使用K-Means进行进一步“细”聚类。所以Canopy+K-Means这种形式聚类算法聚类效果良好。
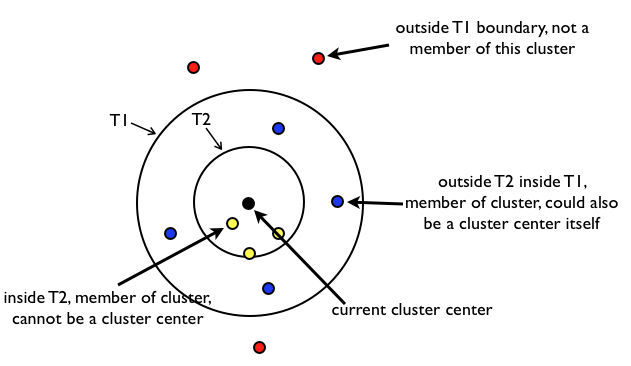
Canopy算法解析:
原始数据集合List按照一定的规则进行排序(这个规则是任意的,但是一旦确定就不再更改),初始距离阈值为T1、T2,且T1>T2(T1、T2的设定可以根据用户的需要,或者使用交叉验证获得)。
在List中随机挑选一个数据向量A,使用一个粗糙距离计算方式计算A与List中其他样本数据向量之间的距离d。
根据第2步中的距离d,把d小于T1的样本数据向量划到一个canopy中,同时把d小于T2的样本数据向量从候选中心向量名单(这里可以理解为就是List)中移除。
重复第2、3步,直到候选中心向量名单为空,即List为空,算法结束。
算法原理比较简单,就是对数据进行不断遍历,T2
canopy效果图如下:
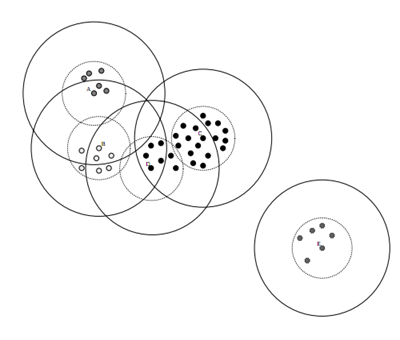
Canopy算法优势:
Kmeans对噪声抗干扰较弱,通过Canopy对比较小的NumPoint的Cluster直接去掉 有利于抗干扰。
Canopy选择出来的每个Canopy的centerPoint作为Kmeans比较科学。
只是针对每个Canopy的内容做Kmeans聚类,减少相似计算的数量。
Canopy算法缺点:
算法中 T1、T2(T2 < T1) 的确定问题(也有专门的算法去描述,但可以自己多次试错
Python实现:


1 #-*- coding: utf-8 -*-
2 importmath3 importrandom4 importnumpy as np5 importmatplotlib.pyplot as plt6
7 classCanopy:8 def __init__(self, dataset):9 self.dataset =dataset10 self.t1 =011 self.t2 =012
13 #设置初始阈值
14 defsetThreshold(self, t1, t2):15 if t1 >t2:16 self.t1 =t117 self.t2 =t218 else:19 print('t1 needs to be larger than t2!')20
21 #使用欧式距离进行距离的计算
22 defeuclideanDistance(self, vec1, vec2):23 return math.sqrt(((vec1 - vec2)**2).sum())24
25 #根据当前dataset的长度随机选择一个下标
26 defgetRandIndex(self):27 return random.randint(0, len(self.dataset) - 1)28
29 defclustering(self):30 if self.t1 ==0:31 print('Please set the threshold.')32 else:33 canopies = [] #用于存放最终归类结果
34 while len(self.dataset) !=0:35 rand_index =self.getRandIndex()36 current_center = self.dataset[rand_index] #随机获取一个中心点,定为P点
37 current_center_list = [] #初始化P点的canopy类容器
38 delete_list = [] #初始化P点的删除容器
39 self.dataset =np.delete(40 self.dataset, rand_index, 0) #删除随机选择的中心点P
41 for datum_j inrange(len(self.dataset)):42 datum =self.dataset[datum_j]43 distance =self.euclideanDistance(44 current_center, datum) #计算选取的中心点P到每个点之间的距离
45 if distance <46>
47 current_center_list.append(datum)48 if distance <49 delete_list.append>
50 #根据删除容器的下标,将元素从数据集中删除
51 self.dataset =np.delete(self.dataset, delete_list, 0)52 canopies.append((current_center, current_center_list))53 returncanopies54
55
56 defshowCanopy(canopies, dataset, t1, t2):57 fig =plt.figure()58 sc = fig.add_subplot(111)59 colors = ['brown', 'green', 'blue', 'y', 'r', 'tan', 'dodgerblue', 'deeppink', 'orangered', 'peru', 'blue', 'y', 'r',60 'gold', 'dimgray', 'darkorange', 'peru', 'blue', 'y', 'r', 'cyan', 'tan', 'orchid', 'peru', 'blue', 'y', 'r', 'sienna']61 markers = ['*', 'h', 'H', '+', 'o', '1', '2', '3', ',', 'v', 'H', '+', '1', '2', '^',62 '', '.', '4', 'H', '+', '1', '2', 's', 'p', 'x', 'D', 'd', '|', '_']63 for i inrange(len(canopies)):64 canopy =canopies[i]65 center =canopy[0]66 components = canopy[1]67 sc.plot(center[0], center[1], marker=markers[i],68 color=colors[i], markersize=10)69 t1_circle =plt.Circle(70 xy=(center[0], center[1]), radius=t1, color='dodgerblue', fill=False)71 t2_circle =plt.Circle(72 xy=(center[0], center[1]), radius=t2, color='skyblue', alpha=0.2)73 sc.add_artist(t1_circle)74 sc.add_artist(t2_circle)75 for component incomponents:76 sc.plot(component[0], component[1],77 marker=markers[i], color=colors[i], markersize=1.5)78 maxvalue =np.amax(dataset)79 minvalue =np.amin(dataset)80 plt.xlim(minvalue - t1, maxvalue +t1)81 plt.ylim(minvalue - t1, maxvalue +t1)82 plt.show()83
84
85 if __name__ == "__main__":86 dataset = np.random.rand(500, 2) #随机生成500个二维[0,1)平面点
87 t1 = 0.6
88 t2 = 0.4
89 gc =Canopy(dataset)90 gc.setThreshold(t1, t2)91 canopies =gc.clustering()92 print('Get %s initial centers.' %len(canopies))93 showCanopy(canopies, dataset, t1, t2)
View Code
6.间隔统计量 Gap Statistic
根据肘部法则选择最合适的K值有事并不是那么清晰,因此斯坦福大学的Robert等教授提出了Gap Statistic方法。
这里我们要继续使用上面的
。Gap Statistic的定义为:
这里
指的是
的期望。这个数值通常通过蒙特卡洛模拟产生,我们在样本里所在的矩形区域中(高维的话就是立方体区域)按照均匀分布随机地产生和原始样本数一样多的随机样本,并对这个随机样本做K-Means,从而得到一个
。如此往复多次,通常20次,我们可以得到20个
。对这20个数值求平均值,就得到了
的近似值。最终可以计算Gap Statisitc。而Gap statistic取得最大值所对应的K就是最佳的K。
Gap Statistic的基本思路是:引入参考的测值,这个参考值可以有Monte Carlo采样的方法获得。
B是sampling的次数。为了修正MC带来的误差,我们计算sk也即标准差来矫正Gap Statistic。
选择满足
的最小的k作为最优的聚类个数。
Python实现:


1 importscipy2 from scipy.spatial.distance importeuclidean3 from sklearn.cluster importKMeans as k_means4
5 dst =euclidean6
7 k_means_args_dict ={8 'n_clusters': 0,9 #drastically saves convergence time
10 'init': 'k-means++',11 'max_iter': 100,12 'n_init': 1,13 'verbose': False,14 #'n_jobs':8
15 }16
17
18 def gap(data, refs=None, nrefs=20, ks=range(1, 11)):19 """
20 I: NumPy array, reference matrix, number of reference boxes, number of clusters to test21 O: Gaps NumPy array, Ks input list22
23 Give the list of k-values for which you want to compute the statistic in ks. By Gap Statistic24 from Tibshirani, Walther.25 """
26 shape =data.shape27
28 if notrefs:29 tops = data.max(axis=0)30 bottoms = data.min(axis=0)31 dists = scipy.matrix(scipy.diag(tops -bottoms))32 rands = scipy.random.random_sample(size=(shape[0], shape[1], nrefs))33 for i inrange(nrefs):34 rands[:, :, i] = rands[:, :, i] * dists +bottoms35 else:36 rands =refs37
38 gaps =scipy.zeros((len(ks),))39
40 for (i, k) inenumerate(ks):41 k_means_args_dict['n_clusters'] =k42 kmeans = k_means(**k_means_args_dict)43 kmeans.fit(data)44 (cluster_centers, point_labels) =kmeans.cluster_centers_, kmeans.labels_45
46 disp =sum(47 [dst(data[current_row_index, :], cluster_centers[point_labels[current_row_index], :]) forcurrent_row_index48 inrange(shape[0])])49
50 refdisps = scipy.zeros((rands.shape[2],))51
52 for j in range(rands.shape[2]):53 kmeans = k_means(**k_means_args_dict)54 kmeans.fit(rands[:, :, j])55 (cluster_centers, point_labels) =kmeans.cluster_centers_, kmeans.labels_56 refdisps[j] =sum(57 [dst(rands[current_row_index, :, j], cluster_centers[point_labels[current_row_index], :]) for
58 current_row_index inrange(shape[0])])59
60 #let k be the index of the array 'gaps'
61 gaps[i] = scipy.mean(scipy.log(refdisps)) -scipy.log(disp)62
63 return ks, gaps
View Code
十、聚类效果评估
1.簇内误方差(SSE)
在对簇的划分中,我们就使用了SSE作为目标函数来划分簇。当KMeans算法训练完成后,我们可以通过使用inertia属性来获取簇内的误方差,不需要再次进行计算。
1 #用来存放设置不同簇数时的SSE值
2 distortions =[]3 for i in range(1,11):4 km = KMeans(n_clusters=i,init="k-means++",n_init=10,max_iter=300,tol=1e-4,random_state=0)5 km.fit(x)6 #获取K-means算法的SSE
7 distortions.append(km.inertia_)8 #绘制曲线
9 plt.plot(range(1,11),distortions,marker="o")10 plt.xlabel("簇数量")11 plt.ylabel("簇内误方差(SSE)")12 plt.show()
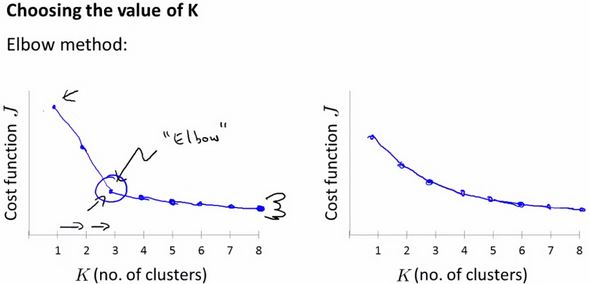
可以使用图形工具肘方法,根据簇的数量来可视化簇内误方差。通过图形可以直观的观察到k对于簇内误方差的影响。也可以用来确定K值。
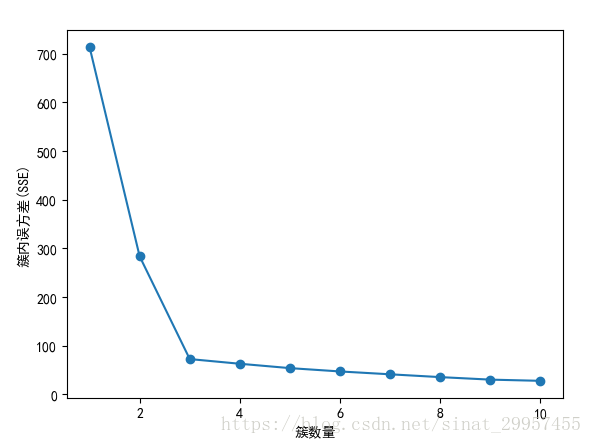
通过上图可以发现,当簇数量为3的时候出现了肘型,这说明k取3是一个不错的选择。但不一定所有的问题都能用肘部法则来解决,如下图右图中,肘部不明显。因此肘部法则只是一种可尝试的方法。
2、轮廓图定量分析聚类质量
轮廓分析(silhouette analysis),使用图形工具来度量簇中样本的聚集程度,除k-means之外也适用于其他的聚类算法。通过三个步骤可以计算出当个样本的轮廓系数(silhouette coefficient):
1、将样本x与簇内的其他点之间的平均距离作为簇内的内聚度a
2、将样本x与最近簇中所有点之间的平均距离看作是与最近簇的分离度b
3、将簇的分离度与簇内聚度之差除以二者中比较大的数得到轮廓系数,计算公式如下
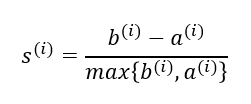
轮廓系数的取值在-1到1之间。当簇内聚度与分度离相等时,轮廓系数为0。当b>>a时,轮廓系数近似取到1,此时模型的性能最佳。
1 km = KMeans(n_clusters=3,init="k-means++",n_init=10,max_iter=300,tol=1e-4,random_state=0)2 y_km =km.fit_predict(x)3 importnumpy as np4 from matplotlib importcm5 from sklearn.metrics importsilhouette_samples6 #获取簇的标号
7 cluster_labels =np.unique(y_km)8 #获取簇的个数
9 n_clusters =cluster_labels.shape[0]10 #基于欧式距离计算轮廓系数
11 silhoutte_vals = silhouette_samples(x,y_km,metric="euclidean")12 #设置y坐标的起始位置
13 y_ax_lower,y_ax_upper=0,014 yticks=[]15 for i,c inenumerate(cluster_labels):16 #获取不同簇的轮廓系数
17 c_silhouette_vals = silhoutte_vals[y_km ==c]18 #对簇中样本的轮廓系数由小到大进行排序
19 c_silhouette_vals.sort()20 #获取到簇中轮廓系数的个数
21 y_ax_upper +=len(c_silhouette_vals)22 #获取不同颜色
23 color = cm.jet(i /n_clusters)24 #绘制水平直方图
25 plt.barh(range(y_ax_lower,y_ax_upper),c_silhouette_vals,26 height=1.0,edgecolor="none",color=color)27 #获取显示y轴刻度的位置
28 yticks.append((y_ax_lower+y_ax_upper) / 2)29 #下一个y轴的起点位置
30 y_ax_lower +=len(c_silhouette_vals)31 #获取轮廓系数的平均值
32 silhouette_avg =np.mean(silhoutte_vals)33 #绘制一条平行y轴的轮廓系数平均值的虚线
34 plt.axvline(silhouette_avg,color="red",linestyle="--")35 #设置y轴显示的刻度
36 plt.yticks(yticks,cluster_labels+1)37 plt.ylabel("簇")38 plt.xlabel("轮廓系数")39 plt.show()
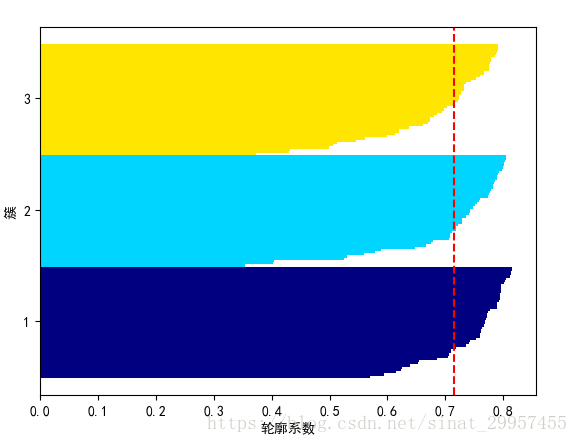
通过轮廓图,我们能够看出样本的簇数以及判断样本中是否包含异常值。为了评价聚类模型的性能,可以通过评价轮廓系数,也就是图中的红色虚线进行评价。
类似SSE,也可以做出不同k值下的效果图:
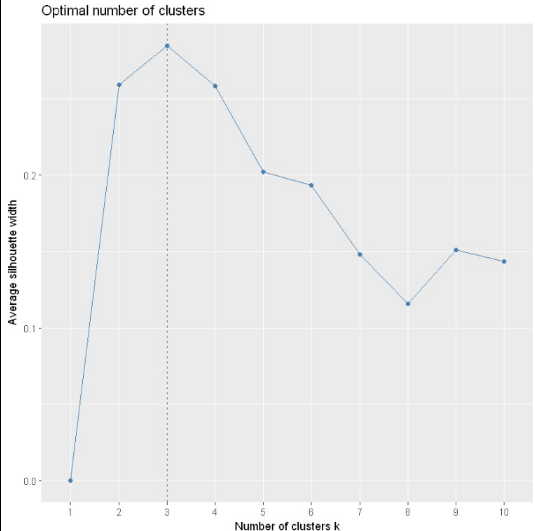
可以看到也是在聚类数为3时轮廓系数达到了峰值,所以最佳聚类数为3
十一、MATLAB实现
最后针对使用MATLAB的给出代码,细节与上文类似:
代码一:
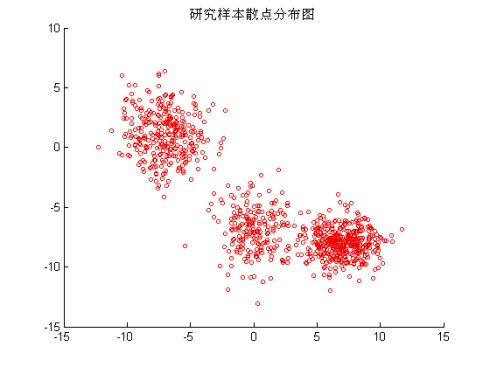
生成随机二维分布图形,三个中心
1 % 使用高斯分布(正态分布)2 % 随机生成3个中心以及标准差3 s = rng(5,'v5normal');
4 mu = round((rand(3,2)-0.5)*19)+1;5 sigma = round(rand(3,2)*40)/10+1;6 X = [mvnrnd(mu(1,:),sigma(1,:),200); ...7 mvnrnd(mu(2,:),sigma(2,:),300); ...8 mvnrnd(mu(3,:),sigma(3,:),400)];9 % 作图10 P1 =figure;clf;11 scatter(X(:,1),X(:,2),10,'ro');
12 title('研究样本散点分布图')
分层聚类:
1 eucD = pdist(X,'euclidean');
2 clustTreeEuc = linkage(eucD,'average');
3 cophenet(clustTreeEuc,eucD);4
5 P3 =figure;clf;6 [h,nodes] = dendrogram(clustTreeEuc,20);7 set(gca,'TickDir','out','TickLength',[.002 0],'XTickLabel',[]);

调用k-means,分成三类
1 [cidx3,cmeans3,sumd3,D3] = kmeans(X,3,'dist','sqEuclidean');
2 P4 =figure;clf;3 [silh3,h3] = silhouette(X,cidx3,'sqeuclidean');
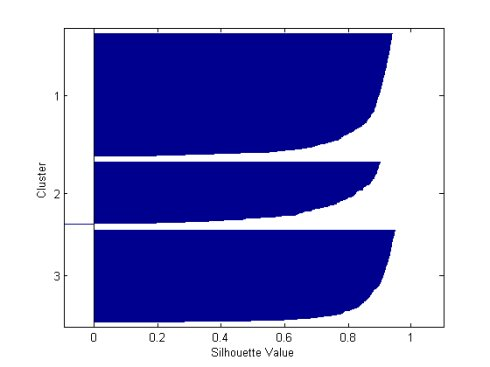
结果显示:
1 P5 =figure;clf2 ptsymb = {'bo','ro','go',',mo','c+'};
3 MarkFace = {[0 0 1],[.8 0 0],[0 .5 0]};4 hold on
5 for i =1:3
6 clust = find(cidx3 ==i);7 plot(X(clust,1),X(clust,2),ptsymb{i},'MarkerSize',3,'MarkerFace',MarkFace{i},'MarkerEdgeColor','black');
8 plot(cmeans3(i,1),cmeans3(i,2),ptsymb{i},'MarkerSize',10,'MarkerFace',MarkFace{i});
9 end
10 hold off
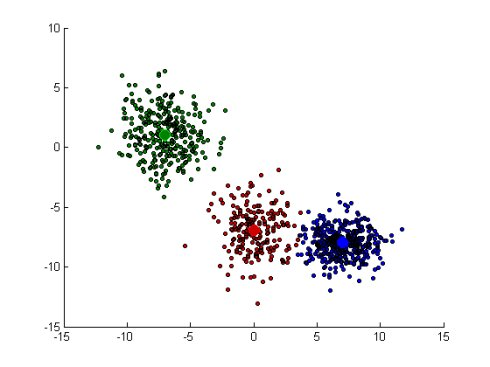
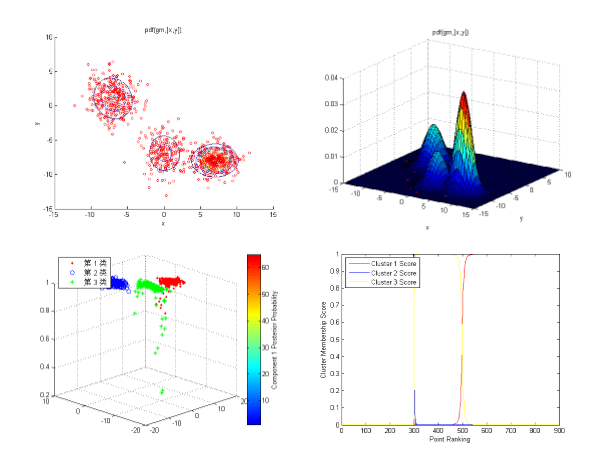
分别用等高线、分布图、热能图和概率图展示结果


1 % 等高线2 options = statset('Display','off');
3 gm = gmdistribution.fit(X,3,'Options',options);
4
5 P6 =figure;clf6 scatter(X(:,1),X(:,2),10,'ro');
7 hold on
8 ezcontour(@(x,y) pdf(gm,[x,y]),[-15 15],[-15 10]);9 hold off
10
11 P7 =figure;clf12 scatter(X(:,1),X(:,2),10,'ro');
13 hold on
14 ezsurf(@(x,y) pdf(gm,[x,y]),[-15 15],[-15 10]);15 hold off
16 view(33,24)17
18
19
20 热能图21 cluster1 = (cidx3 == 1);22 cluster3 = (cidx3 == 2);23 % 通过观察,K均值方法的第二类是gm的第三类24 cluster2 = (cidx3 == 3);25 % 计算分类概率26 P =posterior(gm,X);27 P8 =figure;clf28 plot3(X(cluster1,1),X(cluster1,2),P(cluster1,1),'r.')
29 grid on;hold on
30 plot3(X(cluster2,1),X(cluster2,2),P(cluster2,2),'bo')
31 plot3(X(cluster3,1),X(cluster3,2),P(cluster3,3),'g*')
32 legend('第 1 类','第 2 类','第 3 类','Location','NW')
33 clrmap = jet(80); colormap(clrmap(9:72,:))34 ylabel(colorbar,'Component 1 Posterior Probability')
35 view(-45,20);36 % 第三类点部分概率值较低,可能需要其他数据来进行分析。37
38 % 概率图39 P9 =figure;clf40 [~,order] = sort(P(:,1));41 plot(1:size(X,1),P(order,1),'r-',1:size(X,1),P(order,2),'b-',1:size(X,1),P(order,3),'y-');
42 legend({'Cluster 1 Score' 'Cluster 2 Score' 'Cluster 3 Score'},'location','NW');
43 ylabel('Cluster Membership Score');
44 xlabel('Point Ranking');
View Code
AIC准则寻找最优分类
1 AIC = zeros(1,4);2 NlogL =AIC;3 GM = cell(1,4);4 for k = 1:4
5 GM{k} =gmdistribution.fit(X,k);6 AIC(k)=GM{k}.AIC;7 NlogL(k) =GM{k}.NlogL;8 end
9 [minAIC,numComponents] = min(AIC);
代码二(简单版):
%随机初始化中心点,可以随机取数据集中的任意k个点
function centroids=kMeansInitCentroids(X, K)
centroids= zeros(K, size(X, 2));
randidx= randperm(size(X, 1));
centroids= X(randidx(1: K), :);
end%对每个点,找到其所属的中心点,即距离其最近的中心点。 返回一个向量,为每个点对应的中心点id。
function idx=findClosestCentroids(X, centroids)
K= size(centroids, 1);for i = 1 : size(X, 1)
min_dis= +inf;for j = 1: K
now_dis= sum((X(i, :) - centroids(j, :)).^2);if now_dis
min_dis=now_dis;
idx(i)=j;
end
end
end
end%用每个中心点统领的那类点的均值,更新中心点。
function centroids=computeCentroids(X, idx, K)
[m n]=size(X);
cnt=zeros(K, n);for i = 1: m
cnt(idx(i), :)= cnt(idx(i), :) + 1;
centroids(idx(i), :)= centroids(idx(i), :) +X(i, :);
end
cnt= 1 ./cnt;
centroids= cnt .*centroids;
end
centroids=kMeansInitCentroids(X, k);for iter = 1: iterations
idx=findClosestCentroids(X, centroids);
centroids=computeMeans(X, idx, K);
end
十二、参考链接:
49>46>

