- 1Linux环境中的git
- 2Android 9.0 系统systemui状态栏下拉左滑显示通知栏右滑显示控制中心模块的流程分析_android 通知栏 左右
- 3double和float的精度和取值范围_double范围
- 4CNN卷积神经网络学习笔记(特征提取)_cnn提取三维数据特征
- 5VGG16网络模型_vgg16模型图
- 6机器学习系列(13)_PCA对图像数据集的降维_02_图片 pca降维
- 7unity3d功能脚本大全_gui.drawtexture(new rect(0, 0, screen.width, scree
- 85个ai工具导航网站,最新最全的ai网址_ai产品行业看哪些网站
- 9Exit 0、exit 1、exit -1 的区别_exit(0)和exit(-1)
- 10Oracle listener lsnrctl_lsnrctl 启动特定实例
多目标优化
赞
踩
1. 多目标优化概念
相对单目标优化的任何两解都可以依据单一目标比较其好坏,可以得出没有争议的最优解。
多目标优化是指在某个场景下需要达到多个目标,但在现实情况下,目标间一般存在冲突,无法同时达到最优,一个目标的优化以其他目标劣化为代价,因此很难出现唯一最优解,取而代之的是在他们中间做出协调和折衷处理,使总体的目标尽可能的达到最优。
多目标优化问题主要由目标函数以及施加于决策变量的约束条件的数学模型表示。
2. 传统多目标优化方法
2.1 线性规划
将多目标按权重线性组合转化为单目标,解集由权重的变化产生。
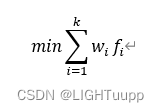
2.2 折衷规划
综合优化兼顾各个目标的性能要求,根据各个单目标的理想点评估,得到最接近理想点的非支配解。
2.3 ɛ-约束
从所有的k个目标中选择一个作为优化的目标,剩余的( k - 1)个目标则通过加界限的方式转化为约束条件。对于最小化的目标,加入上界作为限制条件,对于最大化的目标则加入下界作为限制条件。
3. 进化算法求解多目标问题
3.1 遗传算法GA
遗传算法 ( Genetic Algorithm ) 是受自然进化理论启发的一系列搜索算法。通过模仿自然选择和繁殖的过程,遗传算法可以为涉及搜索,优化和学习的各种问题提供高质量的解决方案。
遗传算法具有以下概念:基因型(Genotype)、种群(Population)、适应度函数(又称目标函数)(Fitness function)、遗传算子(选择(Selection)、交叉(Crossover)、变异(Mutation))
3.2 粒子群算法PSO
粒子群优化算法 ( Particle Swarm Optimization )是一种源于对鸟群捕食行为的研究而发明的进化计算技术,具有全局最优解和局部最优解,是基于群体协作的迭代优化算法。
PSO初始化为一群随机粒子(随机解),然后通过迭代找到最优解,在每一次迭代中,粒子通过跟踪两个“极值”来更新自己。第一个就是粒子本身所找到的最优解,这个解叫做个体极值pBest,另一个极值是整个种群找到的最优解,这个极值是全局极值gBest。另外也可以不用整个种群而只是用其中一部分最优粒子的邻居,那么在所有邻居中的极值就是局部极值。
3.3 模拟退火SA
模拟退火算法来源于固体退火原理,是一种基于概率的算法,将固体加温至充分高,再让其徐徐冷却,加温时,固体内部粒子随温升变为无序状,内能增大,而徐徐冷却时粒子渐趋有序,在每个温度都达到平衡态 ,最后在常温 时达到基态,内能减为最小。
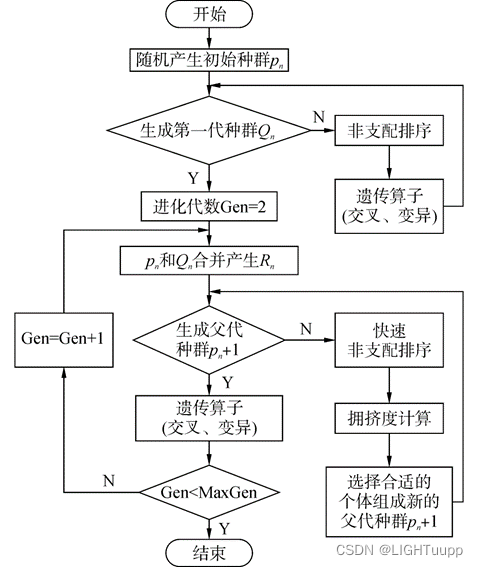
4. NSGA-Ⅱ
NSGA-Ⅱ算法是带有精英保留策略的快速非支配多目标优化算法,是一种生成Pareto前沿的遗传算法。
4.1 Perato支配
支配:由于多个目标函数的存在、对于可行解(满足优化条件的解),无法利用传统的大小关系比较进行优劣关系比较和排序,定义多目标情况下的个体间关系。
Perato支配:又称Perato占优,对于两个可行解 I1 和 I2,对所有目标而言,I1 均优于 I2,则我们称 I1 支配I2。
4.2 Perato最优解
Perato最优解又称非支配解,对于解A而言,在变量空间中找不到其他的解能够优于解A (注意这里的优于一定要两个目标函数值都优于A对应的函数值),那么解A就是Perato最优解。
4.3 Perato前沿
Perato最优解经目标函数映射构成了该优化问题的Pareto前沿面。
4.4 非支配排序
假设种群大小为P,遍历整个种群,计算每个个体p的被支配个数n_p和该个体支配的解的集合S_p这两个参数。对整个种群分层,形成各支配等级。
- # 非支配排序
- def non_domination_sort(population, pop_size, f_num, x_num):
- #non_domination_sort 初始种群的非支配排序和计算拥挤度
- #初始化pareto等级为1
- pareto_rank=1
- fronts={}#初始化一个字典
- fronts[pareto_rank]=[] # 前沿
- pn={}
- ps={}
- for i in range(pop_size):
- #计算出种群中每个个体p的被支配个数n和该个体支配的解的集合s
- pn[i]=0#被支配个体数目n
- ps[i]=[]#支配解的集合s
- for j in range(pop_size):
- less=0
- equal=0
- greater=0
- for k in range(f_num):
- if (population[i].fitness[k]<population[j].fitness[k]):
- less=less+1
- elif (population[i].fitness[k] == population[j].fitness[k]):
- equal=equal+1
- else:
- greater=greater+1
- if (less==0 and equal!=f_num):
- pn[i]=pn[i]+1
- elif (greater==0 and equal!=f_num):
- ps[i].append(j)
- if (pn[i]==0):
- # Individual对象 有四个属性 chromosome,nnd,paretorank,f
- population[i].paretorank=1#储存个体的等级信息
- fronts[pareto_rank].append(i)
- #求pareto等级为pareto_rank+1的个体
- while (len(fronts[pareto_rank])!=0):
- temp=[]
- for i in range(len(fronts[pareto_rank])):
- if (len(ps[fronts[pareto_rank][i]])!=0):
- for j in range(len(ps[fronts[pareto_rank][i]])):
- pn[ps[fronts[pareto_rank][i]][j]]=pn[ps[fronts[pareto_rank][i]][j]]-1#nl=nl-1
- if pn[ps[fronts[pareto_rank][i]][j]]==0:
- population[ps[fronts[pareto_rank][i]][j]].paretorank= pareto_rank + 1#储存个体的等级信息
- temp.append(ps[fronts[pareto_rank][i]][j])
- pareto_rank=pareto_rank+1
- fronts[pareto_rank]=temp
- return fronts, population

4.3 拥挤度
为了使得到的解在目标空间中更加均匀,这里引入了拥挤度概念,从二目标优化问题来看,就像是该个体在目标空间所能生成的最大外接矩形(该矩形不能触碰目标空间其他的点)的边长之和。
- # 拥挤度排序
- def crowding_distance_sort( population_non,fronts, f_num, x_num):
- #计算拥挤度
- ppp=[]
- #按照pareto等级对种群中的个体进行排序
- temp=sorted(population_non, key=lambda Individual:Individual.paretorank)#按照pareto等级排序后种群
- index1=[]
- for i in range(len(temp)):
- index1.append(population_non.index(temp[i]))
- #对于每个等级的个体开始计算拥挤度
- current_index = 0
- for pareto_rank in range(len(fronts) - 1):#计算F的循环时多了一次空,所以减掉,由于pareto从1开始,再减一次
- nd=np.zeros(len(fronts[pareto_rank + 1]))#拥挤度初始化为0
- y=[]#储存当前处理的等级的个体
- yF=np.zeros((len(fronts[pareto_rank + 1]), f_num))
- for i in range(len(fronts[pareto_rank + 1])):
- y.append(temp[current_index + i])
- current_index=current_index + i + 1
- #对于每一个目标函数fm
- for i in range(f_num):
- #根据该目标函数值对该等级的个体进行排序
- index_objective=[]#通过目标函数排序后的个体索引
- objective_sort=sorted(y, key=lambda Individual:Individual.fitness[i])#通过目标函数排序后的个体
- for j in range(len(objective_sort)):
- index_objective.append(y.index(objective_sort[j]))
- #记fmax为最大值,fmin为最小值
- fmin=objective_sort[0].fitness[i]
- fmax=objective_sort[len(objective_sort)-1].fitness[i]
- #对排序后的两个边界拥挤度设为1d和nd设为无穷
- yF[index_objective[0]][i]=float("inf")
- yF[index_objective[len(index_objective)-1]][i]=float("inf")
- #计算nd=nd+(fm(i+1)-fm(i-1))/(fmax-fmin)
- j=1
- while (j<=(len(index_objective)-2)):
- pre_f=objective_sort[j-1].fitness[i]
- next_f=objective_sort[j+1].fitness[i]
- if (fmax-fmin==0):
- yF[index_objective[j]][i]=float("inf")
- else:
- yF[index_objective[j]][i]=float((next_f-pre_f)/(fmax-fmin))
- j=j+1
- #多个目标函数拥挤度求和
- nd=np.sum(yF,axis=1)
- for i in range(len(y)):
- y[i].nnd=nd[i]
- ppp.append(y[i])
- return ppp

4.3 精英保留
精英保留策略可以避免最优个体不会因为杂交操作而被破坏。遗传算法中的基因,并不一定真实地反映了待求解问题的本质,因此各个基因之间未必就相互独立,如果只是简单地进行杂交,很可能把较好的组合给破坏了。对遗传算法来说,能否收敛到全局最优解是其首要问题。
把群体在进化过程中迄今出现的精英个体(适应度值最高的个体)不进行配对交叉而直接复制到下一代中,具有精英保留的标准遗传算法是全局收敛的。
- def elitism(N,combine_chromo2,f_num,x_num):
- chromo=[]
- index1=0
- index2=0
- #根据pareto等级从高到低进行排序
- chromo_rank=sorted(combine_chromo2, key=lambda Individual:Individual.paretorank)
- flag=chromo_rank[N-1].paretorank
- for i in range(len(chromo_rank)):
- if (chromo_rank[i].paretorank==(flag)):
- index1=i
- break
- else:
- chromo.append(chromo_rank[i])
- for i in range(len(chromo_rank)):
- if (chromo_rank[i].paretorank==(flag + 1)):
- index2=i
- break
- temp=[]
- aaa=index1
- if (index2==0):
- index2=len(chromo_rank)
- while (aaa<index2):
- temp.append(chromo_rank[aaa])
- aaa=aaa+1
- temp_crowd=sorted(temp, key=lambda Individual:Individual.paretorank, reverse=True)
- remainN=N-index1
- for i in range(remainN):
- chromo.append(temp_crowd[i])
- return chromo

5. 变异
遗传算法引入变异的目的有两个:
一是使遗传算法具有局部的随机搜索能力。当遗传算法通过交叉算子已接近最优解邻域时,利用变异算子的这种局部随机搜索能力可以加速向最优解收敛。显然,此种情况下的变异概率应取较小值,否则接近最优解的积木块会因变异而遭到破坏。
二是使遗传算法可维持群体多样性,以防止出现未成熟收敛现象。此时收敛概率应取较大值。
- def cross_mutation(population_parent,x_num, x_min, x_max, pc, pm, yita1, yita2,fitness_function_name):
- #模拟二进制交叉和多项式变异
- ###模拟二进制交叉
- population_offspring=[]
- #随机选取两个父代个体
- for i in range(round(len(population_parent) / 2)):
- parent_1=round(len(population_parent) * random.random())
- if (parent_1==len(population_parent)):
- parent_1= len(population_parent) - 1
- parent_2=round(len(population_parent) * random.random())
- if (parent_2==len(population_parent)):
- parent_2= len(population_parent) - 1
- while (parent_1==parent_2):
- parent_1=round(len(population_parent) * random.random())
- if (parent_1==len(population_parent)):
- parent_1= len(population_parent) - 1
- individual_parent1=population_parent[parent_1]
- individual_parent2=population_parent[parent_2]
- individual_off1=individual_parent1
- individual_off2=individual_parent2
- if(random.random()<pc):
- #初始化子代种群
- off1x=[]
- off2x=[]
- #模拟二进制交叉
- for j in range(x_num):
- u1=random.random()
- if(u1<=0.5):
- gama=float((2*u1)**(1/(yita1+1)))
- else:
- gama=float((1/(2*(1-u1)))**(1/(yita1+1)))
- off11=float(0.5*((1+gama)*individual_parent1.chromosome[j]+(1-gama)*individual_parent2.chromosome[j]))
- off22=float(0.5*((1-gama)*individual_parent1.chromosome[j]+(1+gama)*individual_parent2.chromosome[j]))
- #使子代在定义域内
- if (off11>x_max[j]):
- off11=x_max[j]
- elif (off11<x_min[j]):
- off11=x_min[j]
- if (off22>x_max[j]):
- off22=x_max[j]
- elif (off22<x_min[j]):
- off22=x_min[j]
- off1x.append(off11)
- off2x.append(off22)
- individual_off1=Individual(np.array(off1x),fitness_function_name,x_num)
- individual_off2=Individual(np.array(off2x),fitness_function_name,x_num)
- #多项式变异
- if (random.random()<pm):
- off1x=[]
- off2x=[]
- for j in range(x_num):
- low = x_min[j] # 确定上下边界
- up = x_max[j] # 确定上下边界
- delta1 = polynomial_mutation(low, up, individual_off1.chromosome[j],yita2)
- off11=float(individual_off1.chromosome[j]+delta1)
- delta2 = polynomial_mutation(low, up, individual_off2.chromosome[j], yita2)
- off22=float(individual_off2.chromosome[j]+delta2)
- if (off11>x_max[j]):
- off11=x_max[j]
- elif (off11<x_min[j]):
- off11=x_min[j]
- if (off22>x_max[j]):
- off22=x_max[j]
- elif (off22<x_min[j]):
- off22=x_min[j]
- off1x.append(off11)
- off2x.append(off22)
- individual_off1=Individual(np.array(off1x),fitness_function_name,x_num)
- individual_off2=Individual(np.array(off2x),fitness_function_name,x_num)
- population_offspring.append(individual_off1)
- population_offspring.append(individual_off2)
- return population_offspring
-
- # 多项式变异
- def polynomial_mutation(low, up, x,yita2):
- dd1_a = (x - low) / (up - low)
- dd1_b = (up - x) / (up - low)
- if dd1_a < dd1_b:
- dd1 = dd1_a
- else:
- dd1 = dd1_b
- dd1 = (1 - dd1) ** (yita2 + 1)
- u1 = random.random()
- if (u1 < 0.5):
- delta1 = (up - low) * ((2 * u1 + (1 - 2 * u1) * dd1) ** (1 / (yita2 + 1)) - 1)
- else:
- delta1 = (up - low) * (
- 1 - (2 * (1 - u1) + 2 * (u1 - 0.5) * dd1) ** (1 / (yita2 + 1)))
- return delta1



