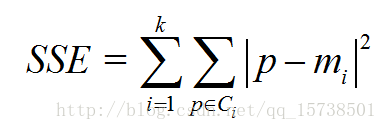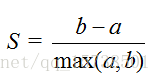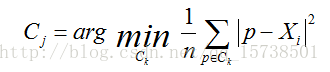- 1网络爬虫之网页数据解析(JSON与JsonPATH)_u8c22
- 2ant design Form 组件总结 结合Modal 自定义Modal的实现 (Upload Input Select DatePicker Cascader)_modalform 退出,渲染oncancel
- 3HarmonyOS学习路之方舟开发框架—学习ArkTS语言(基本语法 四)_arkts开发语言
- 4vue百度地图通过地址名称来获取该地址的经纬度gps_vue 百度地图 标注 获取 经纬度
- 5Unity Koreographer 之 音乐制作插件介绍学习,一般使用步骤介绍(包括:一般音乐游戏制作流程简绍) 一
- 6微信小程序底部实现自定义动态Tabbar_微信下程序中动态使用tabbar
- 7解决使用element-plus时使用el-select-v2组件时,选中后无法移除focus的状态的方法。_element ui el-select-v2
- 8地图常见操作总结_settrafficoff
- 9C# 浮点类型,长度,范围,精度,定义和赋值_c#float中浮点类型
- 10前端炫酷登录页,拿来就能用_好看的前端登陆页面
Kmeans聚类中常用的k值寻找方法-手肘法-轮廓系数法
赞
踩
最近做了一个数据挖掘的项目,挖掘过程中用到了K-means聚类方法,但是由于根据行业经验确定的聚类数过多并且并不一定是我们获取到数据的真实聚类数,所以,我们希望能从数据自身出发去确定真实的聚类数,也就是对数据而言的最佳聚类数。为此,我查阅了大量资料和博客资源,总结出主流的确定聚类数k的方法有以下两类。
1.手肘法
1.1 理论
手肘法的核心指标是SSE(sum of the squared errors,误差平方和),
其中,Ci是第i个簇,p是Ci中的样本点,mi是Ci的质心(Ci中所有样本的均值),SSE是所有样本的聚类误差,代表了聚类效果的好坏。
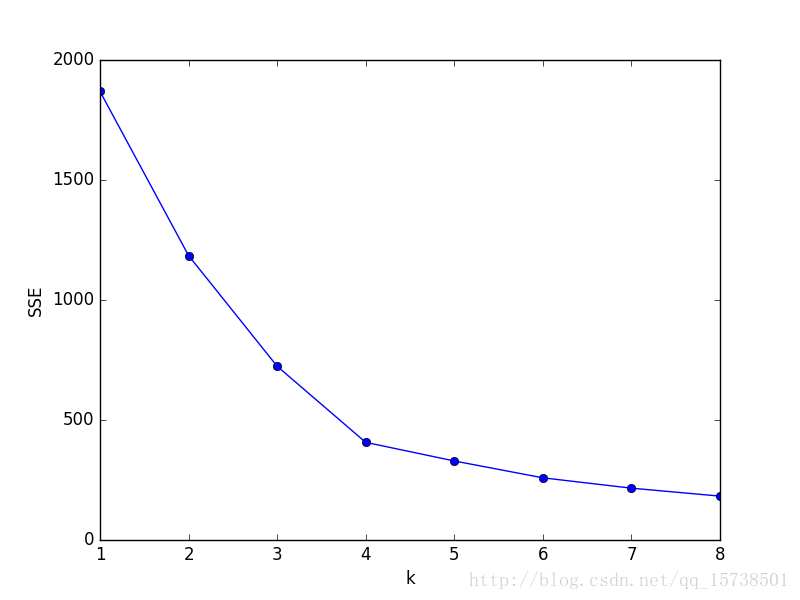
手肘法的核心思想是:随着聚类数k的增大,样本划分会更加精细,每个簇的聚合程度会逐渐提高,那么误差平方和SSE自然会逐渐变小。并且,当k小于真实聚类数时,由于k的增大会大幅增加每个簇的聚合程度,故SSE的下降幅度会很大,而当k到达真实聚类数时,再增加k所得到的聚合程度回报会迅速变小,所以SSE的下降幅度会骤减,然后随着k值的继续增大而趋于平缓,也就是说SSE和k的关系图是一个手肘的形状,而这个肘部对应的k值就是数据的真实聚类数。当然,这也是该方法被称为手肘法的原因。
1.2 实践
我们对预处理后数据.csv 中的数据利用手肘法选取最佳聚类数k。具体做法是让k从1开始取值直到取到你认为合适的上限(一般来说这个上限不会太大,这里我们选取上限为8),对每一个k值进行聚类并且记下对于的SSE,然后画出k和SSE的关系图(毫无疑问是手肘形),最后选取肘部对应的k作为我们的最佳聚类数。python实现如下:
-
import pandas
as pd
-
from sklearn.cluster
import KMeans
-
import matplotlib.pyplot
as plt
-
-
df_features = pd.read_csv(
r'C:\预处理后数据.csv',encoding=
'gbk')
# 读入数据
-
'利用SSE选择k'
-
SSE = []
# 存放每次结果的误差平方和
-
for k
in range(
1,
9):
-
estimator = KMeans(n_clusters=k)
# 构造聚类器
-
estimator.fit(df_features[[
'R',
'F',
'M']])
-
SSE.append(estimator.inertia_)
-
X = range(
1,
9)
-
plt.xlabel(
'k')
-
plt.ylabel(
'SSE')
-
plt.plot(X,SSE,
'o-')
-
plt.show()
- 1
显然,肘部对于的k值为4,故对于这个数据集的聚类而言,最佳聚类数应该选4
2. 轮廓系数法
2.1 理论
该方法的核心指标是轮廓系数(Silhouette Coefficient),某个样本点Xi的轮廓系数定义如下:
其中,a是Xi与同簇的其他样本的平均距离,称为凝聚度,b是Xi与最近簇中所有样本的平均距离,称为分离度。而最近簇的定义是
其中p是某个簇Ck中的样本。事实上,简单点讲,就是用Xi到某个簇所有样本平均距离作为衡量该点到该簇的距离后,选择离Xi最近的一个簇作为最近簇。
求出所有样本的轮廓系数后再求平均值就得到了平均轮廓系数。平均轮廓系数的取值范围为[-1,1],且簇内样本的距离越近,簇间样本距离越远,平均轮廓系数越大,聚类效果越好。那么,很自然地,平均轮廓系数最大的k便是最佳聚类数。
2.2 实践
我们同样使用2.1中的数据集,同样考虑k等于1到8的情况,对于每个k值进行聚类并且求出相应的轮廓系数,然后做出k和轮廓系数的关系图,选取轮廓系数取值最大的k作为我们最佳聚类系数,python实现如下:
-
import pandas
as pd
-
from sklearn.cluster
import KMeans
-
from sklearn.metrics
import silhouette_score
-
import matplotlib.pyplot
as plt
-
-
df_features = pd.read_csv(
r'C:\Users\61087\Desktop\项目\爬虫数据\预处理后数据.csv',encoding=
'gbk')
-
Scores = []
# 存放轮廓系数
-
for k
in range(
2,
9):
-
estimator = KMeans(n_clusters=k)
# 构造聚类器
-
estimator.fit(df_features[[
'R',
'F',
'M']])
-
Scores.append(silhouette_score(df_features[[
'R',
'F',
'M']],estimator.labels_,metric=
'euclidean'))
-
X = range(
2,
9)
-
plt.xlabel(
'k')
-
plt.ylabel(
'轮廓系数')
-
plt.plot(X,Scores,
'o-')
-
plt.show()
- 1
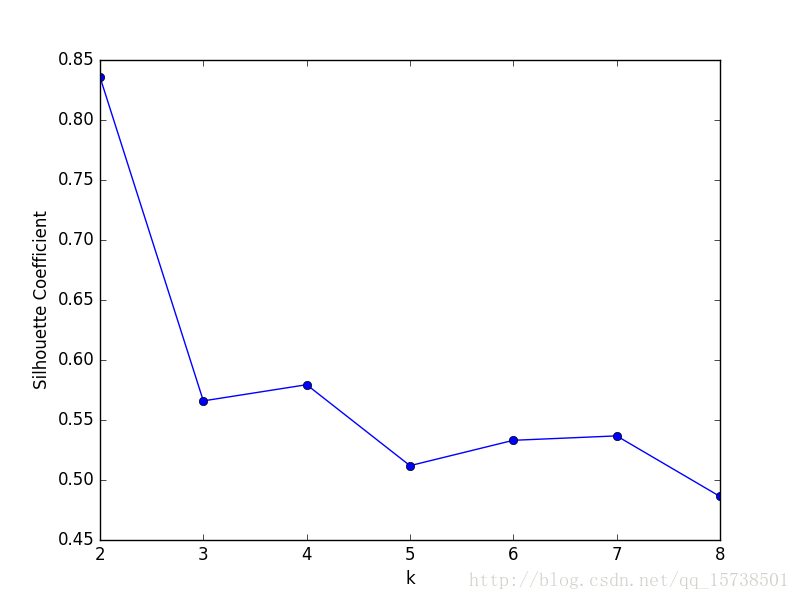
得到聚类数k与轮廓系数的关系图:
可以看到,轮廓系数最大的k值是2,这表示我们的最佳聚类数为2。但是,值得注意的是,从k和SSE的手肘图可以看出,当k取2时,SSE还非常大,所以这是一个不太合理的聚类数,我们退而求其次,考虑轮廓系数第二大的k值4,这时候SSE已经处于一个较低的水平,因此最佳聚类系数应该取4而不是2。
但是,讲道理,k=2时轮廓系数最大,聚类效果应该非常好,那为什么SSE会这么大呢?在我看来,原因在于轮廓系数考虑了分离度b,也就是样本与最近簇中所有样本的平均距离。为什么这么说,因为从定义上看,轮廓系数大,不一定是凝聚度a(样本与同簇的其他样本的平均距离)小,而可能是b和a都很大的情况下b相对a大得多,这么一来,a是有可能取得比较大的。a一大,样本与同簇的其他样本的平均距离就大,簇的紧凑程度就弱,那么簇内样本离质心的距离也大,从而导致SSE较大。所以,虽然轮廓系数引入了分离度b而限制了聚类划分的程度,但是同样会引来最优结果的SSE比较大的问题,这一点也是值得注意的。
总结
从以上两个例子可以看出,轮廓系数法确定出的最优k值不一定是最优的,有时候还需要根据SSE去辅助选取,这样一来相对手肘法就显得有点累赘。因此,如果没有特殊情况的话,我还是建议首先考虑用手肘法。