- 1机器学习与大数据基础知识(一)_大模型 基于规则
- 2MinGW下载并配置gcc/g++编译环境_mingw-get-setup.exe下载
- 3TCP协议之《乱序队列Out-Of-Order》_tcp out of order
- 4超详细,抖音cid广告计划搭建全流程解读
- 5OAuth2.0 认证服务器密码模式提示 无效的grant_type: password_authorizationgranttype password 方式被弃用
- 6C++多线程中volatile关键字的作用_c++多线程共享变量volatile
- 7Poe 和 ChatGPT 有何分別,可以平替吗?_poe chatgpt
- 8【c语言篇】每日一题-pta-实验11-2-9 链表逆置
- 9Android Studio配置夜神模拟器异常解决_android 夜神模拟器运行一次之后就消失了
- 10【Linux】Ubuntu系统下安装与切换多版本gcc/g++_ubuntu 配置指定版本gcc环境
R语言拟合线性混合效应模型、固定效应随机效应参数估计可视化生物生长、发育、繁殖影响因素...
赞
踩
全文链接:https://tecdat.cn/?p=35338
本文将介绍如何设置工作目录、读取数据、标准化数据、拟合线性混合效应模型、提取随机效应参数、绘制相关性图和Dot-and-Whisker图,以帮助研究人员更好地理解数据并进行有效的数据分析(点击文末“阅读原文”获取完整代码数据)。
相关视频
通过这些步骤,我们可以深入了解数据之间的关系和影响,为进一步的数据建模和分析提供基础。
设置工作目录
在进行数据分析之前,确保设置了正确的工作目录,以便读取和保存文件。如果未特别设置,R将使用默认的工作目录。
加载必要的库
加载了tidyverse包,它包含了多个用于数据清洗、处理和可视化的R包,如dplyr、tidyr、ggplot2等。
读取数据
这个数据集是关于某种生物的生殖和生长特性的详细记录。数据集中包含了多个与生物的生长、繁殖和生理特征相关的变量。
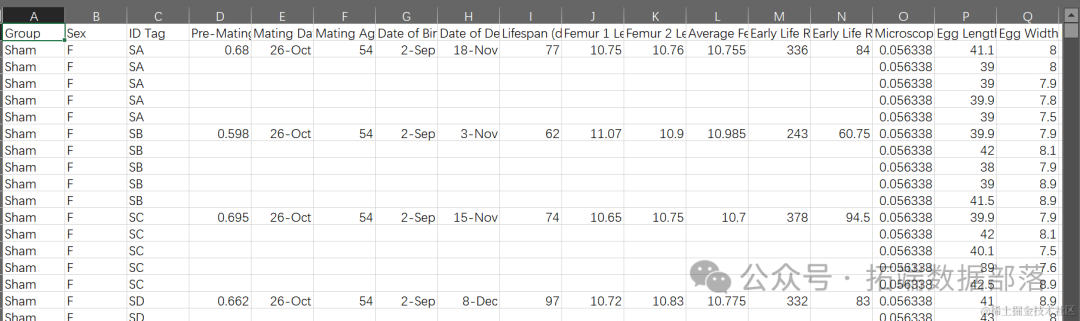
以下是对这些变量的解释:
Group:可能表示不同的实验组、种群或分组,用于比较不同条件下生物的表现。
Sex:生物的性别。
ID Tag:每个个体的唯一标识符,用于追踪和区分不同的个体。
Pre-Mating Weight (~2 weeks) :交配前约两周的体重,这可能用于评估个体的健康状况或发育阶段。
Mating Date 和 Mating Age (days) :交配日期和交配时的年龄(以天为单位)。
Date of Birth、Date of Death 和 Lifespan (days) :出生日期、死亡日期和寿命(以天为单位)。
Femur Length:股骨(或其他长骨)的长度,可能用于评估个体的体型或发育状况。
Early Life Reproductive Output 和 Rate:早期生活阶段的繁殖输出和繁殖率(每四天产卵数)。
Microscope (mm/ocular units) :显微镜的放大倍数或单位转换系数。
Egg Length 和 Width(以及对应的毫米单位):卵的长度和宽度,可能用于评估卵的大小和形状。
Early Life Mass of Eggs 和 Average Mass per Egg:早期生活阶段卵的质量和平均单个卵的质量。
Late Life Reproductive Output 和 Rate:晚期生活阶段的繁殖输出和繁殖率。
Early Life Egg Size Reproductive Effort 和 Egg Mass Reproductive Effort:早期生活阶段基于卵大小或质量的繁殖努力。
Hatching Success 和 Reproductive Success:孵化成功率和繁殖成功率。
Early Life Hatchling vigor:早期生活阶段幼体的活力或生存能力。
Total Reproductive Output:整个生活阶段的总繁殖输出。
Femur Length-related Variables:与股骨长度相关的多个变量,可能用于评估体型对繁殖和其他特性的影响。
Egg Complement Dish Mass 和 Total Dry Egg Complement Mass:与卵的补充或测量相关的质量和干重。
这个数据集用于研究生物的生长、发育、繁殖以及这些特性如何受到不同条件或处理的影响。
lh <- read.csv(file = "Rad1.csv")使用read.csv函数读取名为Rad1.csv的CSV文件,并将其内容存储在变量lh中。
接下来,我们提取了lh数据框中特定的列,并创建了一个新的数据框lh1,用于后续分析。
在此步骤中,我们重命名了列名,使它们更具描述性和简洁性。
对数据进行标准化
标准化是一种常用的数据预处理技术,用于将数据转换为均值为0、标准差为1的尺度,从而消除不同变量之间的量纲差异。
- # c()函数用于删除scale()函数结果中不需要的结构
-
- for (i in 1:length(fitness_vars)) {
-
- lh2[[fitness_vars_sc[i]]] <- c(scale(lh1[[fitness_vars[i]]]))
-
- }
在这个代码块中,我们首先定义了一个包含需要标准化的变量名的向量fitness_vars。然后,我们创建了一个新的变量名向量fitness_vars_sc,该向量在原始变量名后添加了“_s”后缀,以区分原始变量和标准化后的变量。
接下来,我们创建了一个与lh1相同结构的数据框lh2,用于存储标准化后的数据。然后,我们使用循环和scale函数对lh1中指定的变量进行标准化,并将结果存储在lh2中对应的标准化变量名下。c()函数用于确保我们只保留数值结果,去除可能由scale函数产生的其他结构。
完成上述步骤后,lh2数据框将包含与lh1相同的变量,但fitness_vars中列出的变量已经被标准化。这有助于后续的数据分析和建模工作,特别是在需要比较不同尺度或量纲的变量时。
融化代码
首先,我们进行变量的筛选,移除那些没有标准化的变量。
- %>% gather(trait, value, egg.size_s:ovi.rate_s) # 将标准化后的变量从宽格式转换为长格式
-
- %>% na.omit) # 移除包含缺失值的行
在处理缺失值时,首先要考虑的是为什么会出现这些缺失值。
这些缺失值是在数据收集或处理过程中随机产生的。
接下来,我们来拟合线性混合效应模型。
拟合线性混合效应模型
- (value ~ trait:(dose) - 1 +
-
- (trait - 1 | dose) + (trait - 1 | indiv
上述代码中的模型表达式包含了固定效应和随机效应。trait:(dose)表示trait和dose的交互效应,-1用于移除截距。(trait - 1 | dose)和(trait - 1 | individual)分别表示trait随dose和individual的随机效应,这里减去1是为了避免包含截距的随机效应,这通常是为了模型的简化。control参数中的设置用于忽略某些在拟合模型时可能出现的警告。
点击标题查阅往期内容

数据分享|R语言用lme4多层次(混合效应)广义线性模型(GLM),逻辑回归分析教育留级调查数据

左右滑动查看更多

01
02
03
04
从拟合的混合效应模型中提取广义成分
最后,我们从拟合的混合效应模型lmer1中提取随机效应的方差协方差矩阵的对角线元素(即广义成分theta)。表达式检查这些广义成分是否都大于1e-4,结果返回TRUE,表示所有的广义成分都满足这一条件。这通常用于检查随机效应的方差是否显著不为零,从而验证模型中包含随机效应的合理性。
随机效应参数估计
这些参数是以每个随机效应项的相对Cholesky因子进行参数化的。
为了检查随机效应参数是否大于1e-4,我们进行了之前的检查。现在,我们进一步查看这些随机效应的方差和相关性成分。
提取方差和相关性成分
VarCorr(lmer1)
输出结果展示了不同组(individual和dose)以及残差项的随机效应方差和相关性。对于individual组,我们可以看到每个trait的随机效应标准差以及它们之间的相关系数。类似地,对于dose组,也展示了每个trait的随机效应标准差以及它们之间的相关性。最后,还给出了残差的标准差。
相关性图
为了更直观地查看不同变量之间的相关系数矩阵,我们可以绘制相关性图。
- # 为了在相关性图中包含残差方差,我们需要将dose组的方差-协方差矩阵的对角线元素加上残差的方差
-
-
-
-
- # 绘制individual组的混合相关性图,上半部分为椭圆表示相关性
-
-
-
-
- # 绘制dose组的混合相关性图,上半部分为椭圆表示相关性
-
- xed(cov2cor(vv1$dose), upper="ellipse")
上述代码首先加载了包,然后设置了绘图参数以便在同一行上显示两个图。接着,提取了模型lmer1的方差协方差矩阵,并调整了dose组的方差以包含残差方差。最后,使用函数分别绘制了individual组和dose组的混合相关性图,其中上半部分使用椭圆来表示变量之间的相关性。这样,我们可以更直观地了解不同随机效应组内部变量之间的相关性结构。

我们可以理解您已经根据相关性图解读出了几个关键的相关性关系:
mass和size之间存在显著的负相关关系
Success和vigor之间存在显著的正相关关系
Rate和success之间存在显著的负相关关系
Mass和rate之间存在显著的负相关关系
这些关系是通过观察相关性图中的椭圆形状和颜色深浅来推断的。椭圆的方向表示了变量之间的正负相关性,而椭圆的形状和颜色深浅则反映了相关性的强度。
接下来,为了展示线性混合效应模型(LMM)的固定效应估计值及其不确定性。可以帮助我们可视化模型中固定效应的点估计、置信区间以及它们与零线的相对位置。通过添加垂直参考线(geom_vline),我们可以更容易地判断哪些效应的置信区间包含了零,从而判断它们是否显著。
- dlot(lmer1) +
-
- geom_vline(xintercept = 0, lty = 2) + # 添加零线参考
-
- theme_minimal() + # 使用minimal主题进行美化
在这段代码中,lmer1应该是一个已经拟合好的线性混合效应模型对象。(lmer1)会生成一个包含模型中所有固定效应估计值及其置信区间的散点图。geom_vline(xintercept = 0, lty = 2)添加了一条虚线作为零线参考,帮助我们判断效应是否显著(即其置信区间是否跨越零线)。theme_minimal()用于选择一个简洁的主题来美化图形,而labs()函数用于添加图形标题和修改坐标轴标签。
通过查看这个图,我们可以快速地识别出哪些固定效应是显著的(即它们的置信区间没有包含零线),以及效应的大小和方向。
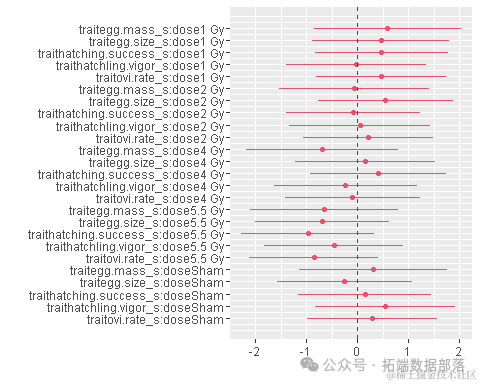
Dot-and-Whisker Plots of Regression Results 是一种非常直观的方式来展示线性回归模型中固定效应的估计值及其不确定性。默认情况下,图中的“whisker”部分表示了95%的置信区间,这可以帮助我们判断估计值的稳定性以及它们是否显著不为零。
我们可以理解,通过查看Dot-and-Whisker Plot,识别出了以下关于变量对value的影响:
正向回归影响:
mass dose 1:这个变量对value有正向影响,意味着当mass dose 1增加时,value也倾向于增加。size dose 2:这个变量同样对value有正向影响。sucess:sucess变量也对value产生正向回归影响。
负向回归影响:
mass dose 4:这个变量对value有负向影响,意味着当mass dose 4增加时,value倾向于减少。rate dose 5:rate dose 5变量对value产生负向回归影响。
这些结论是基于图中估计值的符号以及它们置信区间是否跨越零线来推断的。如果估计值的置信区间完全位于零线的上方,则我们可以认为该变量对value有正向影响;相反,如果置信区间完全位于零线的下方,则变量对value有负向影响。如果置信区间跨越零线,则我们不能确定该变量对value的影响方向是否显著。

点击文末“阅读原文”
获取全文完整代码数据资料。
本文选自《R语言拟合线性混合效应模型、固定效应随机效应参数估计可视化生物生长、发育、繁殖影响因素》。


点击标题查阅往期内容
R语言线性混合效应模型(固定效应&随机效应)和交互可视化3案例
生态学模拟对广义线性混合模型GLMM进行功率(功效、效能、效力)分析power analysis环境监测数据
有限混合模型聚类FMM、广义线性回归模型GLM混合应用分析威士忌市场和研究专利申请数据
如何用潜类别混合效应模型(Latent Class Mixed Model ,LCMM)分析老年痴呆年龄数据
R语言用lme4多层次(混合效应)广义线性模型(GLM),逻辑回归分析教育留级调查数据
R语言混合效应逻辑回归(mixed effects logistic)模型分析肺癌数据
R语言建立和可视化混合效应模型mixed effect model
R语言用Rshiny探索lme4广义线性混合模型(GLMM)和线性混合模型(LMM)
R语言如何解决线性混合模型中畸形拟合(Singular fit)的问题
R语言用WinBUGS 软件对学术能力测验建立层次(分层)贝叶斯模型
R语言用WinBUGS 软件对学术能力测验(SAT)建立分层模型
使用SAS,Stata,HLM,R,SPSS和Mplus的分层线性模型HLM
R语言用WinBUGS 软件对学术能力测验建立层次(分层)贝叶斯模型
SPSS中的多层(等级)线性模型Multilevel linear models研究整容手术数据

![]()



