- 1STM32 学习(系统滴答定时器)_systick->val
- 2工控网络中网络威胁情报与物理安全的关联
- 3华为OD机试C卷-- 查找接口成功率最优时间段(Java & JS & Python & C)
- 4端口的常用端口号_最常用的服务名称及其端口号
- 5全面解读:ChatGPT与大模型技术家族——GPT、AIGC、LLM解析_chatgpt和llm大模型
- 6探索 GitKraken Crack:一款高效、直观的Git客户端管理工具
- 7【高阶数据结构】红黑树详解_stl 红黑树
- 8怎么在neo4j-community-4.4.25版本中创建自定义的数据库?_neo4社区版不能创建数据库吗
- 92023 年全国职业院校技能大赛(高职组) “云计算应用”赛项赛卷 B部分解析_云计算大赛考什么
- 10万字长文丨畅游2024年人工智能趋势、预测和可能性
论文浅尝 - ICLR2022 | OntoProtein:融入基因本体知识的蛋白质预训练
赞
踩
论文题目:OntoProtein: Protein Pretraining With Gene Ontology Embedding
本文作者:张宁豫(浙江大学)、毕祯(浙江大学)、梁孝转(浙江大学)、程思源(浙江大学)、洪浩森(浙江大学)、邓淑敏(浙江大学)、连佳长(浙江大学)、张强(浙江大学)、陈华钧(浙江大学)
发表会议:ICLR 2022
论文链接:https://arxiv.org/pdf/2201.11147.pdf
代码链接:https://github.com/zjunlp/OntoProtein

一、引言
近年来,预训练模型以强大的算法效果,席卷了自然语言处理为代表的各大AI榜单与测试数据集。与自然语言类似,蛋白质的一级结构具有序列特性,这为将语言预训练模型引入蛋白质表示提供了有利条件。然而,蛋白质本质上不同于自然语言文本,其包含了大量预训练目标较难习得的生物学知识。事实上,人类科学家已经积累了海量的关于蛋白质结构功能的生物学知识。那么如何利用这些知识促进蛋白质预训练呢?本文将介绍被ICLR2022录用的新工作:OntoProtein,其提出一种新颖的融入知识图谱的蛋白质预训练方法。
二、蛋白质预训练
蛋白质是控制生物和生命本身的基本大分子,对蛋白质的研究有助于理解人类健康和发展疾病疗法。蛋白质包含一级结构,二级结构和三级结构,其中一级结构与语言具有相似的序列特性。受到自然语言处理预训练模型的启发,诸多蛋白质预训练模型和工具被提出,包括MSA Transformer[1]、ProtTrans[2]、悟道 · 文溯[3]、百度的PaddleHelix等。大规模无监督蛋白质预训练甚至可以从训练语料中习得一定程度的蛋白质结构和功能。然而,蛋白质本质上不同于自然语言文本,其包含了诸多生物学特有的知识,较难直接通过预训练目标习得,且会受到数据分布影响低频长尾的蛋白质表示。为了解决这些问题,我们利用人类科学家积累的关于蛋白质结构功能的海量生物知识,提出融合知识图谱的蛋白质预训练方法。下面首先介绍知识图谱构建的方法。
三、基因知识图谱
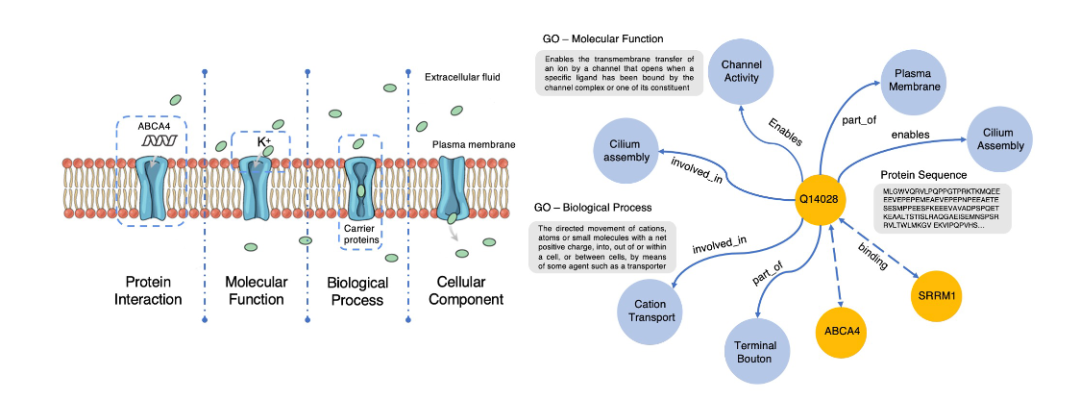
我们通过访问公开的基因本体知识图谱“Gene Ontology(简称Go)”,并将其和来自Swiss-Prot数据库的蛋白质序列对齐,来构建用于预训练的知识图谱ProteinKG25,该知识图谱包含4,990,097个三元组, 其中4,879,951个蛋白质-Go的三元组,110,146 个Go-Go三元组,并已全部开放供社区使用。如下图所示,基于“结构决定功能”的思想,如果在蛋白质预训练过程中显式地告诉模型什么样的结构具备什么样的功能,显然能够促进如蛋白质功能预测、蛋白质交互预测等任务的效果。
四、融入基因知识图谱的蛋白质预训练:OntoProtein
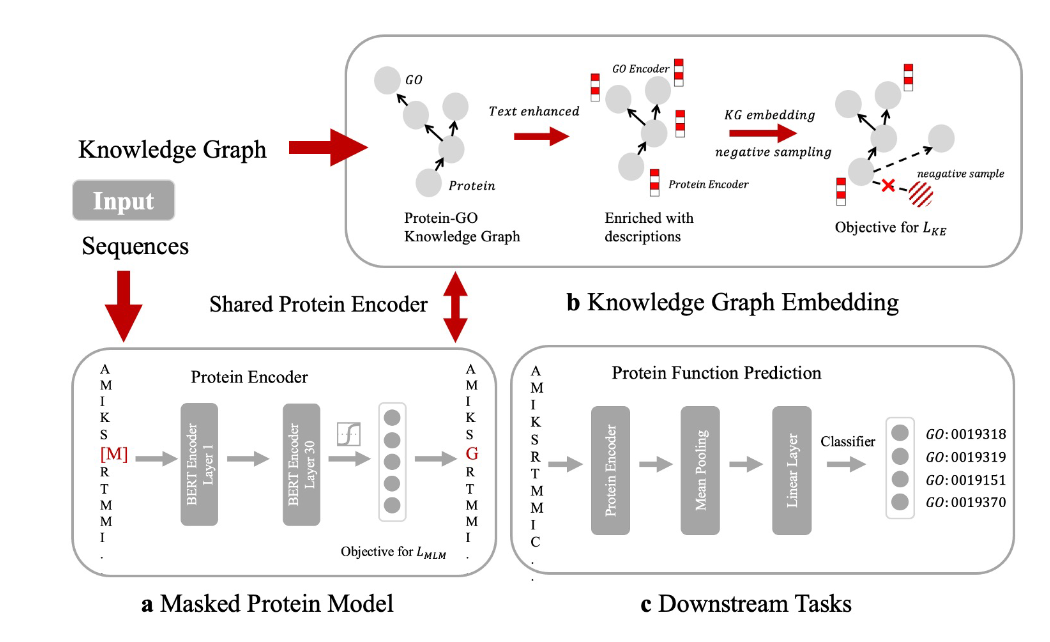
基于构建好的知识图谱,我们设计了一个特殊的蛋白质预训练模型OntoProtein。注意到在预训练输入中包含两种不同的序列:蛋白质序列和描述蛋白质功能、生物过程等的文本描述信息。因此,我们采取两路不同的编码器。对蛋白质序列我们采用已有的蛋白质预训练模型ProtBert进行编码,对文本序列我们采用BERT进行编码。为了更好地进行预训练和融合三元组知识信息,我们采用了两个优化目标。首先是传统的掩码语言模型目标,我们通过随机Mask序列中的一个Token并预测该Token。其次是三元组知识增强目标,我们通过类似知识图谱嵌入学习的方式来植入生物学三元组知识,如下公式所示:
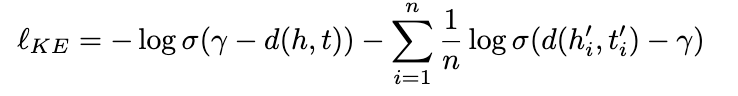
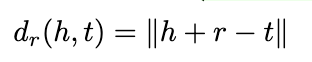
注意到这里的事实知识分为两类不同的三元组,分别是Go-Go和蛋白质-Go,因此我们提出一种知识增强的负采样方法,以获得更有代表性的负样本提升预训练效果,采样方式如下 :
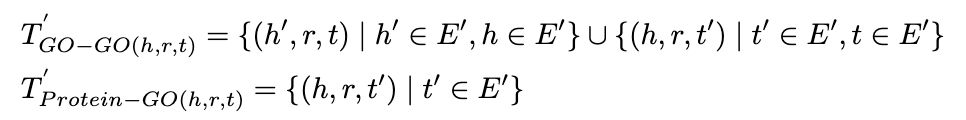
五、实验分析
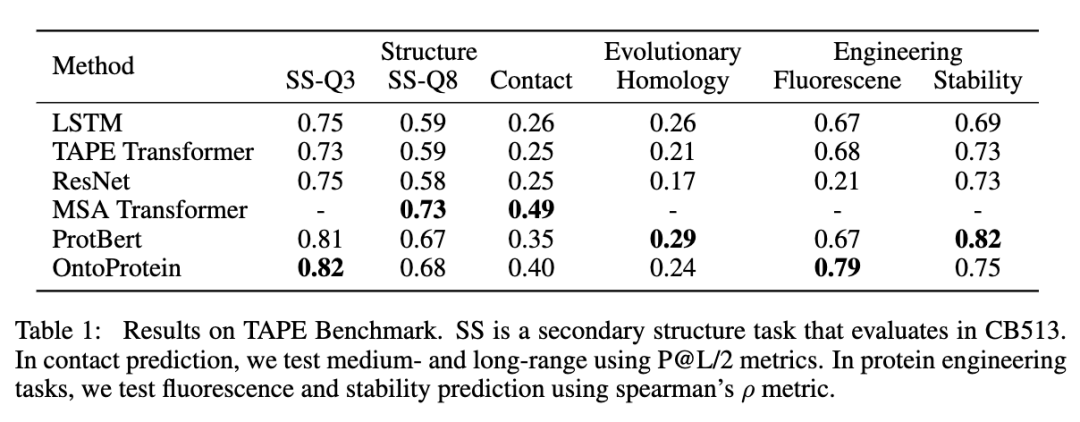
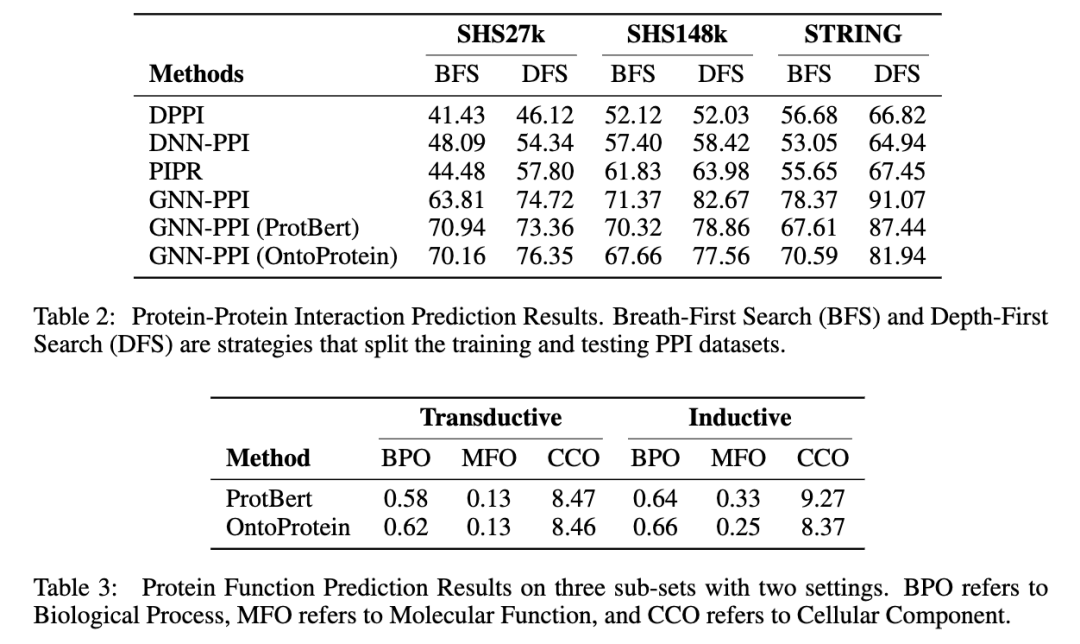
我们在蛋白质测试基准TAPE,以及蛋白质蛋白质交互、蛋白质功能预测(我们参考CAFA竞赛构建了一个新的蛋白质功能预测数据集)上进行了实验。如下表所示,可以发现融合知识图谱的蛋白质预训练方法在一定程度上取得了较好或可比的性能。特别地,我们的方法没有使用同源序列比对(MSA),因此较难超越基于MSA Transformer的方法。详细的实验结果请参见论文,我们会在近期将预训练模型整理并发布到Huggingface上供社区使用。
六、小结与展望
当下蓬勃兴起的 AI for Science 正在促使以数据驱动的开普勒范式和以第一性原理驱动的牛顿范式的深度融合。基于“数据与知识双轮驱动”的学术思想,我们在本文中提出了融合知识图谱的蛋白质预训练方法OntoProtein,并在多个下游任务中验证了模型的效果。在未来,我们将维护好OntoProtein以供更多学者使用,并计划探索融合同源序列比对的知识图谱增强预训练方法以实现更优性能。
[1] MSA Transformer ICML2021
[2] ProtTrans: Towards Cracking the Language of Life’s Code Through Self-Supervised Learning TPAMI2021
[3] Modeling Protein Using Large-scale Pretrain Language Model 2021
OpenKG
OpenKG(中文开放知识图谱)旨在推动以中文为核心的知识图谱数据的开放、互联及众包,并促进知识图谱算法、工具及平台的开源开放。

点击阅读原文,进入 OpenKG 网站。


